Нуклеотидный BLAST
Задание 1. Определение функции и таксономии нуклеотидной последовательности
Последовательность, функции которой нужно определить, была получена при помощи секвенирования по Сэнгеру и расшифрована с помощью программы Unipro UGENE (Практикум 6). Для выполнения этой задачи было проделано выравнивание blastn. Был выбран именно этот вид BLAST, так как мы ничего не знаем о нашей последовательности и нужно найти какую-нибудь последовательность, похожую на нашу. При поиске использованы стандартные значения параметров:
Max target sequences: 100; Expect threshold: 0.05; Word size: 28; Match/Mismatch Scores: 1,-2, поиск по датабазе NucleotideАнализируя выход программы, можно предположить, какому организму принадлежит наша последовательность и какую функцию она выполняет.
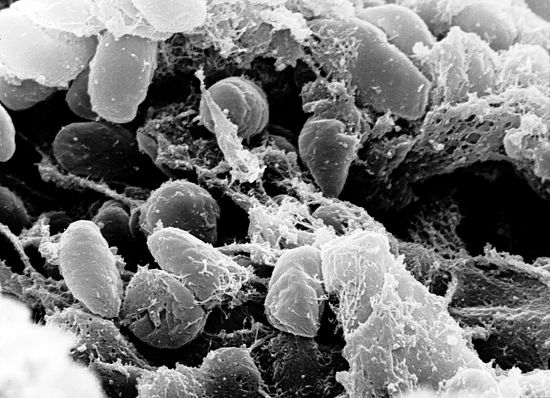
Скорее всего, последовательность принадлежить геному представителя рода Cephalothrix - это такие тонкие, нитевыдные черви, похожие на нематод. Также по выходу выравнивания можно предположить вид: возможно, это Cephalothrix simula (рис. 1)[1].
Наша последовательность, проедположительно, является фрагментом митохондриального генома и кодирует 1 субъединицу цитохром С оксидазы (CO1). Цитохромоксидаза участвует в окислительном фосфорилировании и является важным фрагментом дыхательной цепи.
Выводы о нашем фрагменте были сделаны на основании высокого процента идентичности в выравнивании (>90%) и высокого процента перекрывания.
Поиск генов белков в неаннотированной последовательности
Для выполнения задания был выбран контиг, принадлежащий организму, геном готорого исследовался в практикуме 7: Rattus norvegicus. Длина выбранного контига - 58171 пн. Для предсказания функции белка, закодированного в этом контиге (может быть, частично), была выбрана программа blastx, так как она переводит последовательность нуклеотидов в последовательность аминокислот и ищет сходства именно среди белков (Swissprot). Парамеры поиска:
Database: Swissprot; Organism: exclude Rattus norvegicus (taxid:10116); Expected threshold: 0.05; Matrix: BLOSUM62; Gap Costs: Existence: 11 Extension: 1; Word size: 6
На выходе программы видно, что ближайшие гомологи - белки LINE-1 retrotransposable element ORF2 человека (Homo sapiens) и домовой мыши (Mus musculus). Исследуемый ген крысы на 54% идентичен гену мыши и гены перекрываются на 24%. Эти показатели не идеальны, но объясняются тем, что наш контиг может не полностью содержать ген, кодирующий белок и все-таки, крыса и мышь - не идентичные организмы и в ходе эволюции происходили мутации в гене.
Карта локального сходства гомологичных хромосом двух бактерий
Для построения карты локального сходства двух бактериальных хромосом были выбраны два вида бактерий рода Yersinia: Yersinia similis и Yersinia pestis - возбудитель чумной пневмонии и септической чумы[2] (рис. 2). Геном обеих бактерий состоит из одной полностью расшифрованной хромосомы. Выбранный алгоритм - blastn (megablast), так как нужно сравнить очень близкие последовательности (гомологи). Все параметры взяты по умолчанию (Word size: 28). Результат выравнивания - на рисунке 3 (dot plot). Эти две хромосомы довольно сильно различаются: красным прямоугольником я отметила делецию делецию (вставку участка хромосомы), зеленым - несколько инверсий, фиолетовым - транслокации. Интересно, что вставок или делеций очень мало. Это может быть связано с тем, что инверсии и транслокации редко влияют на фенотип носителя [3], а точечные мутации (вставки иоли делеции) могут серьезно повлиять на его жизнеспособность.

Литература
[1] Faasse, M., & Turbeville, J. (2015). The first record of the north-west Pacific nemertean Cephalothrix simula in northern Europe. Marine Biodiversity Records, 8, E17. doi:10.1017/S1755267214001523
[2] Wikipedia Иерсинии
[3] Wikipenia Инверсия