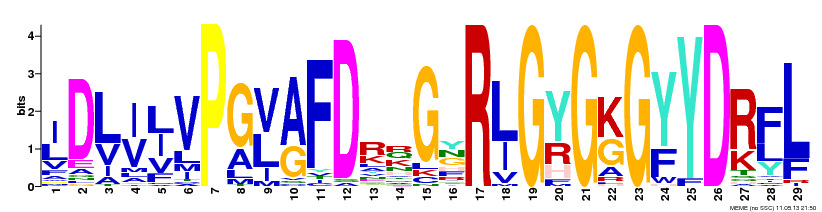Поиск мотивов, программы MEME и MAST
Консольная версия программы MEME
МЕМЕ – наиболее популярная программа множественного локального выравнивания, она находит блоки
(т.е.участки локальных выравниваний без гэпов).
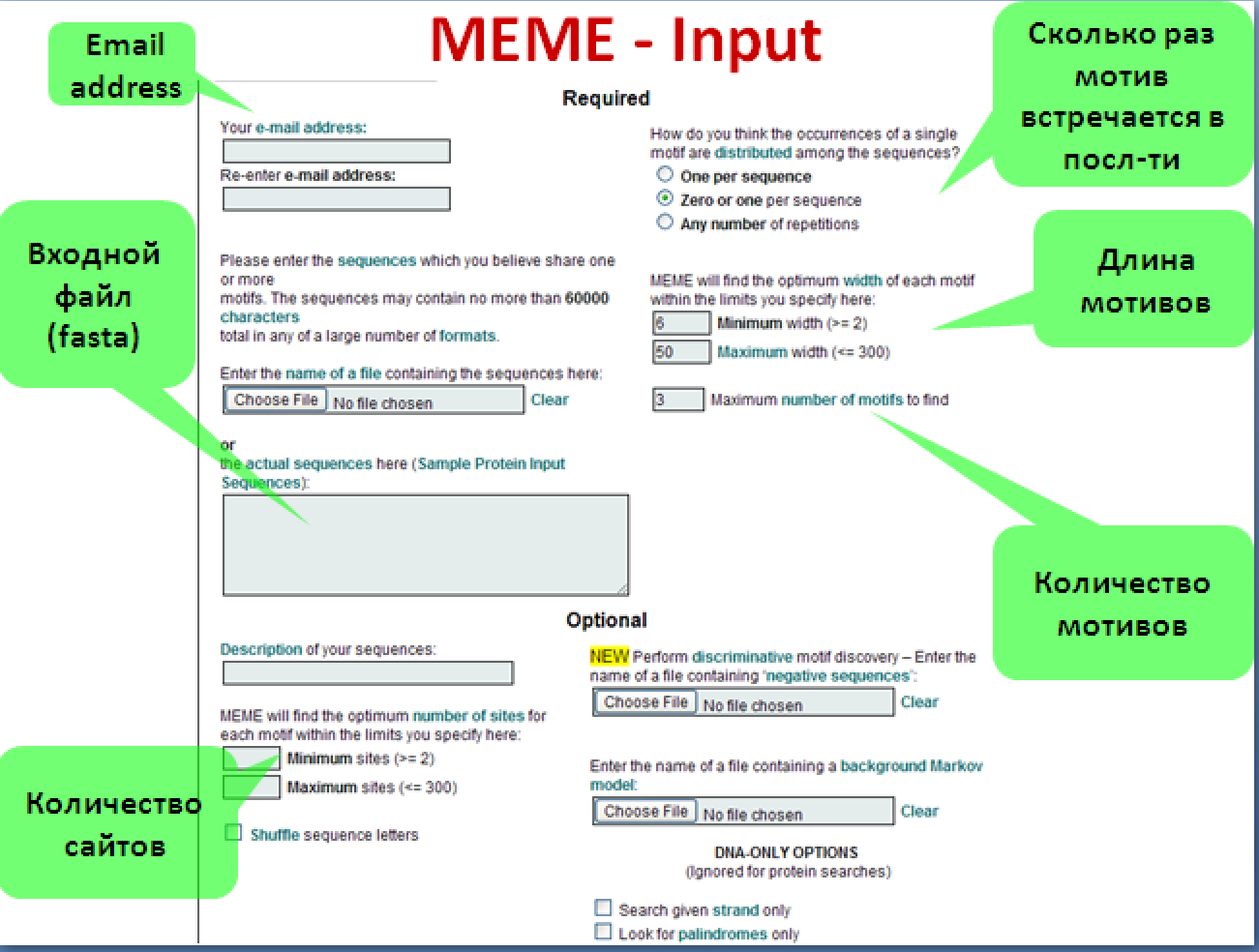
На kodomo введем в командную строку "ememe". Нам предложат ввести файл с последовательностями белков (в нашем случае
это файл в FASTA - формате с последовательностями гомологов белка YQGN_BACSU). Далее, после нажатия Enter
надо ввести название директории, в которую программа запишет результаты своих действий. Теперь посмотрим файл в формате html —
meme.html.
Для гомологов белка YQGN_BACSU программа meme нашла всего один мотив, длиной 29 аминокислотных остатков.
E-value мотива - 2.3e-499, число последовательностей, в которых встретился мотив - 18.
MEME мотивы описаны в виде вероятностно позиционно-спецефичной матрицей, которая показывает вероятность
появление каждой возможной аминокислоты в каждой возможной позиции мотива.
Для визуализации матрицы используется так называемое "LOGO последовательности", содержащее набор букв
(однобуквенное обозначение аминокислот) в кадой позиции мотива.
Индивидуальная высота каждой буквы - вероятность появления ее в данной позиции мотива (чем выше буква, тем чаще она встречается
в последовательностях, обладающих мотивом).
Для дальнейшего изучения рассмотриф файл meme.txt
Создадим файлы с блоками:
из файла meme.txt скопируем выравнивание блока (раздел вида "Motif 1 in BLOCKS format") в отдельный файл motif1.aln.
Вставим первую строчку "CLUSTAL ", это признак данного формата файла с выравниванием.
Переведем файл "motif1.aln" в формат fasta:
$ seqret motif1.aln motif1.fasta (По умолчанию, выходной файл seqret и др. программ EMBOSS - в формате fasta)
Откроем в JalView одновременно и выравнивание последовательностей, и найденный блок; сравним их визуально.
В блоке не все последовательности (нет gi|6321031|ref|NP_011110.1| 5-formyltetrahydrofolate cyclo-ligase [Saccharomyces cerevisiae
S288c]). На нижнем рис.2. красным цветом выделена последовательность, которая отсутствует в блоке.
По ссылке доступен проект JalView с выравненными последовательностями
и с последовательностями из блока.
(Увеличенное изображение при клике на картинке)
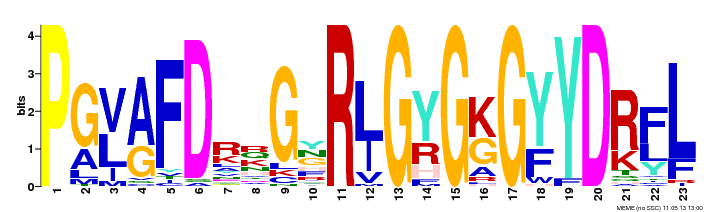
 На рис. слева представлена визуализация блока, найденного программой MEME.
На рис. слева представлена визуализация блока, найденного программой MEME.На рис. снизу выравнивание гомологов белка YQGN_BACSU. Красным цветом выделена последовательность, которая отсутствует в блоке.
Аминокислоты покрашены в соответсnвии с их функциями:
1. Положительно заряженные – Lys(K), Arg(R), His(H) - светло-зеленым цветом
2. Отрицательно заряженные – Asp(D), Glu(E) – синим цветом
3. Полярные – Ser(S), Thr(T), Cys(C), Met(M), Asn(N), Gln(Q) – красным цветом
4. Неполярные – Gly(G), Ala(A), Val(V), Leu(L), Ile(I), Pro(P) – желтым
5. Ароматические – Phe(F), Tyr(Y), Trp(W) – серо-зеленым

Попробуем объяснить почему ее нет.
[IL]D[LV][IV][LIV]VP[GA][VL][AG]FDRxGYR[LI]G[YR]G[KG]G[YF]YD[RK][FL]L - регулярное выражение для блока. В каждой позиции стоят буквы, которые встречаются с частотой больше 0.2.
Проверим нашу выбившуюся последовательность на соответствие данному регулярному выражению. Ниже представлен участок последовательности, не представленной в блоке. Красными стрелками отмечены позиции, которые не подходят под регулярное выражение блока.
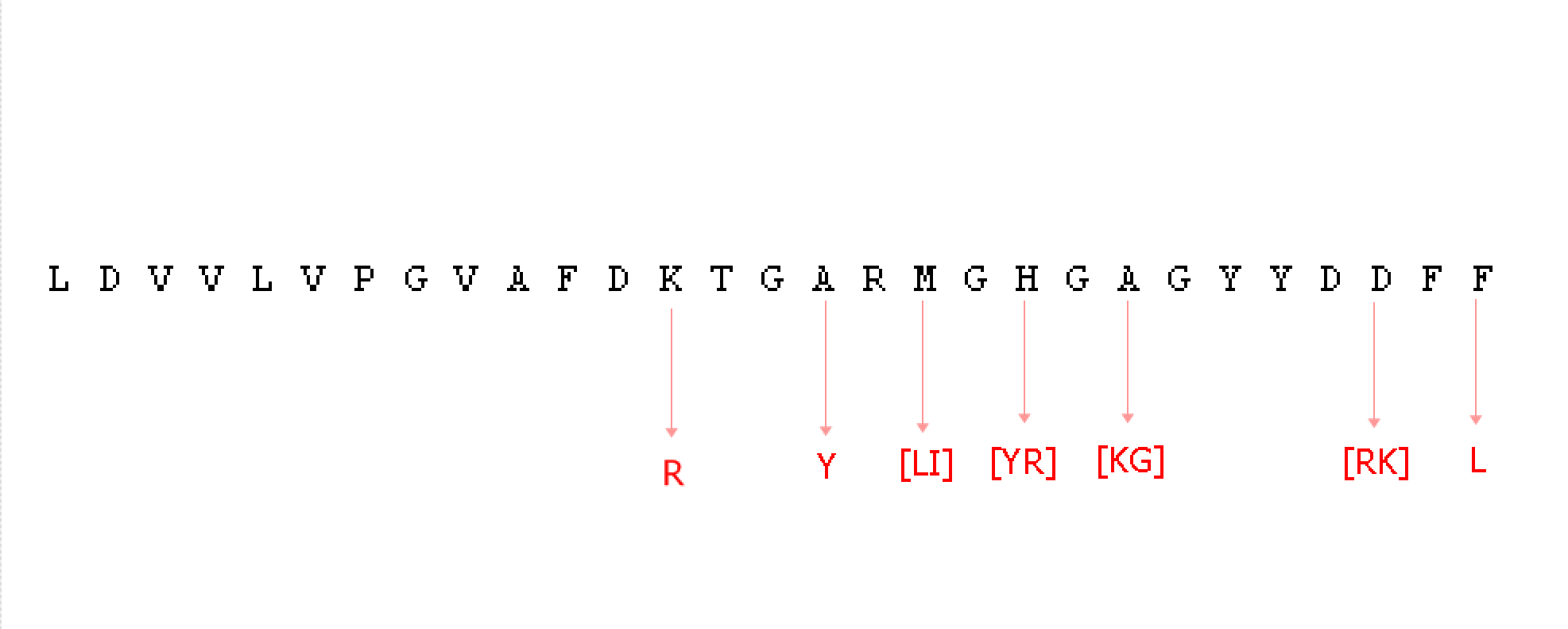
Критическими заменами могли оказаться: [LI] на M (неполярные на полярную), [RK] на D (положительно заряженные на отрицательно), так как они имеют диаметрально противоположенные функциональные группы.
Поиск мотивов при помощи сервиса MEME Suite
Введем с поле "входной файл" - файл в FASTA-формате с последовательностями гомологов белка YQGN_BACSU
(тот же, что и в консольной версии программы).
Нажмем "Start search". Через некоторое время появится страница с результатами.
Из предложенных ссылок выбираем meme.html.
Замечение: остается неясным как долго сервис хранит, полученные результаты.
Сервис meme suit нашел три мотива для гомологов белка YQGN_BACSU. В поле "Discovered Motifs" увидим количество мотивов, значение e-value и количество последовательностей, удовлетворяющих данному мотиву (sites).
(Увеличенное изображение при клике на картинке)
1.4e-434, 35 последовательностей, длина мотива - 23
(Увеличенное изображение при клике на картинке)

9.0e-203, 35 последовательностей, длина мотива - 21
(Увеличенное изображение при клике на картинке)
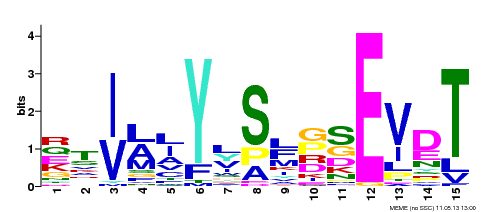
1.9e-088, 33 последовательностей, длина мотива - 15
Поиск найденных мотивов в других последовательностях
(использование программы MAST)
Проведите программой MAST поиск мотивов, найденных программой MEME, в последовательностях,
из которых составлено выравнивание (seed) одного из доменов вашего белка, взятое из Pfam.
(см. Эволюционные домены. Банки Pfam и InterPro)
Извлечем последовательности из выравнивания (то есть уберем знаки пробелов и переведем в fasta-формат) программой degapseq:
$ degapseq PF01812_seed.msf PF01812_seed.fasta
Запустим программу emast:
$ emast -dfile PF01812_seed.fasta meme.txt mastout.html
На вопросы программы отвечаем по умолчанию (нажатием Enter). Откроем полученный файл (mastout.html) браузером.
Мотив, найденный консольной версией программы MEME нашелся во всех 24 последовательностях из PF01812_seed.fasta.
Выравнивание, взятое из Pfam соотвествует мотиву.