Эволюционные домены
На сайте Pfam выбрали домен VirB3, точнее семейство VirB3-подобных белков.
Описание выбранного домена из Pfam
Это семейство включает в себя VirB3 белок из IV подсистемы секреторного пути, который связан с
бактериальными внутренней и наружной мембранами. Сюда так же включается семейство связанного транспортного белка
TrbD, который содержит нуклеотидный связывающий участок и может предоставлять энергию для экспорта
ДНК или экспорта других белков Trb.
AC: PF05101
ID: VirB3
Ссылка на страницу домена в Pfam
Для домена известно 4 архитектуры, для которых доступно 967 последовательностей.
Скачаем выравнивание из Pfam в JalView. Для этого в JalView выберем File -> Fetch Sequences, выбираем нужную базу данных (Pfam), указываем AC семейства. Раскрасим выравнивания по консервативности (ClustalX, By conservation 20%). Сохраним полученное выравнивание как проект и файл fasta.
Получим таблицу с информацией об архитектуре всех последовательностей, содержащих выбранный домен PF05101. Для этого запустим скрипт:
python swisspfam-to-xls.py -z swisspfam.gz -m pfam-AC.txt -o virb3.xls
Где -z это файл в gz формате (с информацией об архитектуре всех последовательностей Uniprot), -m файл с AC выбранного домена, a -о это название файла: в который будет записана полученная информация. Полученная таблица virb3.xlsx
Для дальнейшей работы были выбраны две доменные архитектуры:
- VirB3 - 843 последовательности
- VirB3, CagE_TrbE_VirB, AAA_10 - 118 последовательностей
В список последовательностей добавим колонки с информацией о таксономической принадлежности. Для этого
скачаем полные записи всех последовательностей. Зайдем на сайт Uniprot
далее пройдем по вкладке Retrieve, введем список AC наших последовательностей
из таблицы и нажмем Retrieve. Сохраним в формате flat text в файл flat_taxonomy.txt.
В процессе этого можно столкнуться с небольшими трудностями: некоторые записи были перемещены или вовсе удалены из Uniprot.
Replaced (замены): Q0VUW8, Q4LCF6, Q5W3K0
Deleted (удалены): A9G1S9, B5KAH0, B5S0J4, D5QQW2, D5QR94, D6L2F2, E1XYS7, E1Y010, E8L365, E8L7X4, E8L8D4, E8LA38, F5K7W0,
F5KDX5, F5KS58, G3YW95, G4IK48, G4IRK1, G4K6L8, G4K6U6, G5GZ47, G6IE57, G6IE57, G6IE57, G6IEB0, G6ILU9, I0YEP3,
I0YG32, I0YGT5, Q2VLF9, Q3R4Q0, Q3RAK5
Получим таксононмию:
python uniprot-to-taxonomy.py -i flat_taxonomy.txt -o taxonomy.txt
Из таксона Bacteria выберем два подтаксона Alphaproteobacteria (A), Betaproteobacteria (B) для архитектуры VirB3, CagE_TrbE_VirB, AAA_12 и три подтаксона Gammaproteobacteria (G), Epsilonproteobacteria (E), Fusobacteriales (F) для архитектуры VirB3.
Из каждой архитектуры были отобраны последовательности из заданных подтаксонов (22 и 25 для первой и второй архитектур соответственно). Они представленны на листе Favourites virb3.xlsx, а также отмечены желтым и зеленым цветом на основной таблице для VirB3, CagE_TrbE_VirB, AAA_12 и VirB3 соответственно). Для того что бы получить выравнивнивания только отобранных последовательностей будем использовать скрипт (файл aln.fasta - это выравнивания полученные с pfam):
python filter-alignment.py -i aln.fasta -m favourites.txt -o aln_selected.fasta
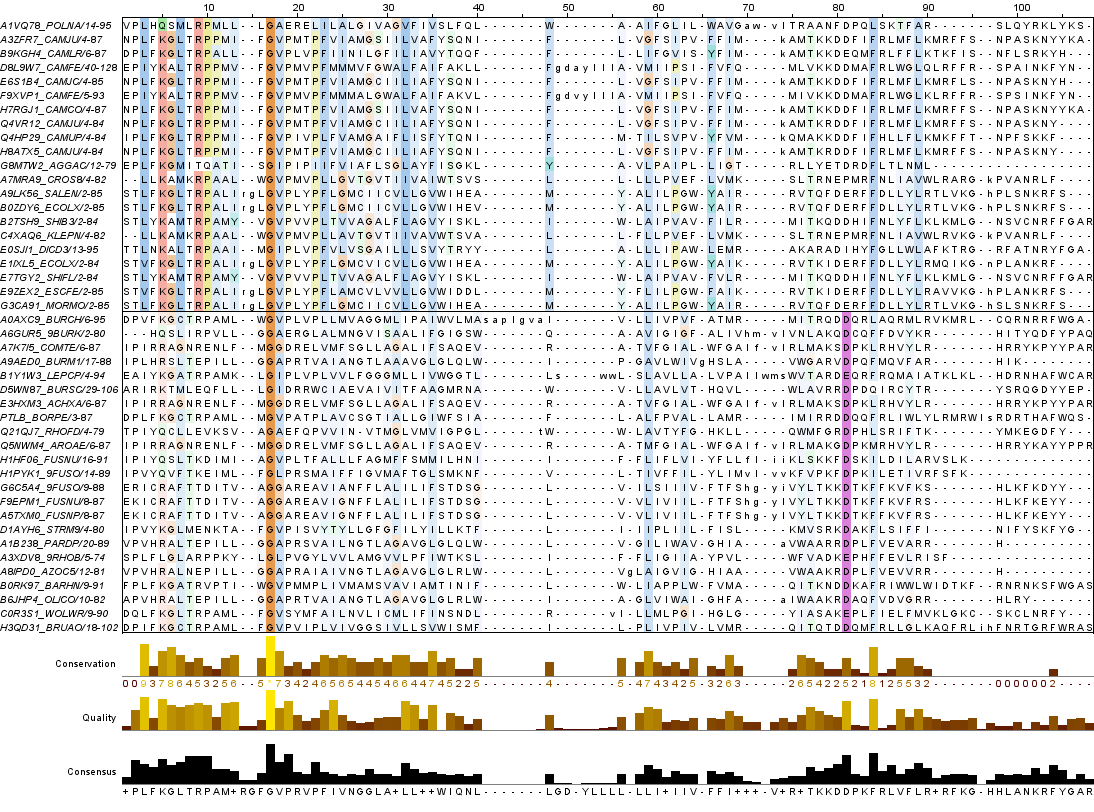
Только он не работает, поэтому вытащим последовательности вручную. Полученный файл с выравниваниями aln_fav.fasta откроем в JalView. Удалим пустые колонки (Edit - Remove Empty columns), создадим группы, по одной на каждую архитектуру, зададим раскраску в каждой группе (ClustalX, By conservation 30 для архитектуры VirB3, CagE_TrbE_VirB, AAA_12 и 20 для VirB3). Полученное выравнивание можно увидеть на рисунке 1, а так же в файле evol_fav.jar
В домене хорошо видны несколько консервативных участков. Но в целом, мы видим довольно много неконсервативных участков.
Построение филогенетического дерева по выравниванию представителей домена
Цифрой "3" начинаются последовательности, принадлежащие организмам, которые имеют трехдоменную архитектуру (VirB3, CagE_TrbE_VirB, AAA_10), цифрой "1" - однодоменную (VirB3).
Таблица 1. Сокращение подтаксонов
Подтаксон |
Сокращение |
Alphaproteobacteria | A |
Betaproteobacteria | B |
Epsilonproteobacteria | E |
Fusobacteriales | F |
Gammaproteobacteria | G |
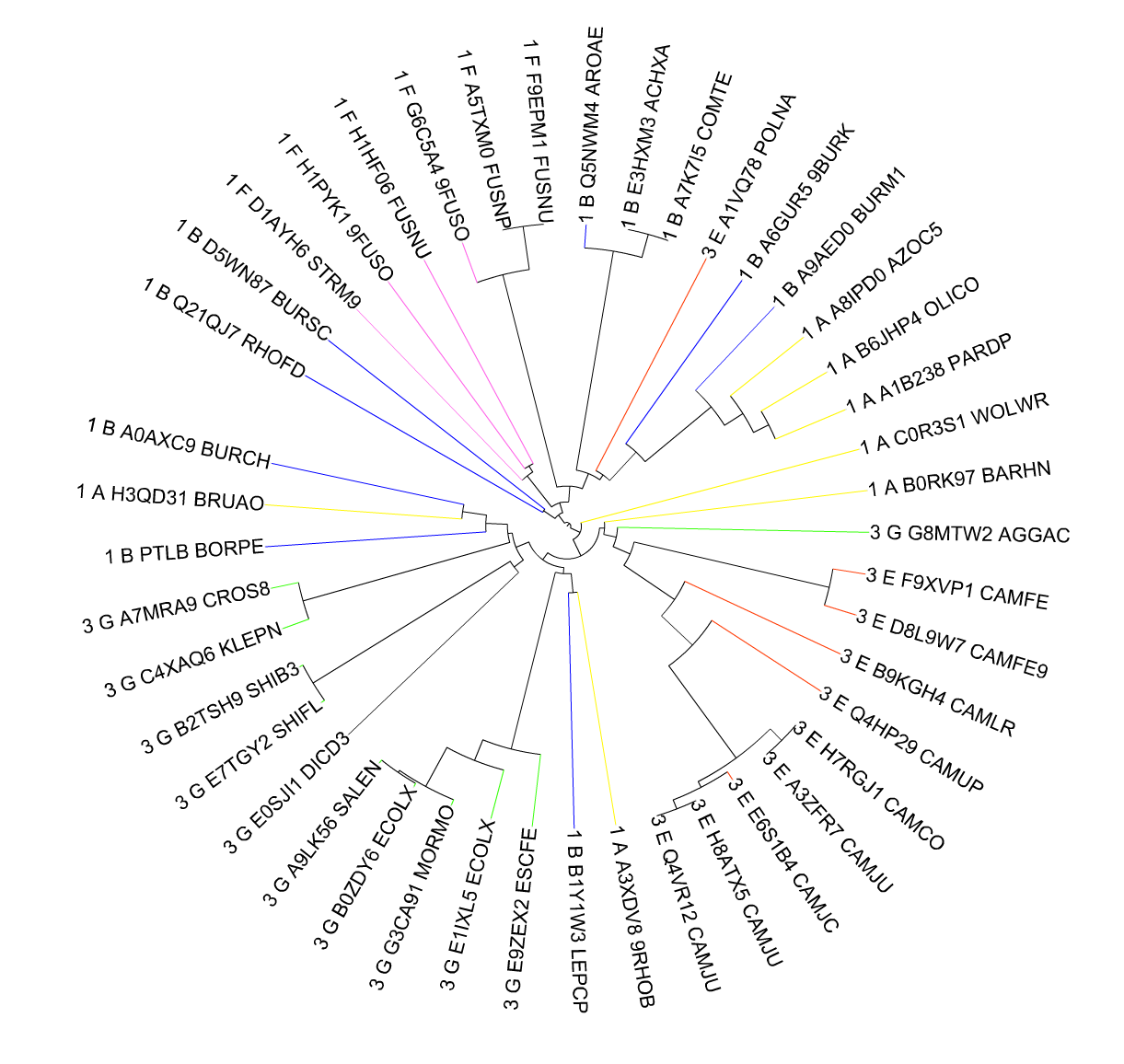
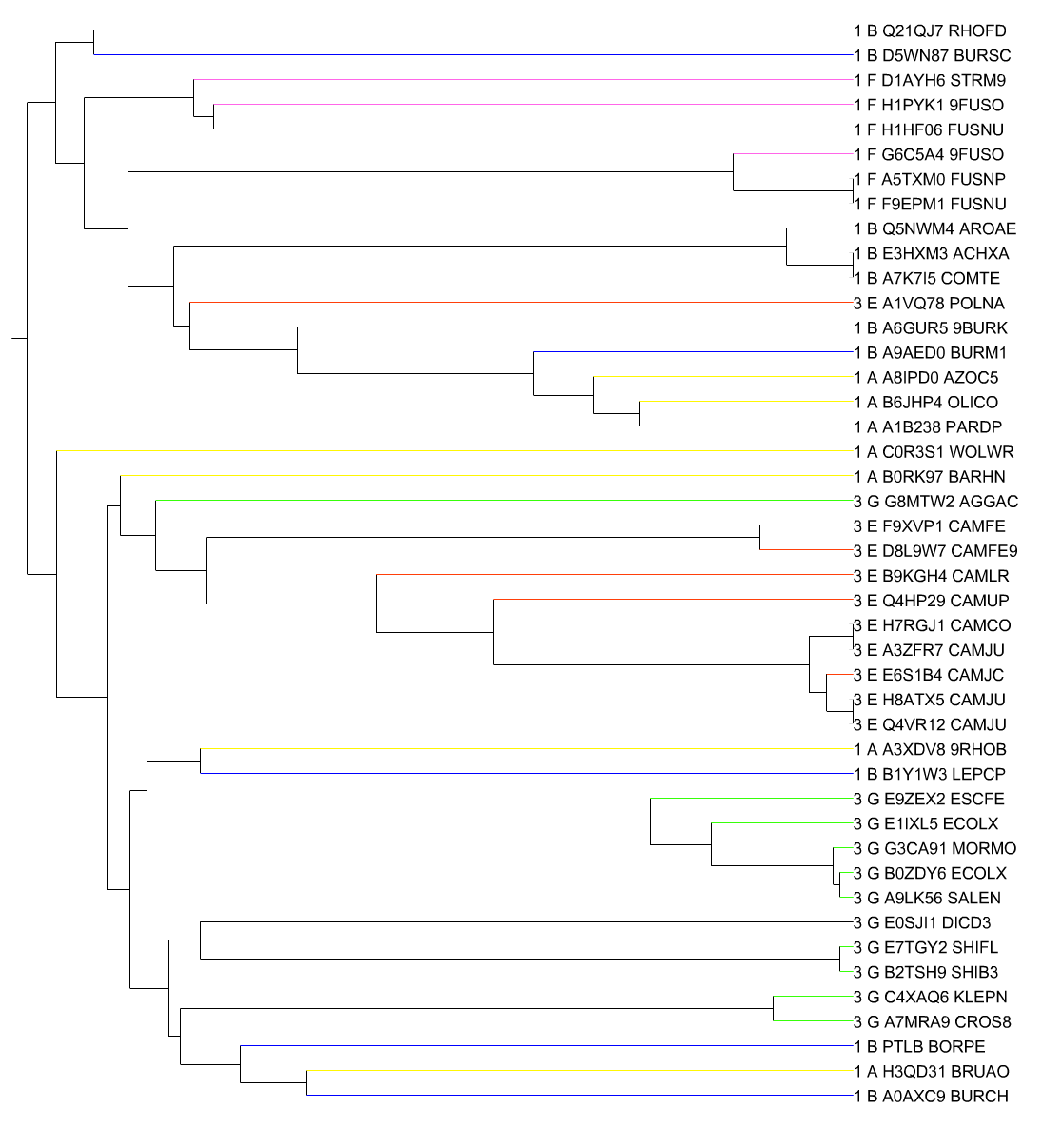
Отформатированные последовательности с удобными именами будем использвать для построения филогенетического дерева в JalView методом Neighbor-Joining Using % Identity.
(Увеличенное изображение при клике на картинке)

Скобочная формула дерева представлена в файле tree.nwk.
С использованием сервиса ITOL была создана следующая окраска листьев:
3_E красный
3_G зеленый
1_В синий
1_F розовый
1_А желтый
Было построено еще одно дерево методом UPGMA в MEGA (предполагает молекулярные часы, строит укоренённое дерево) с параметрами по умолчанию.. Оно так же было визуализированно в iTOL. Круглое дерево для этого метода представлено на рисунке 5.
Рыжим цветом отмечены 3E последовательности, фиолетовым - 1F, синим - 3G.
(Увеличенное изображение при клике на картинке)
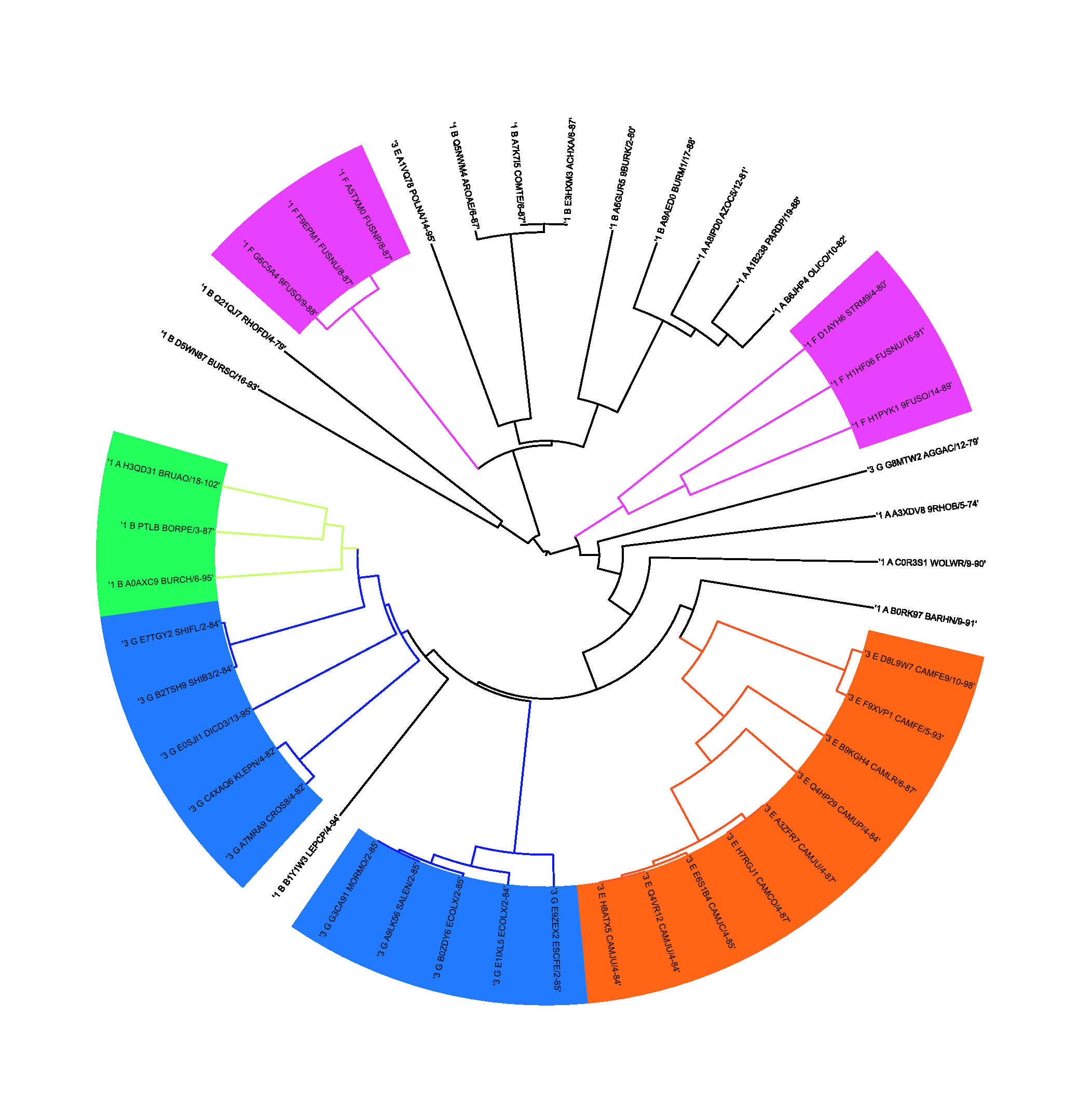
Исходя из приведнных деревьев можно предположить гипотезу об эволюции архитектур с выбранным доменом. Домены архитектур хорошо различаются программой. На получившихся деревьях видно, что у предка сначала произошло разделение на однодоменные, а потом на трехдоменные архитектуры. Домены подсемейства 3E, 3G образовывают хорошо различимые отдельные клады.
Источники информации:
-
Shirasu K1, Kado CI, Membrane location of the Ti plasmid VirB proteins involved in the biosynthesis of
a pilin-like conjugative structure on Agrobacterium tumefaciens, FEMS Microbiol Lett (1993).
Ссылка на текст статьи. PMID: 8405938 -
Alt-Morbe J1, Stryker JL, Fuqua C, Li PL, Farrand SK, Winans SC, The conjugal transfer system of Agrobacterium
tumefaciens octopine-type Ti plasmids is closely related to the transfer system of an IncP plasmid and distantly
related to Ti plasmid vir genes, J Bacteriol (1996)
Ссылка на текст статьи. PMID: 8763954