Множественное выравнивание
1. Создание репрезентативной выборки гомологов белка YXIM_BACSU
Для того, чтобы создать выборку гомологов белка yxiM_bacsu, проще всего воспользоваться программой BLAST.
Найдём последовательности белков прокариот, наиболее близкие к последовательности исходного белка, исключив из поиска домен Eukarya и филум Firmicutes, добавив их названия в поле Organism и поставив галочку напротив exclude. Таким образом, находки будут только из прокариот, причём без филума, в который входит Bacillus subtilis - там были бы находки с очень большим весом, что исказило бы результаты поиска. Для начала значения Expect threshold и Max target sequences выставим на 1 и 500 соответственно.
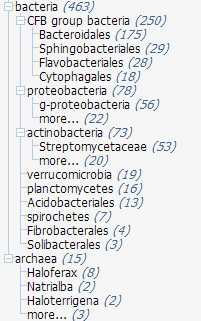
В выдаче находка под номером 478 имеет значение e-value 1e-04, ниже следуют не очень хорошие выравнивания, ограничимся первыми 478 хитами: запустим поиск снова, на этот раз со значением Expect threshold 1e-04. С помощью выдачи GenBank получим дерево таксонов, к которым принадлежат организмы, в которых обнаружены гомологи (см. рис.1), и создадим файл с последовательностями отобранных гомологов и исходного белка.
|
| Рис.1. Дерево таксонов организмов, в которых найдены гомологи белка yxiM_bacsu, отобранных для репрезентативной выборки |
По такому же принципу составим выборку гомологов из эукариотических организмов (fasta-файл с последовательностями можно найти здесь). Окончательные параметры поиска для составления обеих выборок представлены в таблице 1.
Таблица 1. Параматры BLAST, использованные для составления выборки
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количество хитов |
| По прокариотам | blastp (protein blast) |
RefSeq | exclude Eukarya exclude Firmicutes |
1e-04 | 500 |
| По эукариотам | blastp (protein blast) |
RefSeq | Eukarya | 7e-04 | 50 |
Нагляднее всего представить выборку в виде таблицы с пречислением таксонов, к которым относятся отобранные гомологи (см. таблицу 2).
Таблица 2. Встречаемость гомологов белка yxiM в различных таксонах
| Домен | Филум/Царство | Название организма | Идентификатор белка |
| Archaea | Euryarchaeota | Halopiger xanaduensis SH-6 | YP_004585795.1 |
| Haloferax volcanii DS2 | YP_003534586.1 | ||
| Bacteria | Acidobacteria group | Arthrobacter sp. SJCon | ZP_21140790.1 |
| Brachybacterium faecium DSM 4810 | YP_003156277.1 | Actinobacteria | Streptomyces sp. PAMC26508 | YP_007863706.1 |
| Streptomyces venezuelae ATC 10712 | YP_006882151.1 | ||
| Arthrobacter phenanthrenivorans Sphe3 | YP_004243075.1 | ||
| CFB group bacteria | Spirosoma linguale DSM 74 | YP_003386080.1 | |
| Niastella koreensis GR20-10 | YP_005010948.1 | ||
| Echinicola vietnamensis DSM 17526 | YP_007225329.1 | ||
| Flavobacterium johnsoniae UW101 | YP_001196553.1 | ||
| Bacteroides thetaiotaomicron VPI-5482 | NP_813084.1 | ||
| Alistipes sp. JC136 | ZP_10996974.1 | ||
| Bacteroides vulgatus ATCC 8482 | YP_001297532.1 | ||
| Bacteroides sp. HPS0048 | WP_002559351.1 | ||
| Firmicutes | Bacillus subtilis subsp. subtilis 6051-HGW | P42304 | |
| Planctomycetes | Planctomyces brasiliensis DSM 5305 | YP_004272342.1 | |
| Planctomyces maris | WP_002648236.1 | ||
| Proteobacteria | Xanthomonas axonopodis Xac29-1 | YP_007634668.1 | |
| Xanthomonas axonopodis pv. citrumelo F1 | YP_004849764.1 | ||
| Caulobacter segnis ATCC 21756 | YP_003593797.1 | ||
| Spirochaetes | Treponema saccharophilum | WP_002704685.1 | |
| Veruccomicrobia | Opitutus terrae PB90-1 | YP_001818241.1 | |
| Opitutaceae bacterium TAV5 | ZP_09594401.1 | ||
| Eukarya | Fungi | Nectria haematococca mpVI 77-13-4 | XP_003041737.1 |
| Magnaporthe oryzae 70-15 | XP_003711617.1 | ||
| Sordaria macrospora k-hell | XP_003351132.1 | ||
| Aspergillus oryzae RIB40 | XP_001820336.1 | ||
| Aspergillus niger CBS 513.88 | XP_001402292.2 | ||
| Aspergillus oryzae RIB40 | XP_001823387.1 | ||
| Aspergillus oryzae RIB40 | XP_001822334.1 | ||
| Fusarium graminearum PH-1 | XP_385024.1 | ||
| XP_383706.1 | |||
| Schizophyllum commune H4-8 | XP_003038737.1 | ||
| Tuber melanosporum Mel28 | XP_002837400.1 | ||
| Podospora anserina S mat+ | XP_001906534.1 | ||
| Penicillium chrysogenum Wisconsin 54-1255 | XP_002559978.1 | ||
| Talaromyces stipitatus ATCC 10500 | XP_002486322.1 | ||
| Neurospora crassa OR74A | XP_958094.1 | ||
| Phaeosphaeria nodorum SN15 | XP_001798248.1 | ||
| Neosartorya fischeri NRRL 181 | XP_001261200.1 | ||
| Aspergillus fumigatus Af293 | XP_756059.1 | ||
| Sclerotinia sclerotiorum 1980 | XP_001587019.1 | ||
| Brachybacterium_faecium_DSM_4810 | YP_003156277.1 | ||
| Viridiplantae | Ricinus communis | XP_002537810.1 |
2. Множественное выравнивание гомологов белка YXIM_BACSU
Создав fasta-файл с последовательностями гомологов и исходного белка, используем его в качестве входных данных для программы выравнивания на сайте UniProt. Сохраним выравнивание в формате fasta, чтобы редактировать его через Jalview. Там добавим строки STRUCTURE (с отметками α-спиарлей и β-листов), BLOCKS (где отмечены блоки выравнивания), LIGAND (где отмечены аминокислотные остатки, связывающие лиганд) и ACT SITE (где отмечены аминокислотные остатки, входящие в активный сайт по данным записи белка yxiM в базе данных SRS).
Изображение выравнивания представлено на рисунке 2.

|
| Рис.2. Изображение выравнивания последовательностей гомологов белка yxiM. В строке BLOCKS буквами B обозначены блоки, в строке LIGAND буквой М отмечены аминокислотные остатки, связывающиеся с ионом марганца, буквой S - с сульфат-ионом. Аминокислотные остатки, входящие в активный сайт обозначены буквой А в строке ACT SITE. Полноразмерное изображение можно найти здесь. |
3. Результаты анализа множественного выравнивания гомологов белка YXIM_BACSU
В получившемся выравнивании можно выделить хорошо выровненный участок (на позициях 190-540) и плохо выровненные части (всё остальное). На этом участке расположена большая часть блоков[1], они примерно совпадают с элементами вторичной структуры. Колонки гэпов наоборот, расположены в промежутках между α-спиарлями и β-листами, никогда полностью не совпадают с ними. Так как вторичная структура гомологов консервативна на этом участке, можно предположить, что здесь находится функционально важный домен.[2]
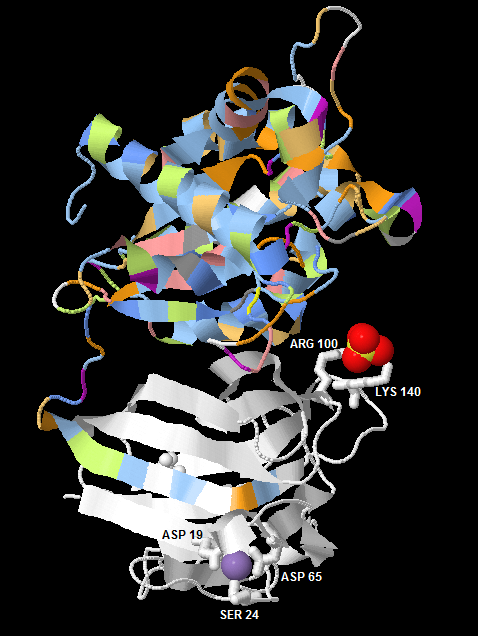
PDB-файл белка yxiM_bacsu содержит информацию о двух лигандах - ионе марганца и сульфат-ионе (подробности на этой странице). Однако остатки, связывающие эти лиганды совсем не консервативны и даже отсутствуют у большинства гомологов (см. рис.3). Отсюда можно сделать два вывода. Либо N-концевой участок белка, где они расположены, является доменом, функционирующим только у B. subtilis, и они всё же играют какую-то роль. Либо эти два иона на самом деле не являются лигандами белка, они нужны были для кристаллизации при исследовании структуры и попали в запись по ошибке. Вторая версия выглядит более вероятной.
|
| Рис.3. Вторичная структура белка yxiM. Толстым wireframe выделены остатки, связывающие лиганды. Атомы лигандов покрашены стандартными цветами и изображены шарообразно. Наиболее консервативные аминокислотные остатки окрашены наиболее интенсивно, малоконсервативные - бледнее. Остатки, связывающие лиганд, окрашены белым, из-за отсутствия консервативности. |
В то же время, аминокислотные остатки, входящие в активный сайт белка, более консервативны. Серин 186 (220-ая позиция в выравнивании) сохраняется у всех гомологов, глутамат 354 (480-ая позиция в выравнивании) у половины и у нескольких заменён на сходный по свойствам аспартат.
Хуже всего выровнены N-концы гомологов, потому что там расположены сигнальные последовательности, которые отличаются у различных организмов. Плохо выровнен и С-конец после описанного выше домена. Вероятно, этот участок маловажен для работы белка.
Примечания
[1] - Основными критериями для выделения блоков служили: а) отсутствие гэпов на всей толщине выравнивания; б) высокая консервативность.
[2] - В описании белка на сайте PDB (см. по ссылке)
указано, что это домен SGNH гидролаз.