A- и В- формы ДНК. Структура РНК
Получение моделей структур A-, B- и Z-формы ДНК
Для дальнейшего изучения A-, B- и Z-форм дуплекса ДНК построим их модели с помощью программы fiber пакета 3DNA. Доступ к программам пакета под операционной системой Windows получим через программу Putty, указав следующий путь:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
Получим три pdb-файла со структурами разных форм ДНК, введя три следующие команды, и указав требуемые параметры (повторяющиеся участки для A- и B-форм - ACGT - и их количество для всех трёх форм):
fiber -a gatc_a.pdb fiber -b gatc_b.pdb fiber -z gatc_z.pdb
Отображение структур с помощью JMol
Программа JMol позволяет выделять различные группировки атомов и менять их отображение, чтобы выделить на фоне остальной структуры. На рисунке 1 приведены различные варианты отображения А-формы дуплекса ДНК.
А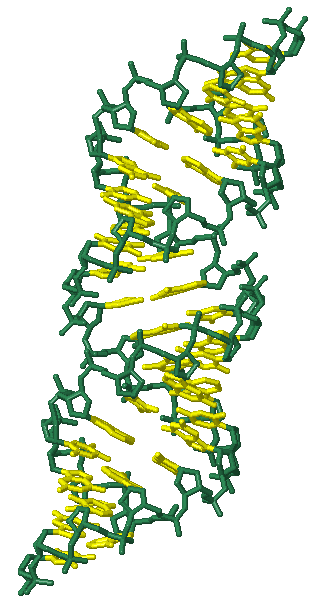
|
B
|
Рис. 1 А-форма дуплекса ДНК. A. Сахарофосфатный остов (выделен командой select mainchain) покрашен тёмно-зелёным цветом, азотистые основания - жёлтым. B. Атомы всех нуклеотидов (выделены командой select all) покрашены стандартными цветами JMol: голубой А, зелёный Т, розовый G, оранжевый С. C. Все аденины (выделены командой select A) в цепи покрашены красным. D. Атом N7 третьего гуанина (выделен командой select G3.N7) покрашен фиолетовым цветом, остальные атомы того же основания - красным. Третий гуанин является первым гуанином в последовательности. | ||
C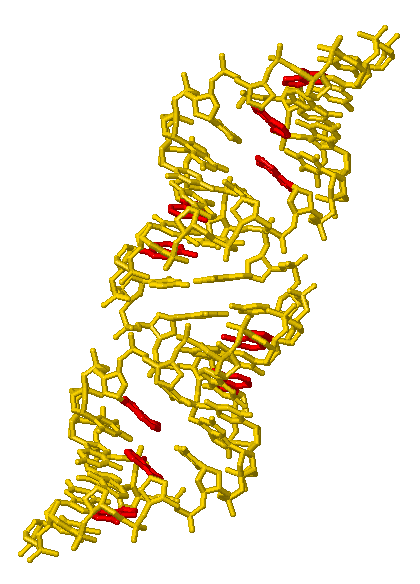
|
D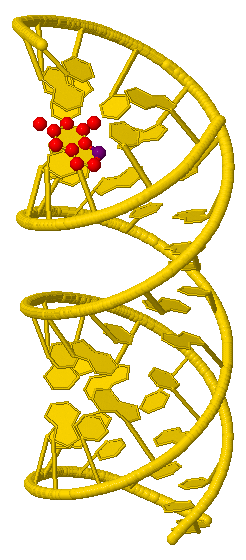
|
|||
Для изучения структуры РНК и ДНК, связанных с белком, на сайте PDB получим файлы со структурами комплексов РНК-белок 1g59 (GLUTAMYL-TRNA SYNTHETASE COMPLEXED WITH TRNA(GLU)) и ДНК-белок 1ddn (DIPHTHERIA TOX REPRESSOR (C102D MUTANT)/TOX DNA OPERATOR COMPLEX). Проверим цепи нуклеиновых кислот на наличие разрывов. Результаты представлены на рисунках 2 и 3. Для дальнейшей работы отдельно сохраним атомы ДНК и РНК (файлы по ссылкам).
|
|
|
| Рис. 2 Проволочная модель молекулы РНК 1g59. Разрывов нет. | Рис. 3 Проволочная модель молекулы ДНК 1ddn. Разрывов нет. |
Сравнение структур 3-х форм ДНК с помощью средств JMol
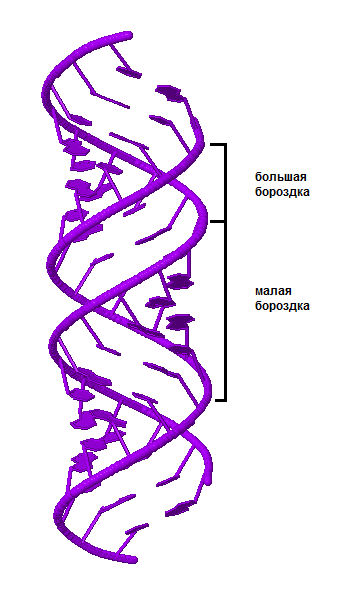
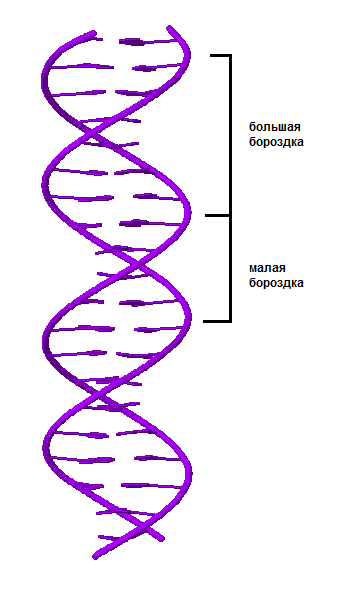
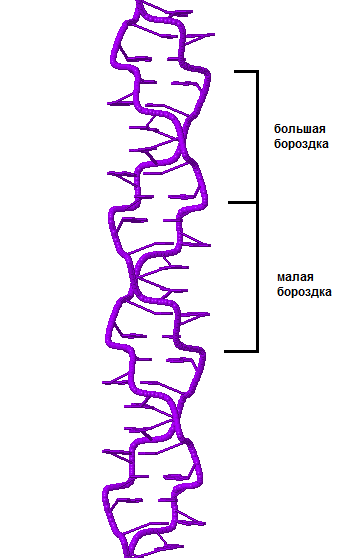
Вернёмся к полученным ранее структурам А-, В- и Z-форм ДНК. У всех трёх форм можно выделить большую и малую бороздки (см. рис. 4, 5, 6). Большая бороздка является более глубокой, она шире малой бороздки в В-форме, и уже её в А- и Z-форме ДНК.
|
|
|
||
| Рис. 4 A-форма дуплекса ДНК. Большая и малая бороздки подписаны. | Рис. 5 B-форма дуплекса ДНК. Большая и малая бороздки подписаны. | Рис. 6 Z-форма дуплекса ДНК. Большая и малая бороздки подписаны. |
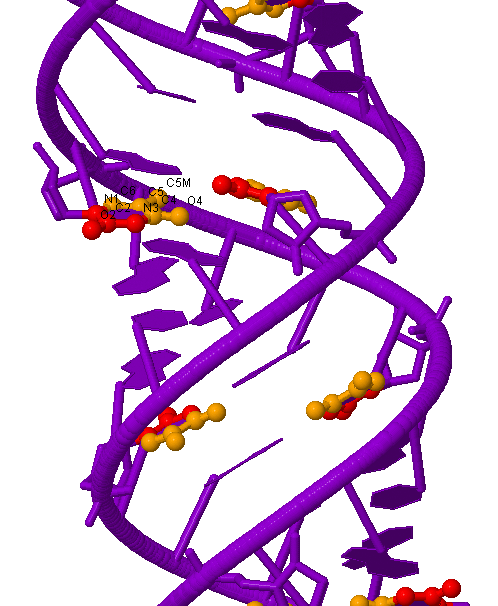
Рассмотрим положение атомов тимина относительно большой и малой бороздок А- и В- форм дуплекса (Z-форма содержит только гуанин и цитозин). На рисунках 7 и 8 показано, что и в А-, и в В-форме к большой бороздке направлены атомы C6, C5, С4, O4 и C5M, а к малой - O2, C2, N1 и N3.

|
|
|
| Рис. 7 Тимин в одной из цепей A-формы дуплекса ДНК. Все атомы основания подписаны. Те, что направлены в сторону большой бороздки окрашены оранжевым цветом, в сторону малой - красным цветом. | Рис. 8 Тимин в одной из цепей В-формы дуплекса ДНК. Все атомы основания подписаны. Те, что направлены в сторону большой бороздки окрашены красным цветом, в сторону малой - оранжевым цветом. |
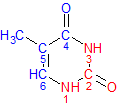
С помощью ChemSketch получим изображение основания с выделенными атомами (рис. 9).
|
Рис. 9 Тимин. Атомы основания пронумерованы, синим цветом выделены атомы, направленные в сторону большой бороздки (C6, C5, С4, O4 и C5M), красным - в сторону малой (O2, C2, N1 и N3). |
С помощью программы JMol измерим различные параметры трёх форм дуплекса ДНК. Результаты представлены в таблице 1. На рисунках 10-12 представлены изображения измерений ширины бороздок.
Таблица 1. Данные измерений трёх форм дуплекса ДНК.
| A-форма | B-форма | Z-форма | |
| Тип спирали | правая | правая | левая |
| Шаг спирали (Å) | 28,03 | 33,75 | 43,5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 7,98 (между атомами Р оснований A25 и C10) |
17,91 (между атомами Р оснований T8 и A29) |
7,2 (между атомами Р оснований G15 и G49) |
| Ширина малой бороздки | 16,97 (между атомами Р оснований C10 и C34) |
11,69 (между атомами Р оснований A29 и T16) |
16,08 (между атомами Р оснований C10 и C50) |
А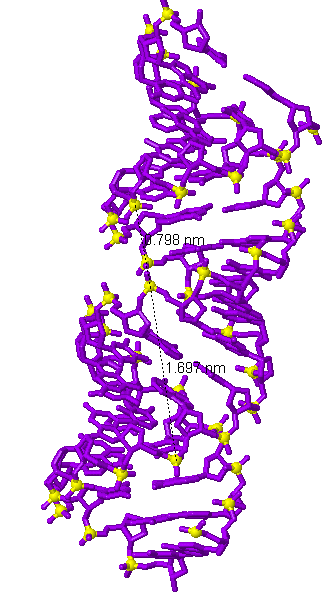
|
B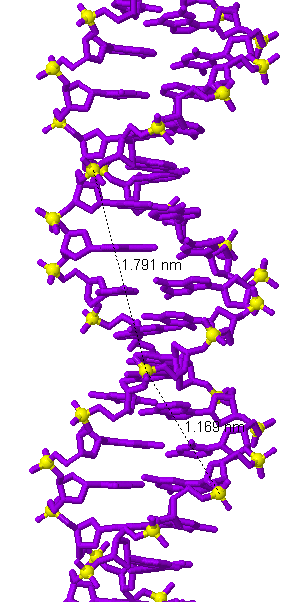
|
C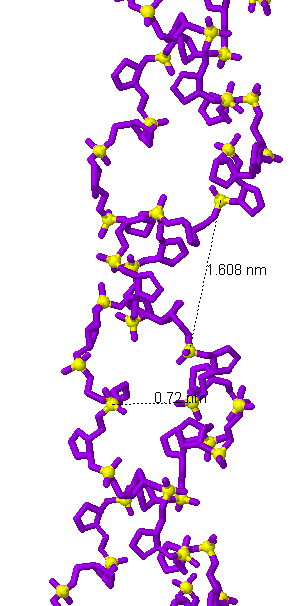
|
Рис. 10 Измерение большой и малой бороздок дуплекса ДНК. Жёлтыми шариками выделены атомы фосфора, между которыми проводилось измерение. Расстояние между атомами фосфора, расположенными на противоположных краях бороздок, отмечено пунктирной линией, над которой подписана длина в нанометрах. А. A-форма ДНК. B. В-форма ДНК. C. Z-форма ДНК. |
||
Сравним те же три структуры по ещё одному параметру - торсионным углам одного из нуклеотидов. Для измерений возьмём тимидил. Он изображён на рисунке 13 с отмеченными углами, которые были измерены. Результаты измерений приведены в таблице 2.
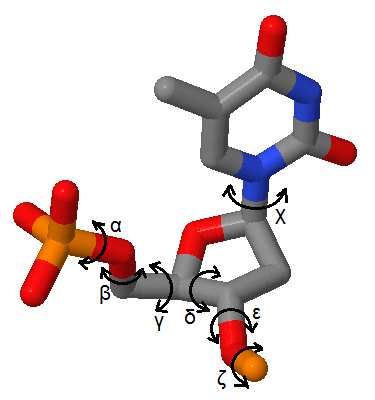
|
Рис. 13 Модель структуры тимидила. Стрелками отмечены торсионные углы, возле стрелок подписаны их названия. |
| Форма дуплекса | α | β | γ | δ | ε | ζ | χ |
| A-форма | 64,1 | 174,8 | 41,7 | 79,1 | 100,4 | -75,1 | -157,2 |
| B-форма | 85,9 | 136,4 | 31,1 | 143,4 | 105,9 | -44,7 | -98 |
Стоит отметить, что сильнее всего в А- и В-формах отличаются углы дельта и хи, что связано со степенью скрученности дуплексов.
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
С помощью команды find_pair -t 1DDN_dna_old.pdb stdout | analyze получим файл с информацией о структуре. Информация о торсионных углах и их средних значениях содержится в файле по ссылке. Нуклеотид №30 из второй цепи показывает сильное отклонение по углам альфа, бета и гамма.
Исользуем команду find_pair -d 1g59_rna_old.pdb для получения файла с информацие о водородных связях в молекуле РНК. Координаты участков, связанных водородными связями:
B:.501_:[..G]G-----C[..C]:.572_:B
B:.502_:[..G]G-*---U[..U]:.571_:B
B:.503_:[..C]C-----G[..G]:.570_:B
B:.504_:[..C]C-----G[..G]:.569_:B
B:.505_:[..C]C-----G[..G]:.568_:B
B:.506_:[..C]C-----G[..G]:.567_:B
B:.507_:[..A]A-----U[..U]:.566_:B
B:.510_:[..G]G-----C[..C]:.525_:B
B:.511_:[..U]U-----A[..A]:.524_:B
B:.512_:[..C]C-----G[..G]:.523_:B
B:.526_:[..G]G-*---A[..A]:.544_:B
B:.527_:[..C]C-----G[..G]:.543_:B
B:.528_:[..G]G-----C[..C]:.542_:B
B:.529_:[..G]G-----C[..C]:.541_:B
B:.530_:[..C]C-----G[..G]:.540_:B
B:.531_:[..C]C-----G[..G]:.539_:B
B:.532_:[..C]C-*---A[..A]:.538_:B
B:.549_:[..G]G-----C[..C]:.565_:B
B:.550_:[..G]G-----C[..C]:.564_:B
B:.551_:[..G]G-----C[..C]:.563_:B
B:.552_:[..G]G-----C[..C]:.562_:B
B:.553_:[..G]G-----C[..C]:.561_:B
B:.554_:[..U]U-*---A[..A]:.558_:B
Выделим эти участки на модели молекулы средствами Jmol и определим, какому стеблю соответствует каждый из найденных участков. Результат на рисунке 14.
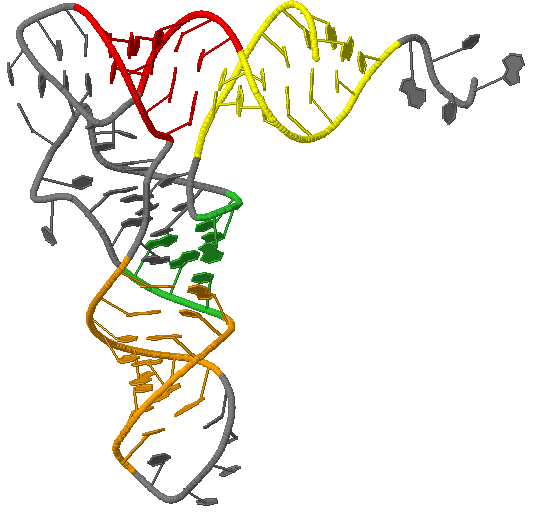
|
Рис. 14 Модель структуры тРНК. Жёлтым цветом выделен акцепторный стебель, красным - Т-стебель, зелёным - дигидроуридиновый, оранжевым - антикодоновый стебель. |
Некоторые пары оснований взаимодействуют неканонически. Всего таких пар нашлось 16. По этой ссылке приведён файл, где обозначены неканонические взаиможействия в исследуемой молекуле.
Неканонические пары, расположенные вне стеблей, стабилизируют структуру молекулы. К ним относятся пары 515-548, 517-555 и 518-556.