1. Гомологи ODO1 (2-oxoglutarate dehydrogenase E1 component) Brucella canis
AC:A9M8Q9Б
Параметры поиска Blast
База данных: UniProtKB/Swiss-prot(swissprot), Максимум находок: 100, Пороговое значение E-value: 0.05, Длина слова-якоря: 5, Матрица: BLOSUM62
Результат выдачи программы Blast - 90 находок: Текстовая выдача программы BLAST (ODO1)
Для выравнивания были отобраны 7 находок, для всех E-value= 0.0 (машинный ноль, находка достоверна), ID отобранных находок: ODO1_BRUC2;ODO1_BRUA1;ODO1_RICFE;ODO1_ECOLI;ODO1_XENLA;ODO1_MOUSE;ODO1_HUMAN

Наличие консервантивных и функционально-консервативных колонок 352-362, 389-399, 319-324, 464-469, где последовательности практически полностью идентичны, свидетельствует о гомологии белков. Такие участки, вероятно, необходимы для правильной каталитической активности этого фермента, и действительно, в записи о белке в Uniprot указано, что участки 352-366 и 464-469 являются сайтами связывания катиона Mg2+ и тиаминпирофосфата - ключевого кофактора и кофермента ODO1, и поэтому сильно подвержены отбору. Несмотря на то, что участки 378-387, 404-414, 439-456, состоящие в основном из несовпадающих аминокислотных остатков и инделей, различаются между бактериями и позвоночными животными и являются вариабельными в разных таксонах (таких участков достаточно много ещё потому, что белок имеет большую длину в примерно 1000 а.о.), выбранные белки гомологичны.
2. Гомологи зрелого вирусного белка
Поисковый запрос: (protein_name:polyprotein) AND (reviewed:true) AND (taxonomy_id:11676). Было найдено 125 записей полипротеинов вируса иммунодефицита человека (ВИЧ). Для изучения был выбран первая запись - Envelope glycoprotein gp160.
ID: ENV_HV192
AC: O12164
Название вируса: Human immunodeficiency virus type 1 group M subtype C (HIV-1)
Название зрелого белка: Surface protein gp120, координаты: 32..504, ссылка на последовательность вырезанного белка gp120: Последовательность gp120
Параметры поиска Blast
База данных: UniProtKB/Swiss-prot(swissprot), Максимум находок: 100, Пороговое значение E-value: 0.05, Длина слова-якоря: 5, Матрица: BLOSUM62
Результат выдачи программы Blast - 77 находок: Текстовая выдача программы BLAST (gp120)
Для выравнивания были отобраны 7 находок, для первых шести E-value= 0.0, для последней E-value=3e-158, ID гомологов: ENV_HV192; ENV_HV1ET; ENV_HV1VI; ENV_HV1H3; ENV_SIVCZ; ENV_HV1YF; ENV_HV1AN
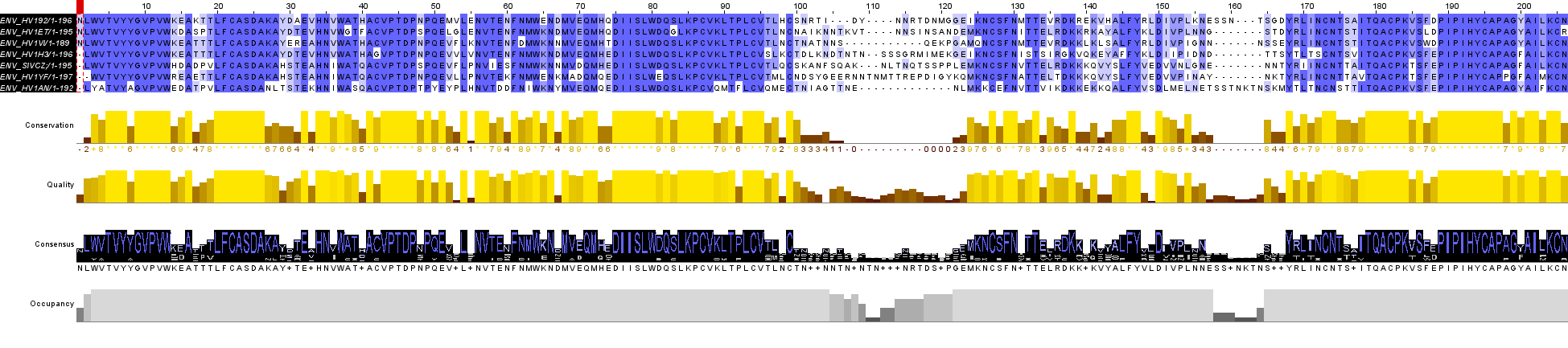
gp120 - это внешний поверхностный гликопротеин, который отвечает за связывание вируса с клетками-мишенями, а именно с клетками, имеющими на своей поверхности белок CD4 (в первую очередь, Т-хелперы, макрофаги, дендритные клетки). Высоко консервативные участки 1-13, 20-28, 41-47, 75-95, 125-130, 178-184б 189-199, где последовательности практически полностью идентичны, свидетельствуют о гомологии. Наличие таких участков, вероятно, необходимо для поддержания пространственной структуры белка и правильного, высоко специфичного связывания с CD4 клеток-мишеней. Участки белка 100-121, 157-167, состоящие в основном из несовпадающих аминокислотных остатков и инделей, скорее всего, обладают высокой вариабельностью (процент идентичности для всех найденных белков не превосходил 75%). Высокая вариабельность gp120 представляет собой серьезную проблему для разработки эффективной вакцины, поэтому исследования направлены на поиск более консервативных эпитопов на gp120.
3. Исследование зависимости E-value от объёма банка
При повторении предыдущего поиска, оставив те же параметры BLAST, но ограничив поиск вирусами (Viruses), количество находок не изменилось (77), но изменились E-value. Новое значение E-value для находки из ENV_HV1AN равно 1e-159 (немного меньше 3e-158).
Значение E-value вычисляется по следующей формуле, где m - длина исходной последовательности, n - размер базы данных B- вес в битах:
Так как m, B не изменяются, то n_вир / n_общ = E - value_вир / E - value_общ = (1 x 10^-159) / (3 x 10^-158) = 0.033 = 3.3%