Филогенетическое устройство отобранных бактерий
Бактерии, отобранные в прошлом задании о филогенетических деревьях имеют филогенетическую структуру, представленную на Рисунке 1.
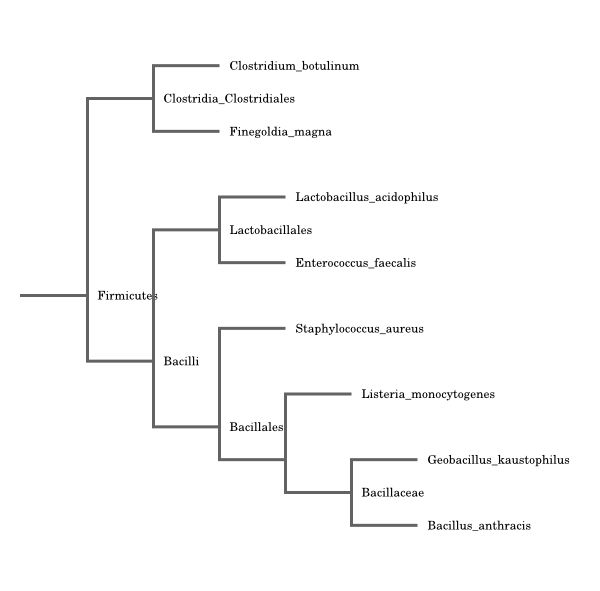
Рисунок 1. Филогенетическое устройство отобранных бактерий
Построено при помощи сервиса TreeVector
Филогенетическое дерево, представленное на Рисунке 1 имеет следующую скобочную формулу:
Скобочная формула дерева на Рисунке 1
((Clostridium_botulinum,Finegoldia_magna)Clostridia_ Clostridiales,((Lactobacillus_acidophilus,Enterococcus_faecalis)Lactobacillales,(Staphylococcus_aureus,(Listeria_monocytogenes,(Geobacillus_kaustophilus,Bacillus_anthracis)Bacillaceae))Bacillales)Bacilli)Firmicutes;
Выбор белков для работы
Для работы отобраны белки с функцией, представленной в Таблице 1.| Функция | Мнемоника |
| Рибосомный белок S2 | RS2 |
Далее при помощи программы seqret из пакета EMBOSS были  получены последовательности отобранных белков.
После чего было проведено множественное выравнивание при помощи программы muscle, результат которого получен
получены последовательности отобранных белков.
После чего было проведено множественное выравнивание при помощи программы muscle, результат которого получен  в fasta-формате. Представление данных выравнивания осуществлено в программе Jalview,
в fasta-формате. Представление данных выравнивания осуществлено в программе Jalview,  файл проекта. Так же результат представлен на Рисунке 2.
файл проекта. Так же результат представлен на Рисунке 2.
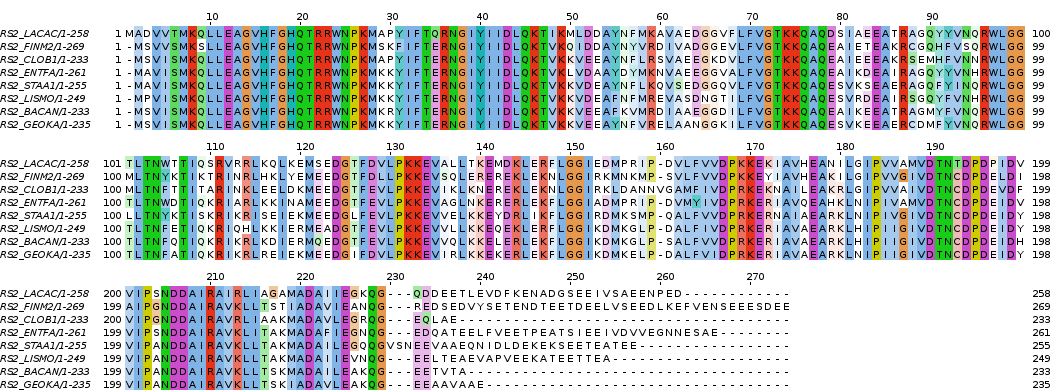
Рисунок 2. Множественное выравнивание отобранных белков. Раскраска по схеме clustalw по проценту идентичности с пороговым параметром 30%. Изображение получено в программе Jalview.
Обзор диагностических позиций выравнивания
По данным выравнивания и данным о филогении можно сделать вывод о диагностических позиций выравнивания. Полученные данные о диагностических позициях представлены в Таблице 2.
| Аминокислота | Диагностическая позиция | Таксон |
| V | 223 | Clostridia Clostridiales |
| G | 204 | Clostridia Clostridiales |
| M | 101 | Clostridia Clostridiales |
| V | 67 | Lactobacillales |
| W | 105 | Lactobacillales |
| M | 88 | Lactobacillales |
Анализ деревьев построенных при помощи JalView.
По данным выравнивания белков построены четыре дерева по следующим алгоритмам:
средняя дистанция по проценту идентичности, Рисунок 3.
средняя дистанция по весу BLOSUM62, Рисунок 4.
соседство по проценту идентичности, Рисунок 5.
соседство по весу BLOSUM62, Рисунок 6.
Отдельно следует отметить дерево, представленное на Рисунке 7, построенное в программе Mega по алгоритму Maximum Parsimony.
Далее сравнение топологии деревьев на Рисунке 3-7 с топологией дерева на Рисунке 1 идет предполагая эквивалентность ветвей полученных по выравниванию белков и ветвей таксонометрического дерева, т.к. в описании различий или сходств ветви называются по мнемонике вида.
Сравнение проводилось при помощи программы на Python с использованием сторонней библиотеки ETE для работы с филогенетическими деревьями. Код программы представлена в Таблице 3.
compare_tree.py
 для сравнения топологии деревьев.
для сравнения топологии деревьев.
Рисунок 3. Дерево белков бактерий, построенное при помощи JalWiev (средняя дистанция по проценту идентичности).
Дерево представленное на Рисунке 3 (Второе в сравнении) имеет следующие особенности по отношению к филогенетическому дереву на Рисунке 1. (Первое в сравнении)
Первое дерево
/-CLOB1
/-|
| \-FINM2
|
--| /-LACAC
| /-|
| | \-ENTFA
\-|
| /-STAA1
\-|
| /-LISMO
\-|
| /-GEOKA
\-|
\-BACAN
Второе дерево
/-LACAC
|
--| /-FINM2
| |
| | /-CLOB1
\-| |
| | /-GEOKA
| | /-|
\-| /-| \-BACAN
| | |
| /-| \-LISMO
| | |
\-| \-STAA1
|
\-ENTFA
Нетривиальные ветви первого дерева
{'BACAN', 'GEOKA'}vs{'FINM2', 'LACAC', 'ENTFA', 'STAA1', 'CLOB1', 'LISMO'}
{'CLOB1', 'FINM2'}vs{'BACAN', 'GEOKA', 'LACAC', 'ENTFA', 'STAA1', 'LISMO'}
{'BACAN', 'GEOKA', 'LISMO'}vs{'LACAC', 'CLOB1', 'ENTFA', 'STAA1', 'FINM2'}
{'ENTFA', 'LACAC'}vs{'BACAN', 'GEOKA', 'FINM2', 'STAA1', 'CLOB1', 'LISMO'}
{'BACAN', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'ENTFA', 'FINM2'}
Нетривиальные ветви второго дерева
{'BACAN', 'CLOB1', 'ENTFA', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'FINM2'}
{'BACAN', 'GEOKA'}vs{'FINM2', 'LACAC', 'ENTFA', 'STAA1', 'CLOB1', 'LISMO'}
{'BACAN', 'ENTFA', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'FINM2'}
{'BACAN', 'GEOKA', 'LISMO'}vs{'LACAC', 'CLOB1', 'ENTFA', 'STAA1', 'FINM2'}
{'BACAN', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'ENTFA', 'FINM2'}
Ветви которые есть в Первом дереве, но отсутствуют во Втором
{'CLOB1', 'FINM2'}vs{'BACAN', 'GEOKA', 'LACAC', 'ENTFA', 'STAA1', 'LISMO'}
{'ENTFA', 'LACAC'}vs{'BACAN', 'GEOKA', 'FINM2', 'STAA1', 'CLOB1', 'LISMO'}
Ветви которые есть во Втором дереве, но отсутствуют в Первом
{'BACAN', 'CLOB1', 'ENTFA', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'FINM2'}
{'BACAN', 'ENTFA', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'FINM2'}
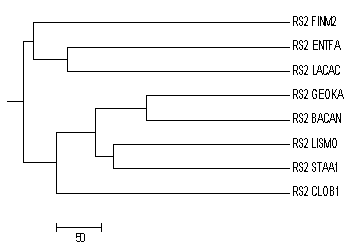
Рисунок 4. Дерево белков бактерий, построенное при помощи JalWiev (средняя дистанция по весу BLOSUM62).
Дерево представленное на Рисунке 4 (Второе в сравнении) имеет следующие особенности по отношению к филогенетическому дереву на Рисунке 1. (Первое в сравнении)
Первое дерево
/-CLOB1
/-|
| \-FINM2
|
--| /-LACAC
| /-|
| | \-ENTFA
\-|
| /-STAA1
\-|
| /-LISMO
\-|
| /-GEOKA
\-|
\-BACAN
Второе дерево
/-FINM2
/-|
| | /-ENTFA
| \-|
| \-LACAC
|
--| /-GEOKA
| /-|
| | \-BACAN
| /-|
| | | /-LISMO
\-| \-|
| \-STAA1
|
\-CLOB1
Нетривиальные ветви первого дерева
{'BACAN', 'GEOKA'}vs{'FINM2', 'LACAC', 'ENTFA', 'STAA1', 'CLOB1', 'LISMO'}
{'CLOB1', 'FINM2'}vs{'BACAN', 'GEOKA', 'LACAC', 'ENTFA', 'STAA1', 'LISMO'}
{'BACAN', 'GEOKA', 'LISMO'}vs{'LACAC', 'CLOB1', 'ENTFA', 'STAA1', 'FINM2'}
{'ENTFA', 'LACAC'}vs{'BACAN', 'GEOKA', 'FINM2', 'STAA1', 'CLOB1', 'LISMO'}
{'BACAN', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'ENTFA', 'FINM2'}
Нетривиальные ветви второго дерева
{'BACAN', 'GEOKA'}vs{'FINM2', 'LACAC', 'ENTFA', 'STAA1', 'CLOB1', 'LISMO'}
{'ENTFA', 'LACAC'}vs{'BACAN', 'GEOKA', 'FINM2', 'STAA1', 'CLOB1', 'LISMO'}
{'LISMO', 'STAA1'}vs{'BACAN', 'GEOKA', 'FINM2', 'LACAC', 'ENTFA', 'CLOB1'}
{'ENTFA', 'FINM2', 'LACAC'}vs{'LISMO', 'BACAN', 'CLOB1', 'STAA1', 'GEOKA'}
{'BACAN', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'ENTFA', 'FINM2'}
Ветви которые есть в Первом дереве, но отсутствуют во Втором
{'CLOB1', 'FINM2'}vs{'BACAN', 'GEOKA', 'LACAC', 'ENTFA', 'STAA1', 'LISMO'}
{'BACAN', 'GEOKA', 'LISMO'}vs{'LACAC', 'CLOB1', 'ENTFA', 'STAA1', 'FINM2'}
Ветви которые есть во Втором дереве, но отсутствуют в Первом
{'LISMO', 'STAA1'}vs{'BACAN', 'GEOKA', 'FINM2', 'LACAC', 'ENTFA', 'CLOB1'}
{'ENTFA', 'FINM2', 'LACAC'}vs{'LISMO', 'BACAN', 'CLOB1', 'STAA1', 'GEOKA'}
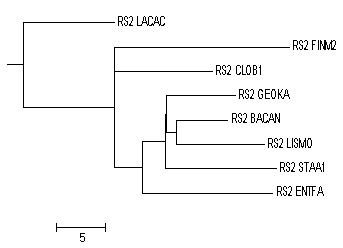
Рисунок 5. Дерево белков бактерий, построенное при помощи JalWiev (соседство по проценту идентичности).
Дерево представленное на Рисунке 5 (Второе в сравнеии) имеет следующие особенности по отношению к филогенетическому дереву на Рисунке 1. (Первое в сравнении)
Первое дерево
/-CLOB1
/-|
| \-FINM2
|
--| /-LACAC
| /-|
| | \-ENTFA
\-|
| /-STAA1
\-|
| /-LISMO
\-|
| /-GEOKA
\-|
\-BACAN
Второе дерево
/-LACAC
|
--| /-FINM2
| |
\-| /-CLOB1
| |
| | /-GEOKA
\-| /-|
| | | /-BACAN
| /-| \-|
| | | \-LISMO
\-| |
| \-STAA1
|
\-ENTFA
Нетривиальные ветви первого дерева
{'BACAN', 'GEOKA'}vs{'FINM2', 'LACAC', 'ENTFA', 'STAA1', 'CLOB1', 'LISMO'}
{'CLOB1', 'FINM2'}vs{'BACAN', 'GEOKA', 'LACAC', 'ENTFA', 'STAA1', 'LISMO'}
{'BACAN', 'GEOKA', 'LISMO'}vs{'LACAC', 'CLOB1', 'ENTFA', 'STAA1', 'FINM2'}
{'ENTFA', 'LACAC'}vs{'BACAN', 'GEOKA', 'FINM2', 'STAA1', 'CLOB1', 'LISMO'}
{'BACAN', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'ENTFA', 'FINM2'}
Нетривиальные ветви второго дерева
{'BACAN', 'CLOB1', 'ENTFA', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'FINM2'}
{'BACAN', 'ENTFA', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'FINM2'}
{'BACAN', 'GEOKA', 'LISMO'}vs{'LACAC', 'CLOB1', 'ENTFA', 'STAA1', 'FINM2'}
{'BACAN', 'LISMO'}vs{'GEOKA', 'FINM2', 'LACAC', 'STAA1', 'ENTFA', 'CLOB1'}
{'BACAN', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'ENTFA', 'FINM2'}
Ветви которые есть в Первом дереве, но отсутствуют во Втором
{'CLOB1', 'FINM2'}vs{'BACAN', 'GEOKA', 'LACAC', 'ENTFA', 'STAA1', 'LISMO'}
{'BACAN', 'GEOKA'}vs{'FINM2', 'LACAC', 'ENTFA', 'STAA1', 'CLOB1', 'LISMO'}
{'ENTFA', 'LACAC'}vs{'BACAN', 'GEOKA', 'FINM2', 'STAA1', 'CLOB1', 'LISMO'}
Ветви которые есть во Втором дереве, но отсутствуют в Первом
{'BACAN', 'CLOB1', 'ENTFA', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'FINM2'}
{'BACAN', 'ENTFA', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'FINM2'}
{'BACAN', 'LISMO'}vs{'GEOKA', 'FINM2', 'LACAC', 'STAA1', 'ENTFA', 'CLOB1'}
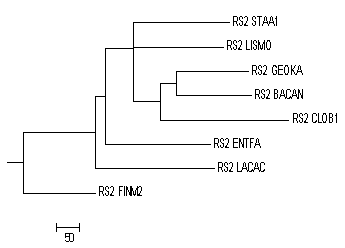
Рисунок 6. Дерево белков бактерий, построенное при помощи JalWiev (соседство по весу BLOSUM62).
Дерево представленное на Рисунке 6 (Второе в сравнении) имеет следующие особенности по отношению к филогенетическому дереву на Рисунке 1. (Первое в сравнении)
Первое дерево
/-CLOB1
/-|
| \-FINM2
|
--| /-LACAC
| /-|
| | \-ENTFA
\-|
| /-STAA1
\-|
| /-LISMO
\-|
| /-GEOKA
\-|
\-BACAN
Второе дерево
/-STAA1
|
/-| /-LISMO
| | |
| \-| /-GEOKA
| | /-|
/-| \-| \-BACAN
| | |
| | \-CLOB1
/-| |
| | \-ENTFA
--| |
| \-LACAC
|
\-FINM2
Нетривиальные ветви первого дерева
{'BACAN', 'GEOKA'}vs{'FINM2', 'LACAC', 'ENTFA', 'STAA1', 'CLOB1', 'LISMO'}
{'CLOB1', 'FINM2'}vs{'BACAN', 'GEOKA', 'LACAC', 'ENTFA', 'STAA1', 'LISMO'}
{'BACAN', 'GEOKA', 'LISMO'}vs{'LACAC', 'CLOB1', 'ENTFA', 'STAA1', 'FINM2'}
{'ENTFA', 'LACAC'}vs{'BACAN', 'GEOKA', 'FINM2', 'STAA1', 'CLOB1', 'LISMO'}
{'BACAN', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'ENTFA', 'FINM2'}
Нетривиальные ветви второго дерева
{'BACAN', 'GEOKA'}vs{'FINM2', 'LACAC', 'ENTFA', 'STAA1', 'CLOB1', 'LISMO'}
{'BACAN', 'CLOB1', 'GEOKA'}vs{'LACAC', 'LISMO', 'ENTFA', 'STAA1', 'FINM2'}
{'BACAN', 'CLOB1', 'ENTFA', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'FINM2'}
{'BACAN', 'CLOB1', 'GEOKA', 'LISMO'}vs{'LACAC', 'ENTFA', 'STAA1', 'FINM2'}
{'BACAN', 'CLOB1', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'ENTFA', 'FINM2'}
Ветви которые есть в Первом дереве, но отсутствуют во Втором
{'CLOB1', 'FINM2'}vs{'BACAN', 'GEOKA', 'LACAC', 'ENTFA', 'STAA1', 'LISMO'}
{'BACAN', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'ENTFA', 'FINM2'}
{'BACAN', 'GEOKA', 'LISMO'}vs{'LACAC', 'CLOB1', 'ENTFA', 'STAA1', 'FINM2'}
{'ENTFA', 'LACAC'}vs{'BACAN', 'GEOKA', 'FINM2', 'STAA1', 'CLOB1', 'LISMO'}
Ветви которые есть во Втором дереве, но отсутствуют в Первом
{'BACAN', 'CLOB1', 'ENTFA', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'FINM2'}
{'BACAN', 'CLOB1', 'GEOKA', 'LISMO'}vs{'LACAC', 'ENTFA', 'STAA1', 'FINM2'}
{'BACAN', 'CLOB1', 'GEOKA'}vs{'LACAC', 'LISMO', 'ENTFA', 'STAA1', 'FINM2'}
{'BACAN', 'CLOB1', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'ENTFA', 'FINM2'}
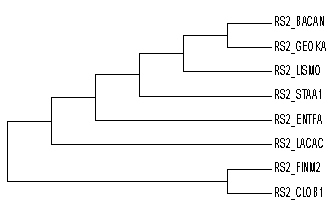
Рисунок 7. Дерево белков бактерий, построенное в программе Mega по алгоритму Maximum Parsimony.
Дерево представленное на Рисунке 7 (Второе в сравнении) имеет следующие особенности по отношению к филогенетическому дереву на Рисунке 1. (Первое в сравнении)
Первое дерево
/-CLOB1
/-|
| \-FINM2
|
--| /-LACAC
| /-|
| | \-ENTFA
\-|
| /-STAA1
\-|
| /-LISMO
\-|
| /-GEOKA
\-|
\-BACAN
Второе дерево
/-BACAN
/-|
/-| \-GEOKA
| |
/-| \-LISMO
| |
/-| \-STAA1
| |
/-| \-ENTFA
| |
--| \-LACAC
|
| /-FINM2
\-|
\-CLOB1
Нетривиальные ветви первого дерева
{'BACAN', 'GEOKA'}vs{'FINM2', 'LACAC', 'ENTFA', 'STAA1', 'CLOB1', 'LISMO'}
{'CLOB1', 'FINM2'}vs{'BACAN', 'GEOKA', 'LACAC', 'ENTFA', 'STAA1', 'LISMO'}
{'BACAN', 'GEOKA', 'LISMO'}vs{'LACAC', 'CLOB1', 'ENTFA', 'STAA1', 'FINM2'}
{'ENTFA', 'LACAC'}vs{'BACAN', 'GEOKA', 'FINM2', 'STAA1', 'CLOB1', 'LISMO'}
{'BACAN', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'ENTFA', 'FINM2'}
Нетривиальные ветви второго дерева
{'BACAN', 'ENTFA', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'FINM2'}
{'BACAN', 'GEOKA'}vs{'FINM2', 'LACAC', 'ENTFA', 'STAA1', 'CLOB1', 'LISMO'}
{'CLOB1', 'FINM2'}vs{'BACAN', 'GEOKA', 'LACAC', 'ENTFA', 'STAA1', 'LISMO'}
{'BACAN', 'GEOKA', 'LISMO'}vs{'LACAC', 'CLOB1', 'ENTFA', 'STAA1', 'FINM2'}
{'BACAN', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'ENTFA', 'FINM2'}
Ветви которые есть в Первом дереве, но отсутствуют во Втором
{'ENTFA', 'LACAC'}vs{'BACAN', 'GEOKA', 'FINM2', 'STAA1', 'CLOB1', 'LISMO'}
Ветви которые есть во Втором дереве, но отсутствуют в Первом
{'BACAN', 'ENTFA', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'FINM2'}
