| Учебный сайт Саши Погорельской |

|
Детальный анализ скрипта, выполненного на одном из практикумов.
Скрипт выполняет построение хромосомной таблицы из GBK-файла, содержащего геном бактерии Syntrophus aciditrophicus SB. Исходным является файл NC_007759.gbk. Полный текст программы, написанной на языке Python, есть здесь (скачать).
При запуске скрипта в командной строке помимо названия скрипта, необходимо указать название исходного GBK-файла. Поэтому первое, что проверяется в программе - было ли введено необходимое количество аргументов. Если пользователь ввел больше или меньше аргументов, то программа выдаст ошибку с пояснением:

При корректном запуске скрипта будет открыт файл, поданный в командной строке, для чтения. Также создастся новый файл с именем Pogorelskaya_NC_007759_genes.txt .Если файл существовал, то вся информация в нем заменяется новой. В файл записывается шапка будущей таблицы. Принято, что такая строка начинается с символа "#". Для того, чтобы с полученным файлом было удобно работать потом, разделителями между столбцами будет табулятор:
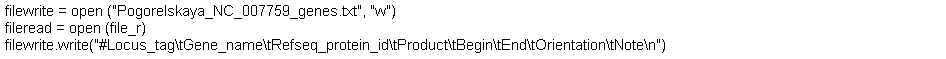
Для систематизации выполняемых команд удобно создать класс, у меня он называется ProteinCodingGene, так будет удобнее обращаться к полученным данным. Этот класс имеет 8 атрибутов, инициализирующую функцию и метод, создающий строку из всех значений атрибутов класса, разделенных табулятором. В конце скрипта будет достаточно вызвать эту функцию для всех найденных белков:

Также рационально ввести функцию, формирующую из данных исходного файла объект класса. Эта функция будет получать на вход 6 параметров и анализировать первый из них - строку, начинавшуюся в исходном файле с букв CDS (Coding DNA Sequence). Вы можете проверить, что эта строка содержит координаты гена. Если ген кодируется на комплементарной приведенной цепи, то перед координатами будет слово complement. Это условие первым проверяется в теле функции (строка разбивается на части по символу "(" и 1 часть сравнивается со словом complement). Существует несколько видов координат.
Во-первых, когда ген состоит из нескольких кусочков, расстояние между которыми не 0, у бактерий чаще всего встречается случай, когда нуклеотид входит в ген дважды, тогда его координаты будут иметь вид "join(x..y;y..z)". Таких промежутков может быть много и они могут быть разбиты на несколько строк. Это будет учитываться ниже.
Во-вторых, координаты могут быть заданы неточно, при этом перед координатой будет стоять символ ">" или "<". Для хромосомной таблицы было условлено, что все гены, кодируемые на прямой цепи будут иметь положительное значение атрибута "chain", а для комплементарной цепи - отрицательное. В случае, когда координаты заданы неточно, это будет значение 111 (-111), если координаты содержат "join", то 11 (-11), иначе 1 (-1). Фактически, все тело функции str_with_CDS состоит из анализа этих координат. Выходным ее значением является объект класса ProteinCodingGene. Эта команда занимает в коде одну строчку благодаря тому, что есть инициализирующая функция класса:

Далее следуют заготовки переменных, использующихся в цикле ниже. prodind, noteind, lineind и ind - переменные булевого типа. Изначальное их значение - False. Для первых двух оно будет меняться, если какой либо из параметров (product или note) длиннее 1 строчки, а для lineind, если координаты занимают больше 1 строчки. Значение ind является True во время анализа блока CDS. Как только он заканчивается, значение ind вновь становится False.
Также создается словарь descdict и ключам locus_tag, gene, protein_id, product, note присваиваются пустые значения. Это служит не только для того, чтобы создать эти объекты, но и в случае отсутствия соответствующих данных о гене в конечной таблице образовалась пустая ячейка. Также создается пустая стока line для использования в цикле и пустой список, в который будут записываться объекты класса ProteinCodingGene:

Далее начинается цикл, построчно анализирующий исходный файл. Первая строка в цикле обрезает все незначащие символы из начала или конца строки. Если строчка оказалась пустой, то осуществляется переход к следующей строке исходного файла. Теперь надо проверить, есть ли булевые переменные со значением True (это означает, что строка является продолжением предыдущей). При этом текущая строка будет добавлена к предыдущей. Также нужно проверить, является ли эта строка конечной (то есть заканчивается символом '"', тогда значение булевой переменной поменяется на False):

Следующая часть цикла - проверка на то, что закончился блок CDS. Это происходит, если первое слово в строке - "misc_feature" или "gene". Случай, когда эти слова включены в какое-то описание гена исключен, так как эта часть цикла вводится через оператор elif, следовательно, в нее программа попадает, если все возможные описания закончены. Если блок CDS закончился на предыдущей строке (значение ind - True), то скрипт создает новый объект в списке gene_list с помощью функции str_with_CDS. После этого обнуляются все значения словаря, строка с координатами и значение ind становится False (должно быть True только во время анализа блока CDS):

Если строка начинается с CDS, то булевым переменным ind и lineind (line - строка, содержащая координаты) присваивается значение True. С помощью цикла по количеству пробелов в исходной строке, все ее части, границами которых служат пробелы, кроме первой, записываются в строку line. Если строка не заканчивается запятой (координаты занимают ровно 1 строку), то переменной lineind присваивается значение False:

Теперь скрипт выходит в основное тело цикла. Квалификаторы записей в исходном файле начинаются с символа "/". Поэтому условием того, что строка содержит нужный квалификатор является наличие первого символа "/" и значение ind - True. После отрезания первого символа, слово, находящееся в строке до равенства должно быть ключом словаря descdict. Так как значения product и note могут занимать не одну строку, их мы рассматриваем отдельно, если строка не закончится на '"', то соответствующие булевые переменные примут значение True. В остальных случаях строка после знака равенства и без кавычек записывается как значение в словарь:

На этом цикл заканчивается, исходный файл, с которого считывались данные, закрывается. Для каждого гена из списка gene_list с помощью метода класса ProteinCodingGene скрипт записывает информацию в новый файл. После этого он закрывается:

Это был последний фрагмент моего скрипта, продуктом его работы является хромосомная таблица.