Содержит ли Swiss-Prot слово "empty"
Теоретическое предсказание
Известны частоты встречаемости каждой аминокислоты в банке Swiss-Prot. Поэтому возможно найти вероятность появления слова "empty" в Swiss-Prot, перемножив эти величины:
p = 0.0675*0.0242*0.047*0.0534*0.0292 ~ 1.2*10-7
Зная общее количество аминокислотных остатков во всём банке Swiss-Prot и вероятность появления слова, можно узнать, сколько раз должно встретиться искомое слово:
N = 1.2*10-7 * 191 670 831 = 22.95 ~ 23 раза
Встречаемость на практике
С помощью PROSITE узнаем число, соответсвующее реальному появлению слова "empty" в Swiss-Prot, для чего используем паттерн E-M-P-T-Y. Итого, 9 мотивов в 9 записях.
Такое несоответсвие может быть связано с тем, что "empty" - пустое слово, т.е. врядли содержит полезную информацию для структуры белка. А с большей вероятностью будут встречаться более информативные мотивы.
Поиск вероятных гомологов белка YojM_BACSU в банке SwissProt с помощью паттернов
Для создания сильного паттерна было использовано множественное выравнивание из практикума №8, в котором были окрашены аминокислоты с выставленым в JalView уровнем консервативности 100%. Опираясь на полученную картину и построен паттерн - результат его применения в таблице 1.
При составлении слабого паттерна ...
| Таблица 1. Результаты поиска гомологов по паттернам. | |||
|---|---|---|---|
| Характеристика паттерна | Паттерн | В скольких последовательностях банка Swiss-Prot найден мотив, удовлетворяющий паттерну? | Все ли последовательности из вашего выравнивания найдены? |
| Сильный | G-x(25,36)-[GAM]-x-H-x-H-x(14,24)-H-x(2)-[PVL]-x(3,18)-[HTE]-x(7,11)-H-x(1,8)-G-D-x(33,46)-[HD]-x(3)-[DN]-x(7,25)-[GV]-x(2)-[GK]-x-[RM]-x(3)-[GA] | 148 | нет |
| Слабый | G-x(23,38)-[GAVLIM]-x-H-x-H-x(12,26)-H-x(2)-[PVLIA]-x(2,20)-[HRTE]-x(6,12)-H-x(1,9)-G-D-x(31,48)-[HRDE]-x(3)-[DNEQ]-x(6,27)-[GAV]-x(2)-[GK]-x-[RKM]-x(3)-[GAV] | 148 | нет |
| Слабый | G-x(23,38)-[GAVLIM]-x-H-x-H-x(12,26)-H-x(2)-[PVLIA]-x(2,20)-[HRTE]-x(6,12)-H-x(0,9)-G-D-x(31,48)-[HRDE]-x(3)-[DNEQ]-x(6,27)-[GAVLI]-x(4)-[RKM]-x(3)-[GAVLI] | 156 | нет |
| Очень слабый | PS51257 and PS00008 and PS00006 and PS00005 and PS00004 | 178 (5000 находок) | нет |
Как видно, не все белки, по которым строились паттерны, найдены (как минимум не хватает Ixodes scapularis). Это связано с тем, что данные об исходных белках брались из банка данных Reference Sequences, а поиск осуществлялся по банку Swiss-Prot. (Провести поиск по самой RefSeq у меня не получилось..)
Второй запрос действительно является более слабым, по сравнению с первым. Приближённое число ожидаемых случайных совпадений в 100 000 последовательностях (50 000 000 аминокислотных остатков) для первого запроса - 4*10-4, а для второго - 0,23. (Числа взяты из "Rich View" Prosite для каждого из запросов). В случае третьего запроса - это 5,9 случайных совпадений.
Также, основываясь на следующем задании, был проведён запрос, которым осуществлялся поиск белков, содержащие паттерны, найденные сервером Prosite в белке YojM_BACSU. К сожалению, запрос оказался абсолютно неинформативным, и в результате нашлись одни лишь явные негомологи; к тому же был достигнут порог в 5000 хитов.
Поиск всех мотивов PROSITE в последовательности белка YojM_BACSU
На сервере ExPASy через Prosite был проведён поиск имеющихся в их банке данных мотивов в белке YojM_BACSU. Результаты представлены в таблице 2.
| Таблица 2. Поиск мотивов в белке YojM_BACSU. | ||||||
|---|---|---|---|---|---|---|
| Идентификатор документа Prosite | Название мотива | Краткое описание мотива | Тип подписи | Паттерн | Специфична ли подпись? | Сколько мотивов нашлось в белке? |
| PS51257 | PROKAR_LIPOPROTEIN | Сайт присоединения липидов для прокариотических мембранных белков | Профиль | Специфична | 1 | |
| PS00008 | MYRISTYL | Сайт N-миристоилирования | Паттерн | G-{EDRKHPFYW}-x(2)-[STAGCN]-{P} | Неспецифична | 5 |
| PS00006 | CK2_PHOSPHO_SITE | Сайт фосфорилирования казеинкиназы II | Паттерн | [ST]-x(2)-[DE] | Неспецифична | 1 |
| PS00005 | PKC_PHOSPHO_SITE | Сайт фосфорилирования протеинкиназы С | Паттерн | [ST]-x-[RK] | Неспецифична | 2 |
| PS00004 | CAMP_PHOSPHO_SITE | Сайт фосфорилирования цАМФ- и цГМФ-зависимой протеинкиназы | Паттерн | [RK](2)-x-[ST] | Неспецифична | 1 |
Проверка гомологичности найденных белков
С помощью сервера UniProt были получены полные аминокислотные последовательности белков, найденных с помощью сильного паттерна из первого задания. Затем на сервере Европейского Биоинформатического Института программой "Muscle" проведено множественное выравнивание. Полученное выравнивание в программе "JalView" окрашено стандартной схемой ClustalX с процентом консервативности в 20%. Результат изображён на рисунке 1. Изображение без ограничения на консервативность можно посмотреть на рисунке 2 (на странице не представлено).
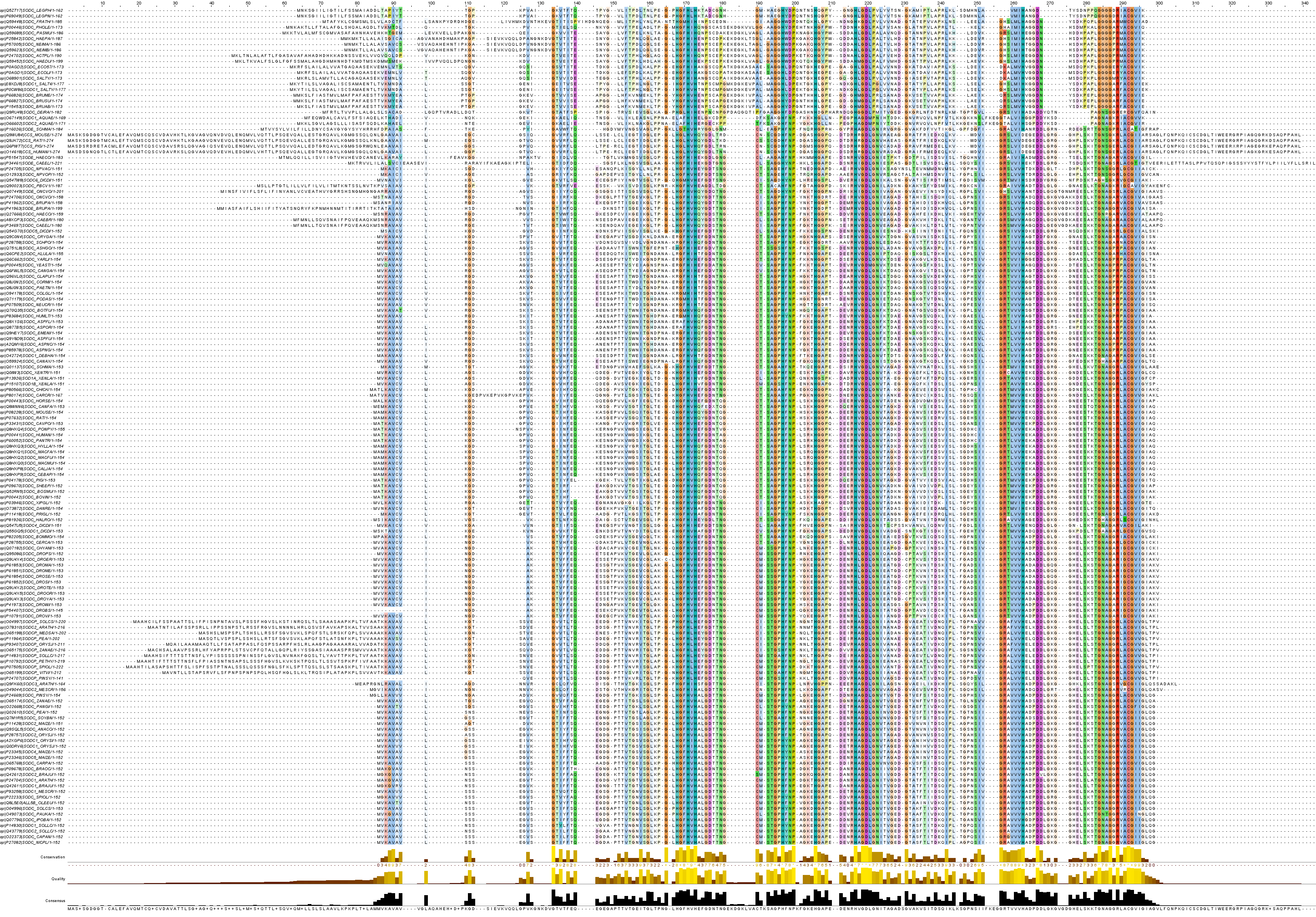 Рисунок 1. Множественное выравнивание найденных белков.
Рисунок 1. Множественное выравнивание найденных белков.Главная задача белка YojM_BACSU - связывать ионы меди и цинка. Как было выяснено в предыдущем практикуме, вторичная структура белка не столь консервативна, сколько аминокислоты, связывающие лиганды. А в полученном выравнивании наглядно прослеживаются консервативные почти по всей длине колонки. Значит, вполне оправдан вывод, что найденные белки действительно являются гомологами белка Yojm_BACU.
Увидеть результат поиска гомологов по сильному паттерну можно на рисунке 3 (на странице не представлен). Как видно, найденные последовательности вполне консервативны.