| FBB MSU site | Main page | About me | Terms |
Transmembrane proteins
OPM database
As transmembrane protein include alpha-helix structure I chose Bacterial cellulose synthase (PDB ID - 4p02).
Class: Alpha-helical polytopic
Family: Cellulose-conducting channel
Cellulose synthase - OPM page
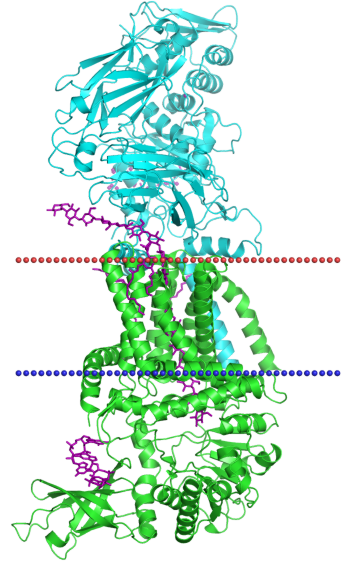
Characteristics of this protein you can see in table 1 and it's visualisation in figure 1.
Table 1. Characteristics of cellulose synthase, ID - 4p02.
| Hydrophobic Thickness | 29.8 ± 1.2 Å | |
| 2 transmembrane subunits | A - Segments: 1( 15- 34), 2( 41- 60), 3( 64- 86), 4( 96- 120), 5( 418- 435), 6( 447- 466), 7( 522- 543), 8( 549- 570) B - Segments: 1(695-716) | |
| Average number of amino acid residues in a transmembrane alpha-helix | 20 | |
| Localization | Bacterial Gram-negative inner membrane |
Figure 1. Visualisation of cellulose synthase, ID - 4p02.
As transmembrane protein include beta-barrel structure I chose Porin OmpF (PDB ID - 3pox).
Class: Beta-barrel transmembrane
Superfamily: Trimeric porins
Family: General Bacterial Porin (GBP)
Porin OmpF - OPM page
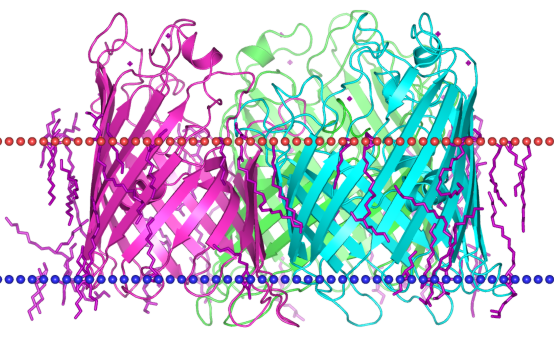
Characteristics of this protein you can see in table 2 and it's visualisation in figure 2.
Table 2. Characteristics of Porin OmpF, ID - 3pox.
| Hydrophobic Thickness | 24.0 ± 0.8 Å | |
| 3 transmembrane subunits (the same subunits) |
A - Segments: 1( 11- 23), 2( 37- 48), 3( 57- 64), 4( 79- 89), 5( 96- 101), 6( 136- 142), 7( 150- 159), 8( 173- 182), 9( 186- 192), 10( 213- 221), 11( 226- 232), 12( 256- 263), 13( 270- 276), 14( 294- 302), 15( 309- 315), 16( 331- 338) B - Segments: 1( 11- 23), 2( 37- 48), 3( 57- 64), 4( 79- 89), 5( 96- 101), 6( 136- 142), 7( 150- 159), 8( 173- 182), 9( 186- 192), 10( 213- 221), 11( 226- 232), 12( 256- 263), 13( 270- 276), 14( 294- 302), 15( 309- 315), 16( 331- 338) C - Segments: 1( 11- 23), 2( 37- 48), 3( 57- 64), 4( 79- 89), 5( 96- 101), 6( 136- 142), 7( 150- 159), 8( 173- 182), 9( 186- 192), 10( 213- 221), 11( 226- 232), 12( 257- 263), 13( 270- 276), 14( 294- 302), 15( 309- 315), 16( 332- 338) | |
| Average number of amino acid residues in a transmembrane beta-chain | 8 | |
| Localization | Bacterial Gram-negative outer membrane |
Figure 2. Visualisation of Porin OmpF, ID - 3pox.
Analise of predicted transmembrane helixes
As transmembrane protein include alpha-helix structure I chose Bacterial cellulose synthase (PDB ID - 4p02). In TMHMM and Phobius research I use A chain of this protein (sequence you can see below).
>4P02:A|PDBID|CHAIN|SEQUENCE MGTVRAKARSPLRVVPVLLFLLWVALLVPFGLLAAAPVAPSAQGLIALSAVVLVALLKPFADKMVPRFLLLSAASMLVMR YWFWRLFETLPPPALDASFLFALLLFAVETFSISIFFLNGFLSADPTDRPFPRPLQPEELPTVDILVPSYNEPADMLSVT LAAAKNMIYPARLRTVVLCDDGGTDQRCMSPDPELAQKAQERRRELQQLCRELGVVYSTRERNEHAKAGNMSAALERLKG ELVVVFDADHVPSRDFLARTVGYFVEDPDLFLVQTPHFFINPDPIQRNLALGDRCPPENEMFYGKIHRGLDRWGGAFFCG SAAVLRRRALDEAGGFAGETITEDAETALEIHSRGWKSLYIDRAMIAGLQPETFASFIQQRGRWATGMMQMLLLKNPLFR RGLGIAQRLCYLNSMSFWFFPLVRMMFLVAPLIYLFFGIEIFVATFEEVLAYMPGYLAVSFLVQNALFARQRWPLVSEVY EVAQAPYLARAIVTTLLRPRSARFAVTAKDETLSENYISPIYRPLLFTFLLCLSGVLATLVRWVAFPGDRSVLLVVGGWA VLNVLLVGFALRAVAEKQQRRAAPRVQMEVPAEAQIPAFGNRSLTATVLDASTSGVRLLVRLPGVGDPHPALEAGGLIQF QPKFPDAPQLERMVRGRIRSARREGGTVMVGVIFEAGQPIAVRETVAYLIFGESAHWRTMREATMRPIGLLHGMARILWM AAASLPKTARDFMDEPARRRRRHEEPKEKQAHLLAFGTDFSTEPDWAGELLDPTAQVSARPNTVAWGSNHHHHHHKLHHH HHH
Below you can see summary table (see table 3).
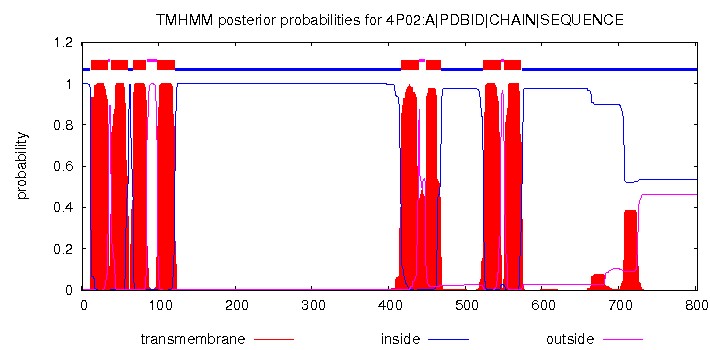
The result of TMHMM research you can see in figure 3.
Ox - protein sequence
Oy - probability of amino acid residue is located inside the membrane, in the external environment or in the cytoplasm
Red line - transmembrane part of protein
Blue line - cytoplasmic (inside) part of protein
Pink line - non cytoplasmic (outside) part of protein
Figure 3. Prediction of transmembrane structure of Cellulose synthase A chain made by TMHMM service.
# 4P02:A|PDBID|CHAIN|SEQUENCE Length: 803 # 4P02:A|PDBID|CHAIN|SEQUENCE Number of predicted TMHs: 8 # 4P02:A|PDBID|CHAIN|SEQUENCE Exp number of AAs in TMHs: 181.11815 # 4P02:A|PDBID|CHAIN|SEQUENCE Exp number, first 60 AAs: 42.73374 # 4P02:A|PDBID|CHAIN|SEQUENCE Total prob of N-in: 0.99963 # 4P02:A|PDBID|CHAIN|SEQUENCE POSSIBLE N-term signal sequence 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 1 11 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 12 34 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 35 37 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 38 60 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 61 66 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 67 84 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 85 98 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 99 121 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 122 416 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 417 439 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 440 448 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 449 468 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 469 523 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 524 546 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 547 550 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 551 573 4P02:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 574 803
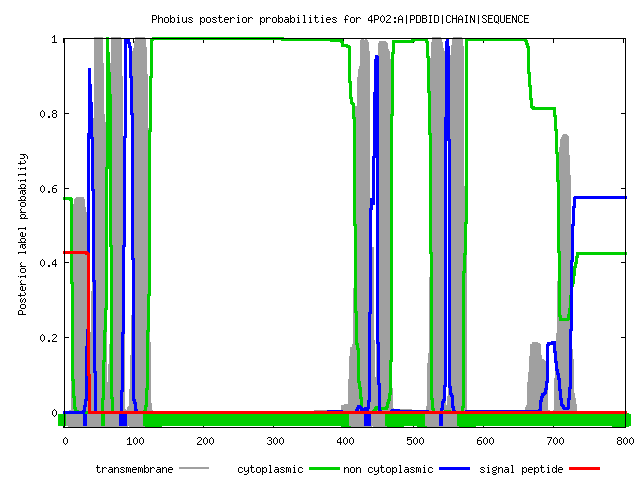
The result of Phobius research you can see in figure 4.
Ox - protein sequence
Oy - probability of amino acid residue is located inside the membrane, in the external environment or in the cytoplasm
Grey line - transmembrane part of protein
Green line - cytoplasmic (inside) part of protein
Blue line - non cytoplasmic (outside) part of protein
Red line - signal peptide
Figure 4. Prediction of transmembrane structure of Cellulose synthase A chain made by Phobius service.
|
ID 4P02:A|PDBID|CHAIN|SEQUENCE FT TOPO_DOM 1 11 CYTOPLASMIC. FT TRANSMEM 12 36 FT TOPO_DOM 37 41 NON CYTOPLASMIC. FT TRANSMEM 42 61 FT TOPO_DOM 62 67 CYTOPLASMIC. FT TRANSMEM 68 87 FT TOPO_DOM 88 98 NON CYTOPLASMIC. FT TRANSMEM 99 122 FT TOPO_DOM 123 416 CYTOPLASMIC. FT TRANSMEM 417 437 FT TOPO_DOM 438 448 NON CYTOPLASMIC. FT TRANSMEM 449 468 FT TOPO_DOM 469 524 CYTOPLASMIC. FT TRANSMEM 525 545 FT TOPO_DOM 546 550 NON CYTOPLASMIC. FT TRANSMEM 551 571 FT TOPO_DOM 572 803 CYTOPLASMIC. |
Table 3. Transmembrane alpha-helixes predicted by TMHMM and Phobius and ones presented in OPM database.
| OPM | 1( 15- 34), 2( 41- 60), 3( 64- 86), 4( 96- 120), 5( 418- 435), 6( 447- 466), 7( 522- 543), 8( 549- 570) |
| TMHMM | 1(12 - 34), 2(38 - 60), 3(67 - 84), 4(99 - 121), 5(417 - 439), 6(449 - 468), 7(524 - 546), 8(551 - 573) |
| Phobius | 1(12 - 36), 2(42 - 61), 3(68 - 87), 4(99 - 122), 5(417 - 437), 6(449 - 468), 7(525 - 545), 8(551 - 571) |
Summary:
The number of predicted helixes is the same as presented in OPM. Coordinates of the predicted helixes are very close to the real ones (may vary by 1-3 residues). So I think in this case predicted transmembrane structure are reliable.
TCDB database
TCID:
- Position 1 - class of transporter protein
- Position 2 - subclass
- Position 3 - family
- Position 4 - subfamily
- Position 5 - the protein
Chosen proteins were found in TCDB.
-
Cellulose synthase, ID - 4p02
TCID - 4.D.3.1.5
- 4 - Group translocators (Белки - транспортеры)
- D - Polysaccharide Synthase/Exporters (Белки, синтезирующие и секретирующие полисахариды)
- 3 - The Glycan Glucosyl Transferase (OpgH) Family (Семейство гликан глюкозил трансфераз)
- 1.5 - Putative cellulose synthase (Целлюлозо-синтаза)
- Substrate - Cellulose
Cellulose synthase and transporter, BcsA, which functions with BcsB (periplasmic protein with an N-terminal TMS) and BcsC (an 18 beta-stranded outer membrane porin). The x-ray structure of the BcsA-B complex has been determined at 3.5 Å resolution (Morgan et al. 2013). Cellulose is synthesized and secreted by the membrane-integrated cellulose synthase. Substrate- and product-bound structures of BcsA provided the basis for substrate recognition and demonstrated the stepwise elongation of cellulose. Structural snapshots showed that BcsA translocates cellulose via a ratcheting mechanism involving a 'finger helix' that contacts the polymer's terminal glucose. Cooperating with BcsA's gating loop, the finger helix moves 'up' and 'down' in response to substrate binding and polymer elongation, respectively, thereby pushing the elongated polymer into BcsA's transmembrane channel. This mechanism was validated by tethering BcsA's finger helix, which inhibits polymer translocation but not elongation (Morgan et al. 2016). [*]
PDB structures: 4HG6, 4P00, 4P02
-
Porin OmpF, ID - 3pox
TCID - 1.B.1.1.1
- 1 - Channels and pores (Каналы и поры)
- B - beta-Barrel porins and other outer membrane proteins (Порины в виде бета-боченков и другие мембранные белки)
- 1 - General bacterial porin family (Семейство, объединяющее порины бактерий)
- 1.1 - OMPF aka TOLF aka CMLB aka COA aka CRY aka B0929
- Substrate - Ceftriaxone, Cefpirome, ions, peptides, Colicin E9, Protamine, Ceftazidime
OmpF general porin. OmpF can deliver peptides of >6 KDa (epitopes) including protamine, through the pore lumen from the periplasm to the outside (Housden et al., 2010; Ghale et al. 2014). For cephalosporin antibiotics, the interaction strength series is ceftriaxone > cefpirome > ceftazidime (Lovelle et al. 2011). An unfolded protein such as colicin E9 can thread through OmpF from the outside to reach the periplasm (Housden et al. 2013). Polynucleotides can pass through OmpF (Hadi-Alijanvand and Rouhani 2015). LPS influences the movement of bulk ions (K+ and Cl-), but the ion selectivity of OmpF is mainly affected by bulk ion concentrations (Patel et al. 2016). OMPs such as OmpF cluster into islands that restrict their lateral mobility, while IMPs generally diffuse throughout the cell. Rassam et al. 2018 demonstrated that when transient, energy-dependent transmembrane connections are formed, IMPs become subjugated by the inherent organisation of OMPs, and that such connections impact IMP function. They showed that while establishing a translocon for import, colicin ColE9 sequesters the IMPs of the proton motive force (PMF)-linked Tol-Pal complex into islands mirroring those of colicin-bound OMPs. Through this imposed organisation, the bacteriocin subverts the outer-membrane stabilizing role of Tol-Pal, blocking its recruitment to cell division sites and slowing membrane constriction. The ordering of IMPs by OMPs via an energised inter-membrane bridge represents an emerging functional paradigm in cell envelope biology (Rassam et al. 2018). [**]
PDB structures: more then 10 (too much for my work)
Translation into Russian:
[*] Целлюлозо-синтаза и транспортер, BcsA, функционирующий вместе с BcsB (периплазматический белок с N-концевой TMS) и BcsC (порин из 18 бета-листов, встроенный в наружную мембрану). Рентгеновская структура комплекса BcsA-B определена с разрешением 3,5 Å (Morgan et al. 2013). Целлюлоза синтезируется и секретируется на поверхность встроенным в мембрану комплексом целлюлозо-синтазы. Субстрат- и продукт-связанные структуры BcsA позволили опознать субстрат и пронаблюдать ступенчатый синтез целлюлозы. Структурные снимки показали, что BcsA перемещает целлюлозу с помощью храпового механизма, используя 'finger helix' (структура ‘палец’ из альфа-спиралей), который взаимодействует с терминальной глюкозой в полимере. В комплексе с петлей BcsA 'finger helix' двигает полимер "вверх" и "вниз" в ответ на его связывание и удлинение, соответственно, таким образом протягивая целлюлозу через трансмембранный канал BcsA. Этот механизм подтверждается тем, что, если блокировать 'finger helix' BcsA, транслокация полимера прекращается, но он продолжает удлиняться (Morgan et al. 2016).
[**] OmpF типичный белок порин. OmpF может доставлять пептиды >6 KDa (эпитопы), в том числе протамин, через просвет поры из периплазматического пространства наружу (Housden et al., 2010; Ghale et al. 2014). Для цефалоспориновых антибиотиков прочность связывания падает в ряду ceftriaxone > cefpirome > ceftazidime (Lovelle et al. 2011). Белки в нативной конформации, например колицин E9, могут проникать в клетку извне через порины (Housden et al. 2013). Полинуклеотиды также могут проходить через OmpF (Hadi-Alijanvand и Rouhani 2015). Липополисахариды (LPS) влияют на движение объемных ионов (K+ и Cl -), но селективность прохода ионов через OmpF в основном зависит от их концентрации (Patel et al. 2016). Комплексы OMP, такие как OmpF, объединяются в островки, с ограниченной боковой подвижностью, в то время как комплексы вроде IMP обычно распределены по всей клетке. Рассам с коллегами в 2018 продемонстрировал, что при формировании переходных, энергозависимых трансмембранных соединений, IMP подчиняются внутренней организации со стороны OMP, более того OMP влияют на функцию IMP. Было показано, что при создании транслокона для импорта колицин ColE9 изолирует IMP от связанного с протонной движущей силой (PMF) комплекса Tol-Pal, путем образования OMP-колицин связанных комплексов в OMP-островках. В результате бактериоцин берет на себя роль наружной мембраны, стабилизируя Tol-Pal, блокируя его захват в сайтах деления клетки и замедляя сужение мембраны. Упорядочение IMP с помощью OMP путем активации внутренне-мембранного моста представляет собой формирующуюся функциональную парадигму в биологии клеточной оболочки (Rassam et al. 2018).
| Term 4 |
| ← Pr11 | Term 5 → |
© Darya Potanina, 2018