Главная
Практикум №12: "Эволюционные домены"
Задание 1. Построение выравнивания представителей домена Pfam белков с разной доменной архитектурой
Для выполнения данного задания был выбран домен ETF-Q-оксидоредуктазы. ID: ETF_QO, AC: PF05187.
Функция домена: принимает электроны от флавопротеина и восстанавливает убихинон во внутренней мембране митохондрий, участвуя в ЭТЦ.
Данный домен присутствует в 22 архитектурах (ссылка).
Проект JalView содержит выравнивание всех последовательностей, содержащих данный домен. Оно же в фоормате Fasta.
Были выбраны 2 доменные архитектуры:
NAD_binding_8, ETF_QO (554 последовательности). Помимо домена ETF_QO, так же присутствует NAD-связывающий домен. В дальнейшем эта
архитектура будет обозначаться так: 2_
 Рис. 1. Архитектура NAD_binding_8, ETF_QO (NAD_binding_8 показан зелёным цветом,
ETF_QO красным).
ETF_QO (199 последовательностей). Здесь только один домен ETF_QO.
Рис. 1. Архитектура NAD_binding_8, ETF_QO (NAD_binding_8 показан зелёным цветом,
ETF_QO красным).
ETF_QO (199 последовательностей). Здесь только один домен ETF_QO.
 Рис. 2. Архитектура ETF_QO (домен показан красным).
Рис. 2. Архитектура ETF_QO (домен показан красным).
Затем нужно было выбрать таксон и подтаксоны. Таксон: cellular organisms, подтаксоны - Eukaryota и Bacteria. Последовательности, принадлежащие этим подтаксонам далее в выравнивани Jalview будут отмечаны, префиксами E_ и P_.
Файл EXcel содержит сводную таблицу с информацией о доменной архитектуре и таксономии ("Сводная таблица").
Затем нужно было выбрать около 20 представителей каждого подтаксона с каждой доменной архитектурой. Результат: лист "tax" в EXcel
(число 360 отражает доменную архитектуру 2, а 150 - 1 (как это получилось, можно понять из сводной таблицы) и проект JalView, содержащий выравнивания 4 групп последовательностей исходного домена (группы по подтаксонам и доменным архитектурам). В выравниваниях - раскраска ClustalX с порогом консервативности 20 % во всех группах. Предварительно из выравнивания были удалены пара последовательностей, которые были выровнены сильно хуже других. В остальном выравнивание
можно считать правильным, так как в нем нет разделений на фрагменты. Стоит заметить, что большая часть последовательностей 1_B визуально
отличается от последовательностей остальных групп, так как у них
отсутствует небольшая начальная часть данного домена. В других группах этого не наблюдается.
Построение дерева
В программе MEGA было построено филогенетическое дерево последовательностей методом максимального правдоподобия (Рис. 3).
Скобочная формула
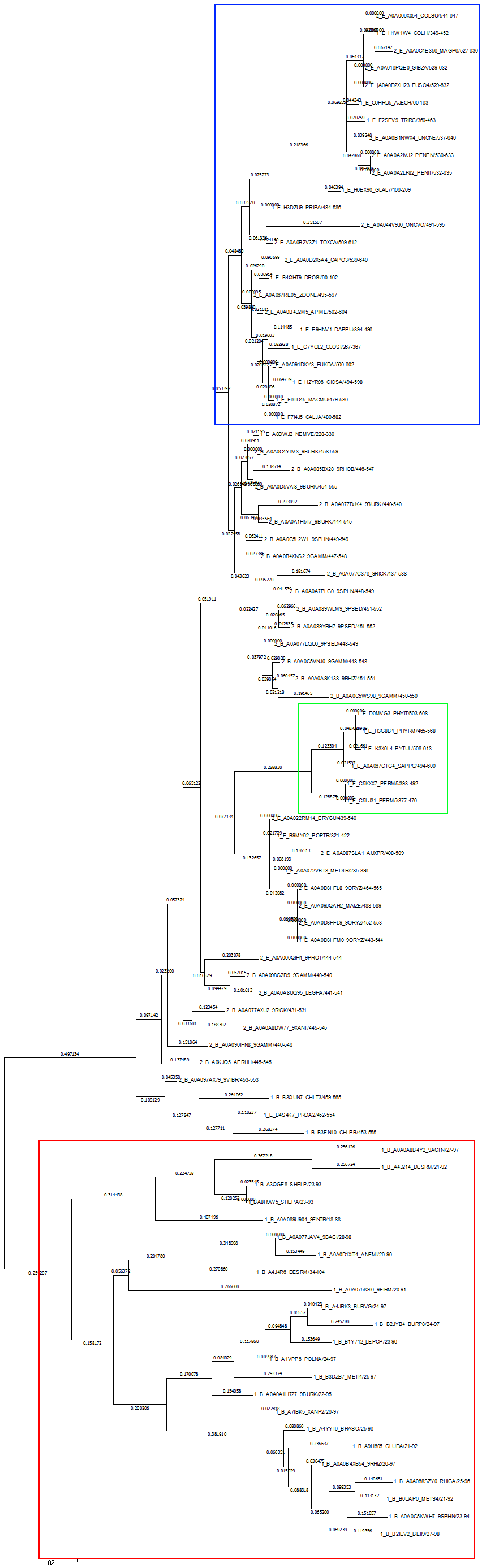 Рис. 3. Филогенетическое дерево данных последовательностей
Рис. 3. Филогенетическое дерево данных последовательностей
Видно, что дерево не разбилось на клады. Стоит лишь отметить, что выделилась клада, состоящая практически полностью из
группы 1_B_ (красная рамка). Также видно, что эукариоты хорошо отличимы от бактерий, то есть по таксономии выделение в отдельную группу произошло (синяя рамка),
но, так как обе архитектуры довольны схожи, группы 1_E_ и 2_E_ расположены на дереве в целом "вперемешку" за исключением ветви (зёлёная рамка),
которая отчётливо отделяет 6 последовательностей 1_E_ от других белков.
Построение профиля подсемейства
Был построен HMM-профиль для единственной выделившейся группы из четырёх - 1_B: ссылка
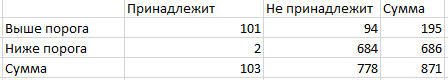
На основе результатов поиска был получен файл EXCEl, содержащий правильные находки .Была построена ROC-кривая (Рис. 4), по которой
возможно определить поровое значение E-value (1,8E-23)
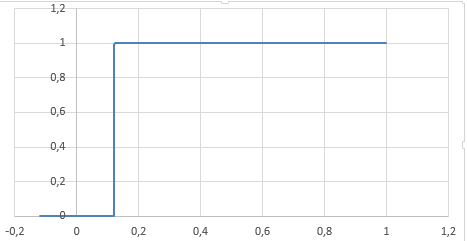 Рис. 4. ROC-кривая.
Рис. 4. ROC-кривая.
Таблица 1