Программа BLAST
Гомологи белка 2-hyoxy-6-oxo-6-phenylhexa-2,4-dienoate hyolase
| Таблица 1. Параметры поиска при запуске BLAST | |||||||
| Параметры поиска | Значение | ||||||
| Database | UniProtKB/Swiss-Prot(swissprot) | ||||||
| Organism | нет ограничений | ||||||
| Algorithm | blastp (protein-protein BLAST) | ||||||
| Max target sequences | 50 | ||||||
| Expect threshold | 10 | ||||||
| Word size | 6 | ||||||
| Matrix | BLOSUM62 | ||||||
| Gap Costs | Existence 11 Extension 1 | ||||||
Текстовый документ с результатами поиска:
здесь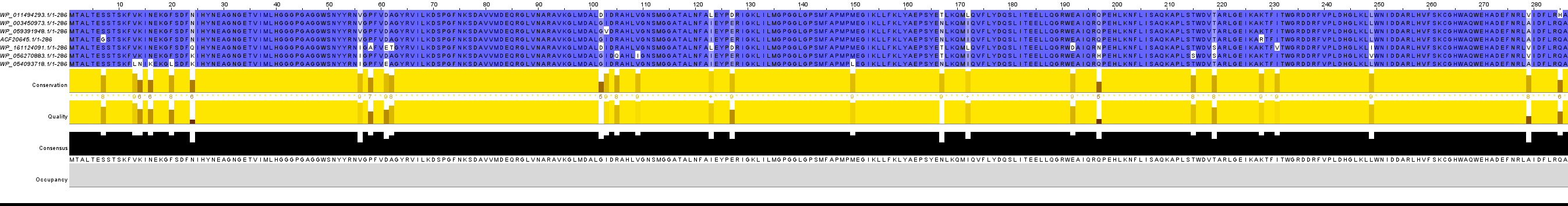
Для выравнивания были выбраны следующие белки: 2-hydroxy-6-oxo-6-phenylhexa-2,4-dienoate hydrolase [Burkholderiaceae], 2-hydroxy-6-oxo-6-phenylhexa-2,4-dienoate hydrolase [Pseudomonas], 2-hydroxy-6-oxo-6-phenylhexa-2,4-dienoate hydrolase [Pseudomonas toyotomiensis], 2-hydroxy-6-oxo-6-phenylhexa-2,4-dienoic acid hydrolase [Achromobacter sp. BP3], 2-hydroxy-6-oxo-6-phenylhexa-2,4-dienoate hydrolase [Malikia spinosa], 2-hydroxy-6-oxo-6-phenylhexa-2,4-dienoate hydrolase [Hydrogenophaga], 2-hydroxy-6-oxo-6-phenylhexa-2,4-dienoate hydrolase [Pseudomonas stutzeri].
Все организмы являются грамм-отрицательными, аэробными, почвенными бактериями, использующие в своем метаболизме углеводороды. 2-hydroxy-6-oxo-6-phenylhexa-2,4-dienoate hydrolase гидролтзует С-С связи, обеспечивая метаболизм бензольных соединений. Это все объясняет гомологичность последовательностей исследуемых белков. Выравнивания имеют большой процент схожести. Во всех последовательностях совпадают участки с 24-56, 63-101, 128-149, 151-166, 250-278 и еще некоторые участки. Моя бактерия Burkholderia xenovorans LB400, Achromobacter sp., Malikia spinosa, Hydrogenophaga входят в порядок Burkholderiales, который родственнен порядку Pseudomonadales - подтвержается при сравнении выравнивааний. При этом ближе всего к последовательностям бактерий рода Pseudomonas последовательность Achromobacter sp. BP3, а на мой белок из прктикума 8 больше всего похожа последовательноть Malikia spinosa.Ссылка на проект Jalview:
Protein alignmentГомологи вирусного белка
| Protein Name | ID | AC | Название вируса |
|---|---|---|---|
| mRNA-capping enzyme nsP1 | POLN_SINDV | P03317 | Sindbis virus (SINV) |
Для дальнейших действий был выбран зрелый белок — mRNA-capping enzyme nsP1, с координатами: 1..540
Ссылка на последовательность в формате fasta:
здесь| Параметры поиска | Значение |
| Database | UniProtKB/Swiss-Prot(swissprot) |
| Organism | нет ограничений |
| Algorithm | blastp (protein-protein BLAST) |
| Max target sequences | 100 |
| Expect threshold | 10 |
| Word size | 3 |
| Matrix | BLOSUM62 |
| Gap Costs | Existence 11 Extension 1 |
Текстовый документ с результатами поиска:
здесьДля выравнивания были выбран белок Polyprotein P1234 следующих вирусов: Sindbis virus, Ockelbo virus, Aura virus, Getah virus, Sagiyama virus
Последовательности данных белков хорошо покрасились по идентичности на участках, например, участки 32-49, 261-300,358-371.Выравнивания были обрезаны по номерам а.к. на которые выровнялись крайние буквы исходного белка. Идентичность выше 20%, поэтому данные белки можно считать гомологичными.
Ссылка на проект Jalview:
Protein alignmentИсследование зависимости E-value от объёма банка
Повторив поиск из предыдущего пункта с ограничением по вирусами (Viruses), мы увидим, что список находок уменьшился со 90 до 87, также изменились значения E-value — увеличились примерно на 1-2 порядка. Значение E-value зависит от размера базы данных линейно, поэтому мы можем примерно оценить долю вирусных белков в Swissprot: по находке белка Q8JJX1 [Salmon pancreas disease virus] 3е-132/8e-131=0,0375
Примерная доля вирусных белков в базе Swiss-prot составляет 0,0375 (3,75%).
Текстовый документ с результатами поиска:
здесь