Занятие 11: работа с программой RasMol
Работа в командном окне и разные типы моделей структуры
Модели 3D структур белков бывают шариковые, проволочные, шарнирные и ленточные. Например, шариковая выглядит следующим образом: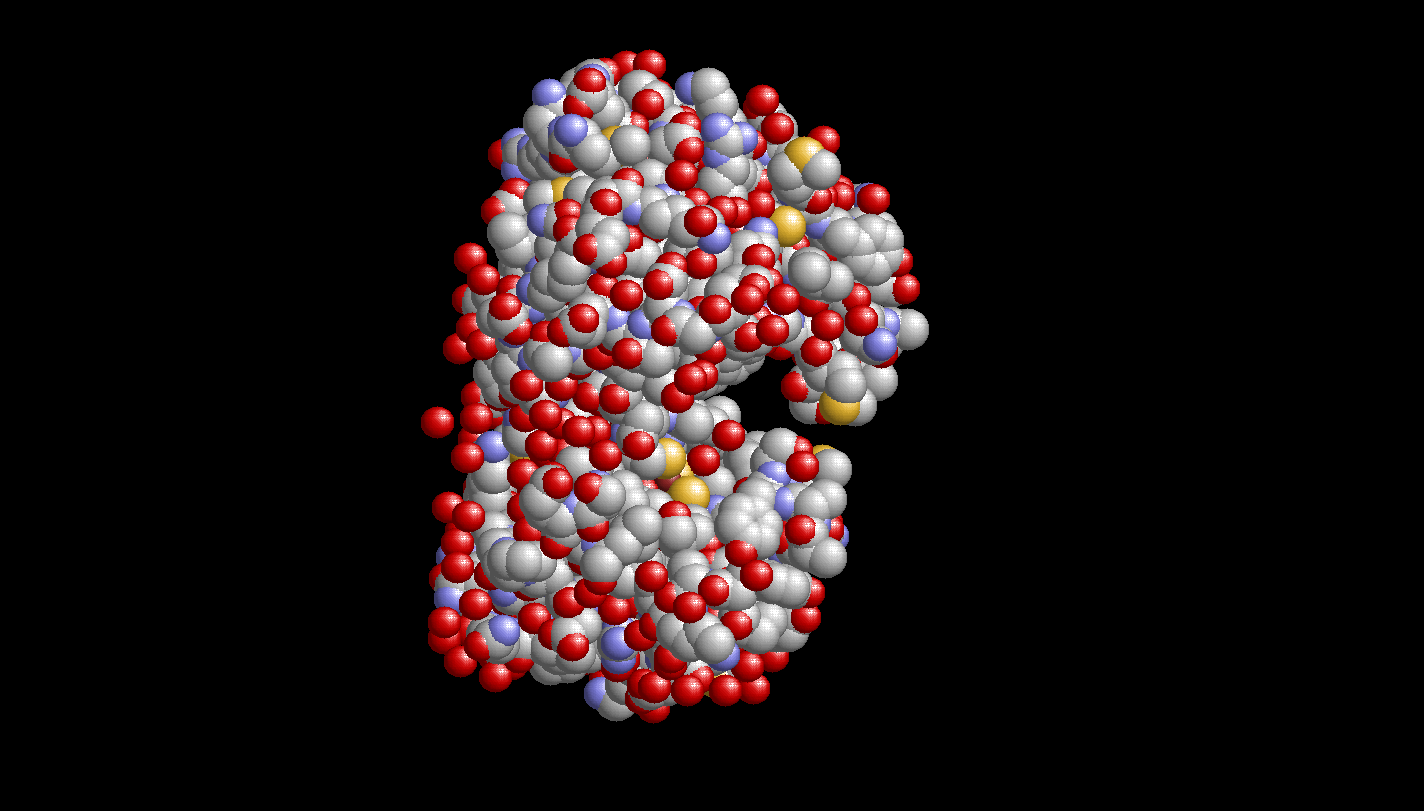
Изображение шариковой модели структуры
Определение молекул в заданной структуре
Пространственная структура CDD_BACSU (PDB ID 1UWZ)
Заголовок структуры: HYDROLASEНазвание структуры: BACILLUS SUBTILIS CYTIDINE DEAMINASE WITH AN ARG56 – ALA SUBSTITUTION (Цитидиндезаминаза сенной палочки с заменой ARG56 – ALA.)
В документе представлены следующие цепи макромолекул:
| Идентификатор цепи | Число остатков | Название молекулы |
| A | 130 | CYTIDINE DEAMINASE |
| B | 130 | CYTIDINE DEAMINASE |
В документе представлены следующие низкомолекулярные вещества:
| ID | Название | Формула | Число молекул | Русское название |
| ZN | ZINC ION | ZN 2+ | 2 | Ион цинка |
| THU | TETRAHYDRODEOXYURIDINE | C9 H14 N2 O5 | 2 | Тетрагидродезоксиуридин |
| HOH | - | H2 O1 | 148 | Вода |
Мы видим, что есть расхождения того, что можно видеть посредством RasMol, с тем, что описано в поле SEQRES pdb-файла. В каждой цепочке «не хватает» по 6 остатков. В поле REMARK находим интересную информацию:
MISSING RESIDUES
THE FOLLOWING RESIDUES WERE NOT LOCATED IN THE EXPERIMENT.
(M=MODEL NUMBER; RES=RESIDUE NAME; C=CHAIN IDENTIFIER; SSSEQ=SEQUENCE NUMBER; I=INSERTION CODE.)
M RES C SSSEQI
HIS A 131
ASP A 132
GLU A 133
ARG A 134
LYS A 135
LEU A 136
HIS B 131
ASP B 132
GLU B 133
ARG B 134
LYS B 135
LEU B 136
Т.е. при проведении эксперимента указанные остатки отсутствовали (причин этому может быть великое множество), и мы увидим только 130 аминокислотных остатков в каждой цепи данного белка.
Создание скрипта
С помощью программы RasMol был создан скрипт, последовательно генерирующий заданные изображения структуры белка. Скрипт создаёт следующие картинки.
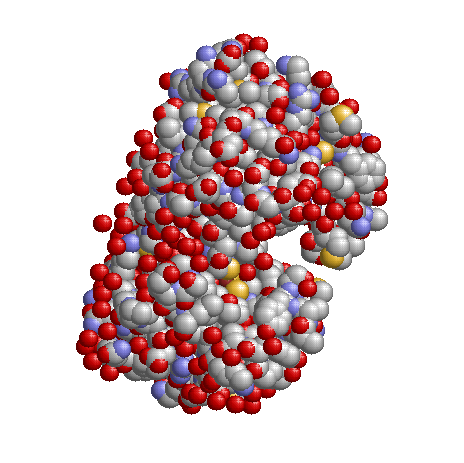
Вся структура в шариковой модели

Структура только белка в остовной модели
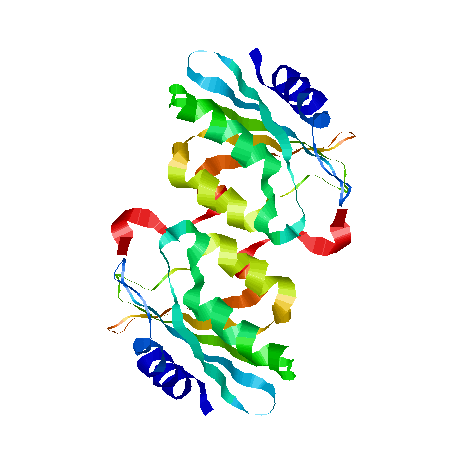
Структура только белка в ленточной модели
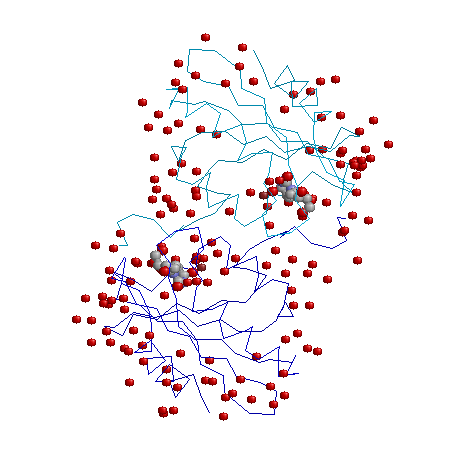
Изображение всех компонентов структуры
Изображение всех компонентов структуры в оригинальном размере.
- Белок представлен в остовной модели, разным цветом отмечены разные полипептидные цепи.
- Лиганды из 2-х и более атомов - в шарнирной модели с раскраской по атомам.
- Молекулы воды и одноатомные лиганды - в шариковой модели (красный цвет).
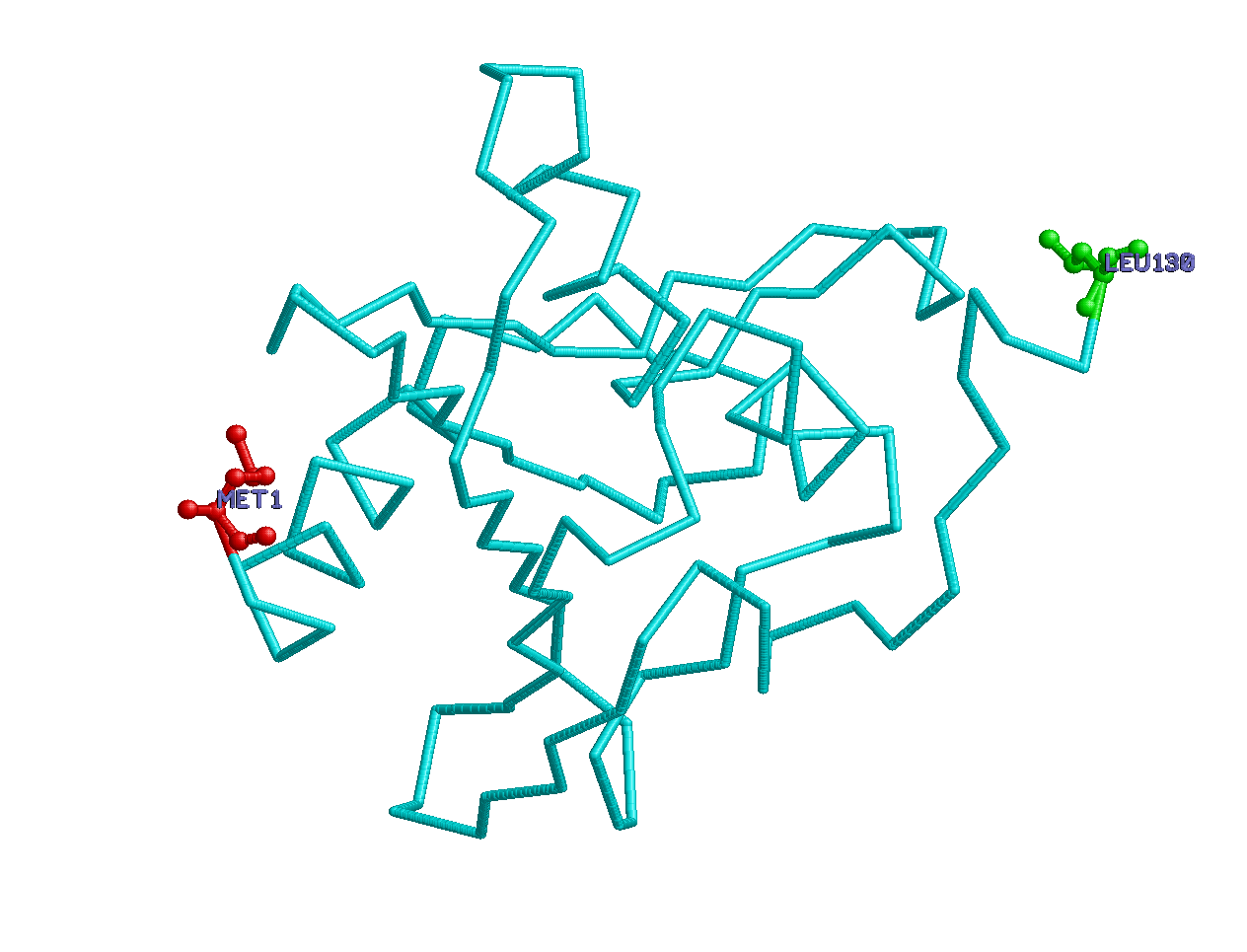
Изображение концевых остатков у одной полипептидной цепи
N- и С-концевые аминокислотные остатки в шарнирной модели, подписаны их названия и номера.
Концевые остатки, шарнирная модель
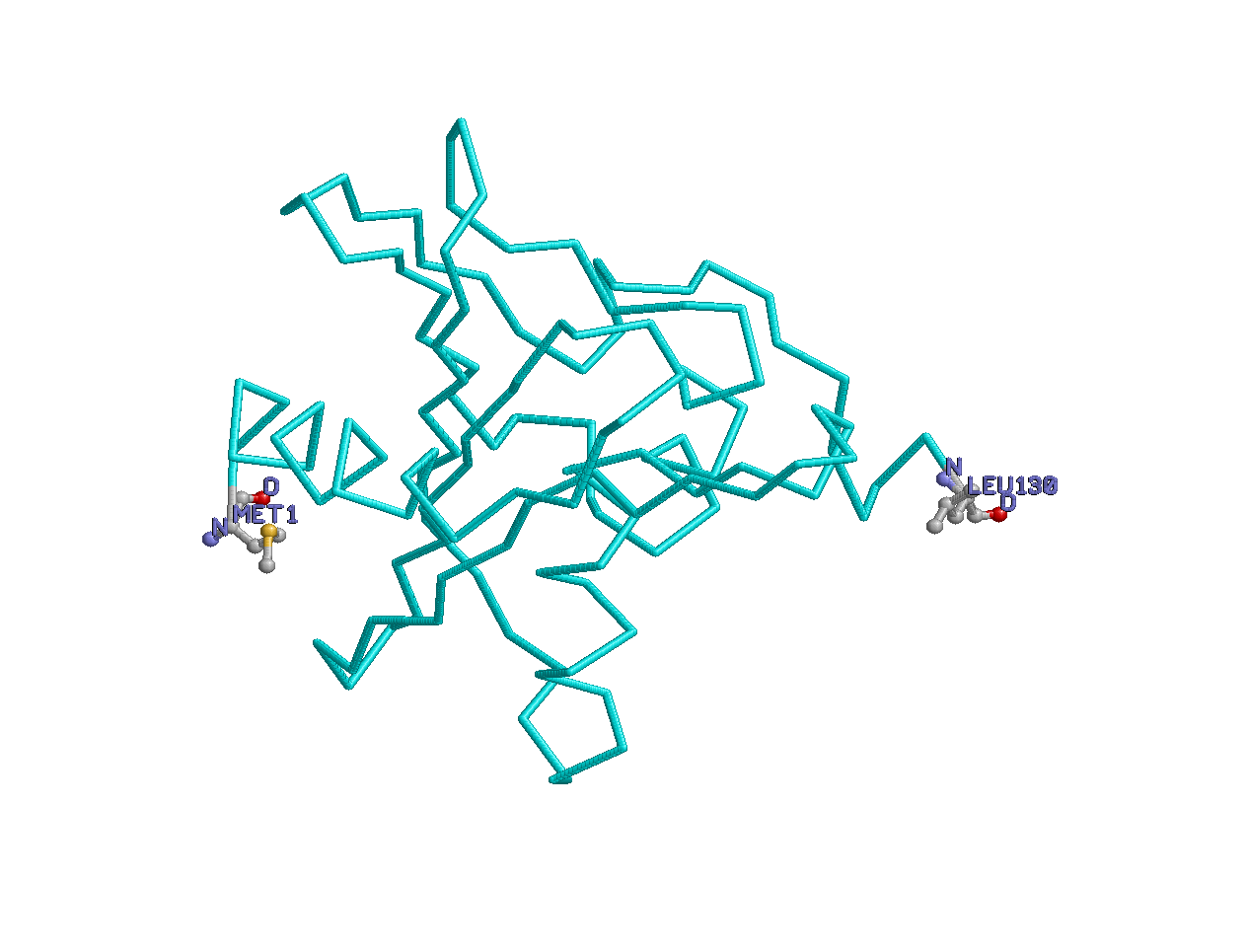
Изображение концевых остатков в оригинальном размере.
Концевые остатки, шарнирная модель
Изображение концевых остатков в оригинальном размере.
Общая форма заданного белка и его размеры
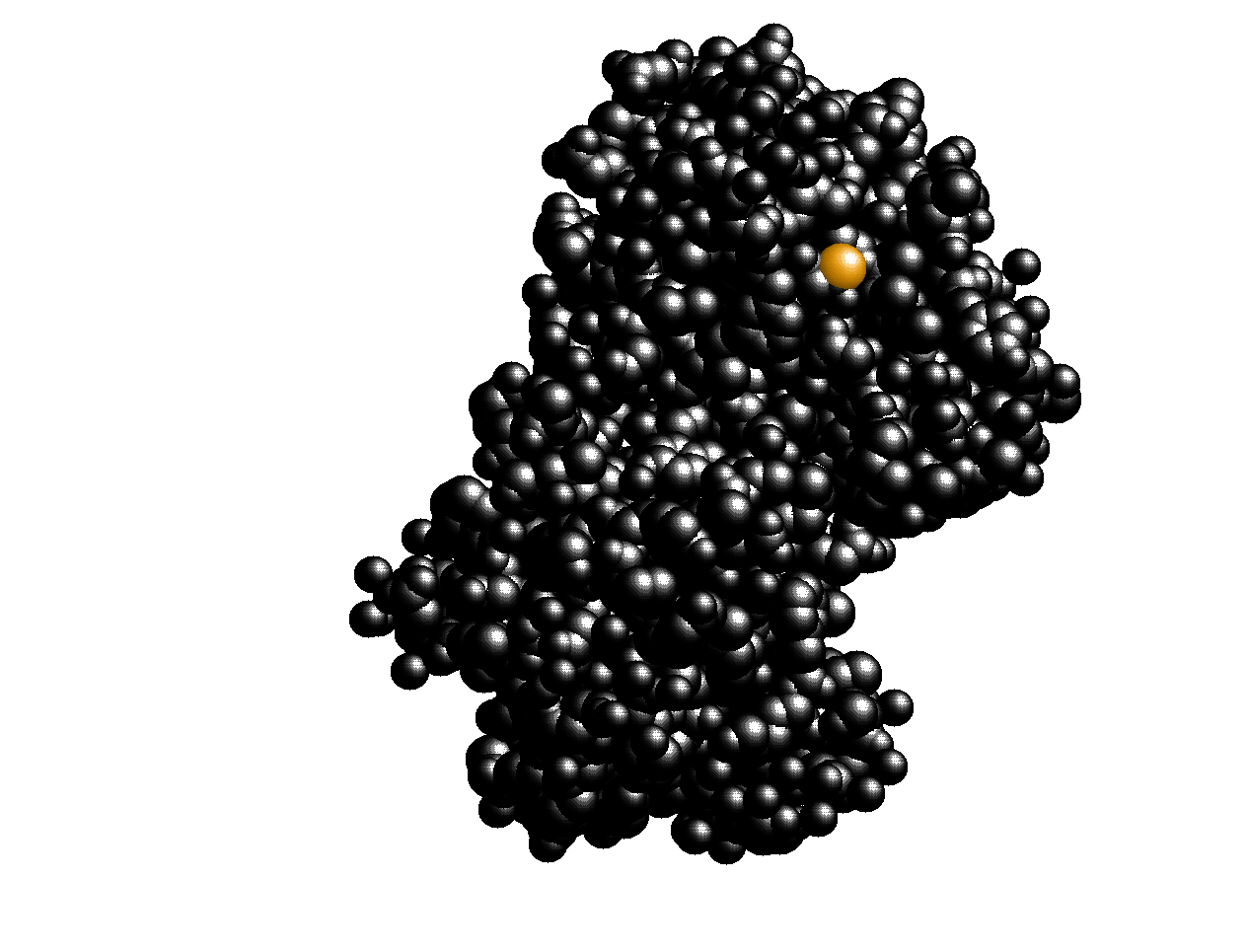
Белковая часть структуры в шариковой модели
Структура, представленная на картинке, на мой взгляд, походит на игрушку-динозаврика ("глаз" выделен для наглядности).
В программе RasMol были измерены размеры структуры. Вот какие данные мы получили:
Distance GlU5B.OE 2- GLU5A.OE2: 61.07 A
Distance LYS23A.CE – PRO80B.CG: 44.35 A
Distance LYS23B.CE – GLU128B.CG: 41.77 A
Аппроксимируем белок подходящей геометрической фигурой, например цилиндром (h≈61Â, r≈43Â).
Объём цилиндра V = π*(43*10-4)2*61*10-4 ≈ 3.54337094 * 10-7 (мкм3).
Сколько молекул Вашего белка поместится в одной клетке Escherichia coli, если аппроксимировать клетку цилиндром r=0.5µm и h=2µm?
Если радиус цилиндра 5*10-7 метров, высота – 2*10-6 метров, то объём такого цилиндра равен V = π*0.52*2 ≈ 1.57079633 (мкм3). Чтобы узнать примерное количество молекул, разделим найденный нами объём цилиндра (≈ объём клетки) на объём, найденный нами на первом шаге. Мы получили число 4 433 056.
Предположим, что все 4400 белков кишечной палочки были синтезированы одновременно и в равных количествах. Предположим, что размеры всех белков одинаковы и равны размерам Вашего белка. Предположим, что в клетке ничего нет, кроме белков. Сколькими молекулами будет представлен каждый белок?
Мы уже вычислили примерное количество молекул белка в одной клетке Escherichia coli.
Чтобы вычислить, сколькими молекулами представлен каждый белок, разделим это число на число белков:
4 433 056 / 4400 ≈ 1 007.
Предел разрешения светового микроскопа оценивается как λ/2, где λ - длина волны. Сколько молекул белка потребуется, чтобы создать объект, видимый под световым микроскопом?
Длина волны света (380-720 нм) – в среднем, 570 нм. Предел разрешения светового микроскопа вычисляется как λ/2 и равен 570/2=285нм. Можно рассматривать цилиндр как со стороны основания, так и со стороны боковой поверхности.
Со стороны основания.
Минимальная видимая в микроскоп площадь будет равна π*5702 ≈ 1 020 703(нм2). Площадь же основания цилиндра, в который мы вписывали молекулу белка - π*4.32 ≈ 58(нм2). Пренебрегая небольшой погрешностью, поделим эти числа и найдём примерное число молекул белка, которые можно увидеть в микроскоп:
1 020 703 / 58 ≈ 17 598 (молекул).Со стороны боковой поверхности.
Минимальная видимая в микроскоп площадь будет равна 5702 ≈ 324 900(нм2). Площадь видимой части (проекции на плоскость) цилиндра можно рассчитать как d*h=2*r*h=2*4.3*6.1 ≈ 52(нм2). Приближенно вычислим искомое число молекул:
324 900 / 52 ≈ 6 248 (молекул).
Сравнение изображений аминокислот и аминокислотных остатков
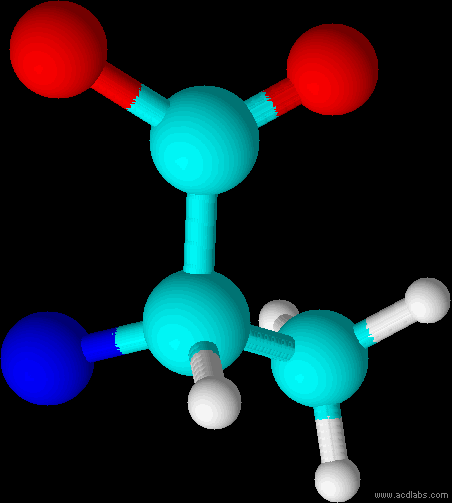
Аланин в ChemSketch
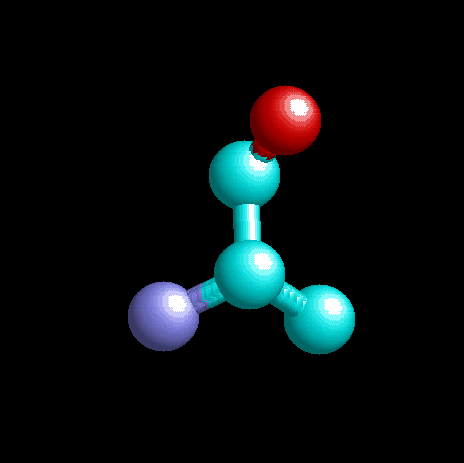
Аланин в RasMol
Ранее с помощью программы ACD ChemSketch было получено изображение аланина. С помощью RasMol можно получить изображение остатка этой аминокислоты в реальном белке, используя команды:
restrict 13:A
centre selected
zoom 700
spacefill 100
wireframe 50
Мы видим, что в полученной в программе RasMol структуре отсутствует атом кислорода. Он потребовался для образования воды при образовании пептидной связи. (Действительно, ведь в ACD ChemSketch мы «рисовали» саму аминокислоту, а не остаток.) Также мы видим, что во втором изображении отсутствуют атомы водорода, вероятно, слишком малые для распознавания при анализе структуры белка.
Ссылки
- Структура белка в формате .pdb.
- Скрипт, последовательно генерирующий заданные изображения структуры, в формате .spt.
- Простой скрипт, приведённый в подсказках, в формате.spt.
