Для построения профиля был выбран домен RF1 белка из шестого практикума для которого на указанной странице строился паттерн семейства на сайте Prosite. Соответствующий ему идентификатор PFAM: PF00472, далее приведена ссылка на страницу семейства в EMBL-EBI - Family: RF-1 (PF00472)
Данный домен был обнаружен у факторов высвобождения пептидной цепи, если не считать значительно меньшее количество белков с неизвестной функцией. Домен отвечает в белке за пептидил-тРНК гидролазную активность. Область RF1 содержит высококонсервативный мотив GGQ, в которой глутамин, судя по всему, координирует воду, которая опосредует гидролиз [1].
Выборка последовательностей была определена для таксона Proteobacteria. Запрос в базу данных Uniprot представленн ниже:
database:(type:pfam id:PF00471) taxonomy:proteobacteria AND reviewed:yes |
muscle -in pr8_out.fasta -out mus_align.fasta |
Данные задачи были выполнены через SSH-клиент на сервере kodomo следующими командами:
hmm2build profile.out mus_align.fasta hmm2calibrate profile.out |
Ради проверки коректности задание так же было выполнено с помощью пакета HMMER 3.0:
hmmbuild try_profile.out pr8_try.stk |
Команда ниже выполнила поиск среди записей банка данных SwissProt.
hmm2search --domE 1000 --domT -50 profile.out /srv/databases/emboss/data/uniprot/uniprot_sprot.fasta > swp_find.out |
Данные выдачи были записаны в отдельный, второй лист "Поиск SW" книги Excel. Последовательности, ранее обнаруженные в Uniprot, были отмечены в колонке "Is in ident-s?" знаком '+'.
Было определено несколько потенциальных порогов и для каждого рассчитаны значения:
Sensуtivity(True Positive/ (True Positive + False negative)
Specificity(True Negative/(False Positive + True Negative),
Precision(True Positive/ (True Positive + False positive))
Параллельно по имеющимся данным средствами Excel были построены гистограмма распредления скоров по убыванию и ROS-кривая:
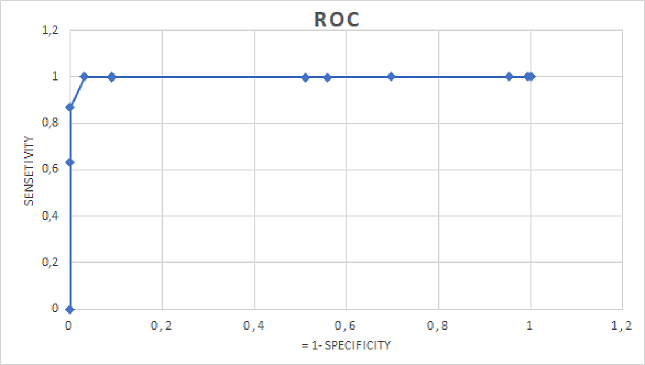 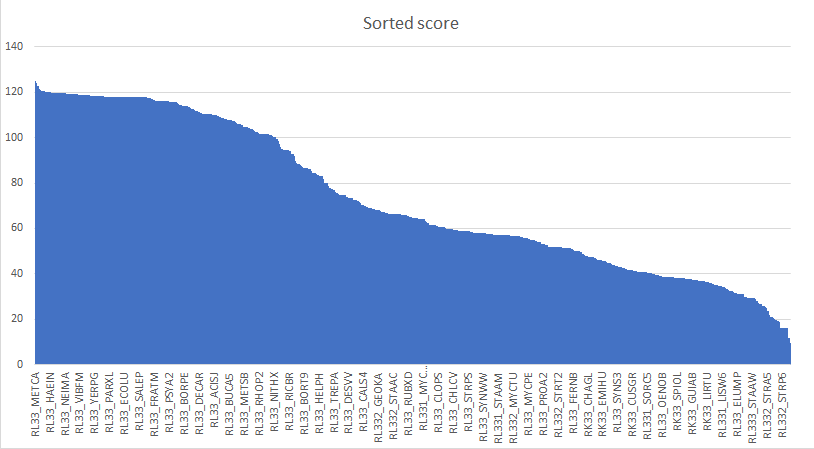 |
По гистограмме с дополнительными вычислениями для проверки корректности был вычислен порог - 88,6. Для него мы имеем оптимальное соотношение величин Sensitivity и Specificity - за счет того, что вторая величина имеет порогоое значение 1, а первая ограничивает точность выборки - примерно составляющую 40 процентов. То есть не набрав неверны последовательностей, мы рискуем потерять около 60% - не утешительная картина, но лучшая из возможных. Ниже представлен данные для этого score:
| 88,6 | Истинные классы(1/0) | |
|---|---|---|
| Предсказательные классы (1/0) | 314 | 0 |
| True | 41 | 586 |
| Sensitivity: 0,36 | Specificity: 1 | Precision: 1 |
Таким образом, вычислим precision порога и профиля: в первом случае считаем процент верных находок относительно всех находок по профилю, во втором случае считаем тот же процент, но в новой выборке, отсекая все до порога. Precision порога мы только что рассчитали.
| Precision порога | Precision профиля |
|---|---|
| 1 | 0,39 |
Вывод: Означенный профиль достаточно функционален в плане определения принадлежности семейству определённой белковой последовательнсти со скором выше порога, однако, он не самый идеальный из возможных. 39% находок по базе это не достаточно большая величина. А выставленный порог, отсекающий 60% верных находок как неверные благоприятной работе не способствует. И хотя величины сравнимы, показания могли бы быть и получше.
На главную страницуВернуться назад
©Solonovich Vera,2017