
 |
Учебный сайт Юдиной А.С. |
Главная |
Обо мне |
Семестры |
||||||||
Реконструкция эволюции доменной архитектуры. Построение выравнивания представителей домена Pfam белков с разной доменной архитектурой.Для работы над данным пректом было выбрано семейство доменов LPMO_10, представители которого находятся в структуре
хитин-связывающих белков (я встречалась с этим белком в первом семестре chitin-binding protein.
Этот домен встречается в ассоциации
с широким набором связывающих целлюлозу доменов. Рассматриваемое семейство доменов состоит из двух очень родственных
белков, которые функционируют как С-1 и С-4 расщепляющие полисахариды монооксидазы. Так же
этот домен встречается в белках, функция которых на сегоднящний момент не известна.
В программе Jalveiw было открыто выравнивание всех, последовательностей содержащих выбранный домен. Раскраска Clustal. Проект выравнивания:lpmo_project.jvp, выравнвание в формате fasta: lpmo.mfa. I этапБыла получена информация об архитектуре всех последовательностей, входящих в данное семейство. Результаты
представлены в виде таблицы (лист PF03067).
II этапТеперь были выбраны две доменные архитектуры:
Теперь из сводной таблицы с применением фильтров были выделены AC последовательностей, содержащих
данные доменные архитектуры.
III этапНа основе сводной таблицы была составлена таблица содержащая AC последовательностей, имеющих доменные архитектуры 1 и 2. В помощью функции ВПР в нее
была перенесена информация по таксономии, добавлен столбец с длинами домена LPMO_10 (из листа PF03067).
Результат работы: PF03067.xlsx IV этапИз исходного выранвивания, полученного на I этапе, были выбраны только выбранные последовательности.
Полученное выравнивание открыто в Jalview, удалены пустые позиции, убраны плохо выровненные концевые участки, удалены явно выбивающиеся последоватлеьности. Произведено деление на группы, согласно принадлежности к доменной архитектуре, выполнена раскраска Clustal по группам с 20% идентичности.
Рисунок 1
В обеих группах наблюдается множетсво гэпов, при этом первая группа 1 архитектуры содержит несколько больше консервативных позиций, для архитектуры 2 (особенно первые 10 последовательностей) характерно наличие участков, не встречающихся в других последовательностях. Но имеются абсолютно консервативные позици идля обеих групп.
Проект выранивания: lpmo_group_project.jvp. Построение филогенетического дерева последовательностей домена.На основе отредактированного в Jalview выравнивания было построено филогенетическое дерево в программе MEGA. Метод - Neighbor Joining, проверка - bootstrap с числом копий 100. Обозначения: 1 - первая архитектура, 2 - вторая архитектура, А - Actinobacteria, P - Proteobacteria. 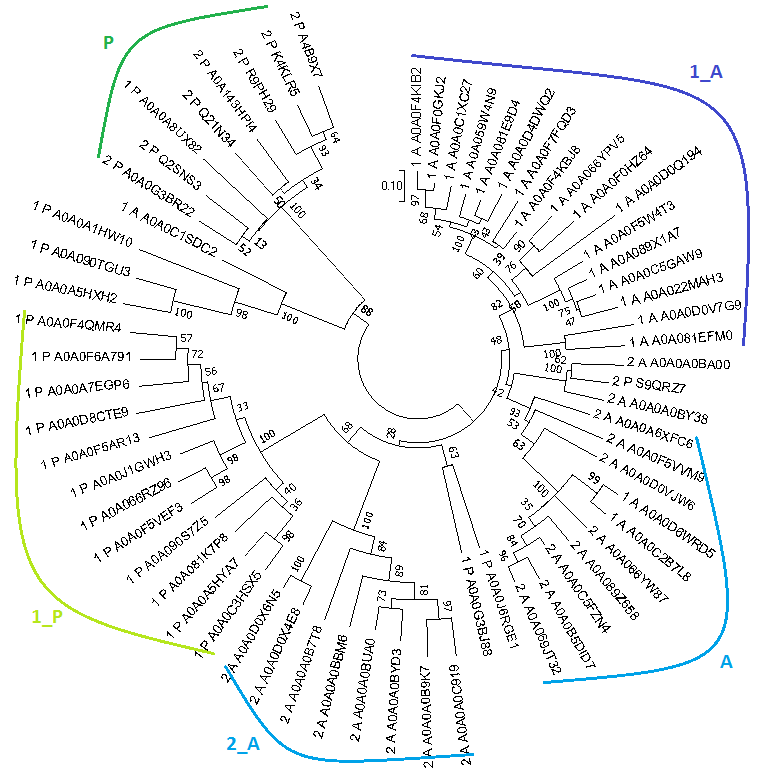 Из дерева видно, что клады соответсвуют либо связке архитектура+подтаксон (2_А, 1_Р, 1_А), либо подтаксону. При этом дерево явно поделено на две "части", соответсвующие подтаксонам. Можно сделать вывод, что архитектуры возникли раньше, чем разделились подтаксоны, так как в обоих подтаксонах присутствуют обе архитектуры. Однако в кладах обоих подтаксонов встречаются ветви, несущие последовательности другого подтаксона, что делает анализ затруднительным, получается четко разделить подтаксоны нельзя. Скобочная форма дерева: nj_lpmo.nwk. |
||||||||||
© Юдина Анастасия, 2016