Abstract
Данная работа представляет собой исследовательский мини-проект, направленный на изучение контактов биомолекул в составе комплексов, а также контактов биомолекул с лигандами. В качестве комплекса для изучения был выбран и рассмотрен трансмембранный белок ABCB10.
В ходе работы была получена информация о белок-белковых контактах, в частности ковалентных и водородных связях, солевых мостиках и гидрофобных контактах. Также была исследована плотность упаковки атомов в гидрофобном ядре. Помимо этого были рассмотрены лиганд-биомолекулярные и нк-белковые контакты и сделано предсказание трансмембранных участков.
1. Introduction
ABCB10 protein is a carrier of the mitochondrial ATP-binding human cassette (ABC). Protein is highly expressed in erythroids, cells that tend to become red blood cells. ABCB10 is embedded in the inner mitochondrial membrane and it is possible that protein pumps its substrate from the mitochondrial matrix into the intermembrane space. Protein ABCB10 plays an important role in mammals, it is involved in the formation of hemoglobin and red blood cells, protects mitochondria from oxidative stress. Scientists have proposed several alternative functions for ABCB10, including transport of heme biosynthesis precursors, stabilization of the SLC25A37 iron transporter, and peptide transport function. The discussion about the role of ABCB10 continues, since there has not been found biochemical evidence of its functions.[1]
| PDB ID | 4AYT |
| PubMed | 23716676 |
| Uniprod ID | Q9NRK6 (ABCBA_HUMAN) |
Общая информация о лигандах структуры 4AYT
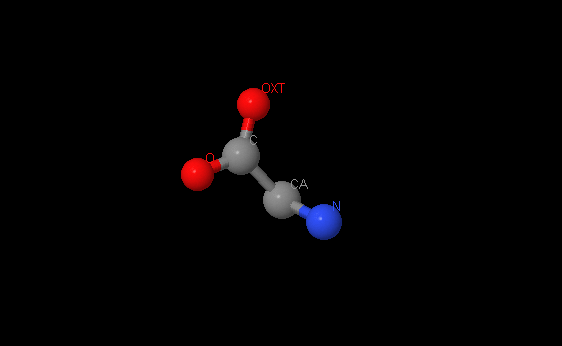
Название: Глицин
Брутто-формула: C2H5NO2
Название по UIPAC: 2-aminoethanoic acid
Молярная масса: 75.07 г/моль
DrugBank ID: DB00145
Порядковый номер: 1717
Аминокислота. Также является тормозящим нейромедиатором.
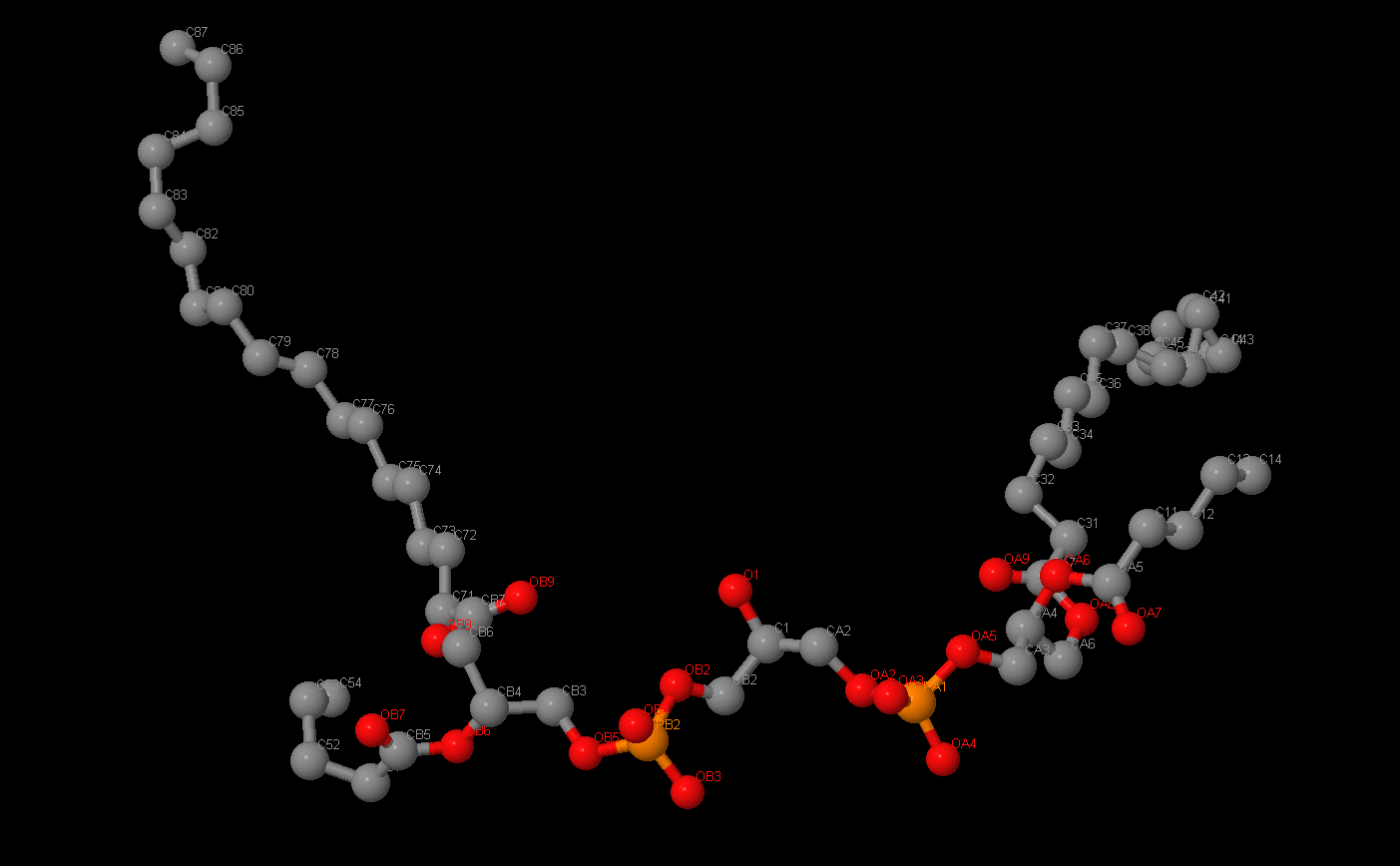
Название: Кардиолипин
Брутто-формула: C81H156O17P2
Название по UIPAC:[3-[[(2R)-2,3-di(octadecanoyloxy)propoxy]-oxido-phosphoryl]oxy-2-hydroxy-propyl] [(2R)-2,3-di(octadecanoyloxy)propyl] phosphate
Молярная масса: 1464.04 г/моль
DrugBank ID:DB03429
Порядковый номер: 1721
"Двойной" фосфолипид, так как вместо 2 остатков жирных кислот имеет 4. Важный компонент внутренней митохондриальной мембраны, где он составляет 20% всех липидов. Обычно находится в метаболически активных клетках сердцами скелетных мышц, в мембранах их митохондрий. Является изолятором и стабилизатором белковых комплексов, связанных с транспортом электронов. Он также "склеивает" эти комплексы.

Название: ион магния
Формула: Mg2+
Название по UIPAC: magnesium(+2)
Молярная масса: 24.30 г/моль
DrugBank ID: DB01378
Порядковый номер: 801
Необходимый участник синтеза белка на всех этапах этого процесса.
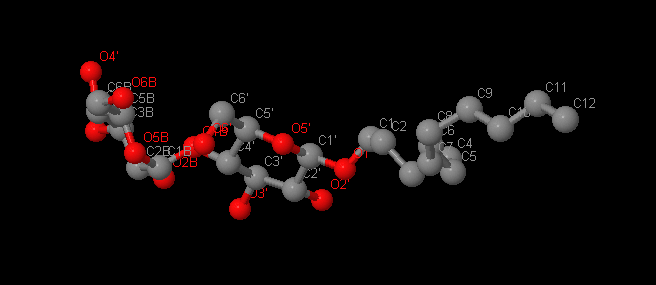
Название: Додецил-Бета-Д-Мальтоксид
Брутто-формула: С24H46O11
Название по UIPAC: (2R,3R,4S,5S,6R)-2-[(2R,3S,4R,5R,6R)-6-dodecoxy-4,5-dihydroxy-2-(hydroxymethyl)oxan-3-yl]oxy-6-(hydroxymethyl)oxane-3,4,5-triol
Молярная масса: 510.62 г/моль
DrugBank ID: DB03279
Порядковый номер: 1720
Детергент для стабилизации и активации ферментов.
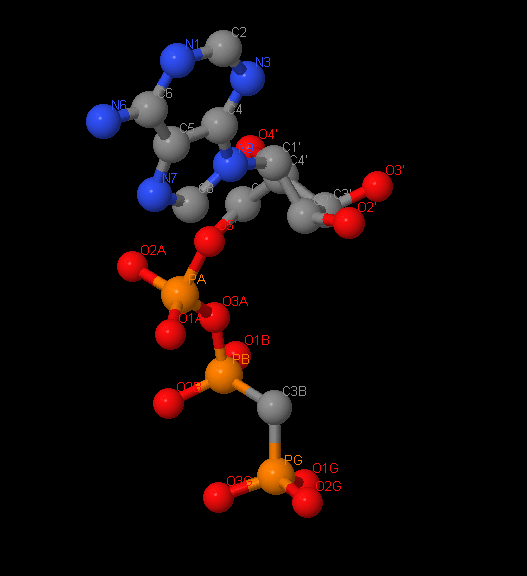
Название: Сложный эфир фосфометилфосфоновой кислоты и аденилатциклазы
Брутто-формула: C11H18N5O12P3
Название по UIPAC: [[[(2R,3S,4R,5R)-5-(6-aminopurin-9-yl)-3,4-dihydroxy-oxolan-2-yl]methoxy-hydroxy-phosphoryl]oxy-hydroxy-phosphoryl]methylphosphonic acid
Молярная масса: 75.07 г/моль
DrugBank ID: DB03909
Порядковый номер: 900
Аналог АТР(Аденозинтрифосфата), где атом кислорода, соединяющий бета и гамма фосфаты, заменен на атом азота. Это сильный конкурентный ингибитор растворимый и мембраносвязанной АТРазы, а также тормозит АТР-зависящие реакции окислительного фосфорилирования.
2. Материалы и методы
...
3. Атлас контактов
3.1 Белок-белковые взаимодействия
3.1.1 Ковалентные связи
Disulfide bond is the type of covalent bonds which connects sulfur atoms (S-S) in two cysteine amino acids. Disulfide bridges in proteins are formed between thiol groups (-SH) of cysteine residues as a result of oxidative folding. The angle between planes where both sulfur atoms are in is approximately 90°. Another sulfur containing amino acid, methionine are not able to form disulfide bridges. We found 3 molecules of cysteine in ABCB10 protein, but they don't form disulfide bridges because of a big distance
Команда "rectrict Cys" позволила нам убедиться в отсутствии данного типа связей.
Peptide bond — bond which links amino acids in protein. Pairing between C-N and carboxyl group C=O makes peptide bond 10% shorter that it should be. The bond becomes double binding, and resonant structures appear. Since it is difficult to rotate two forms of peptide bond emerge (cys- and trans isomers). Cis conformation is energetically don't have advantageous. Therefore, almost all peptide groups in proteins are in trans conformation[2] In ABCB10 protein peptide bonds were measured. The average length of bonds is 1.35 Å.
Результаты измерений представлены в следующей таблице:
| # | Atomic names | Bond lendth (Å) | |
|---|---|---|---|
| Alpha-helices | |||
| 1 | C(183VAL)-N(184ILE) | 1.36 | |
| 2 | C(185SER)-N(186MET) | 1.37 | |
| 3 | C(187SER)-N(188ALA) | 1.36 | |
| 4 | C(383HIS)-N(384VAL) | 1.35 | |
| 5 | C(381GLU)-N(382ALA) | 1.35 | |
| 6 | C(402THR)-N(403GLY) | 1.35 | |
| 7 | C(271LEU)-N(22IlE) | 1.35 | |
| 8 | C(377TYR)-N(378ALA) | 1.35 | |
| 9 | C(371MET)-N(372THR) | 1.35 | |
| 10 | C(654ILE)-N(655LEU) | 1.34 | |
| 310-helix | |||
| 1 | C(622GLU)- N(623PHE) | 1.35 | |
| 2 | C(620PRO)-N(621GLN) | 1.36 | |
| 3 | C(621GLN)-N(622GLU) | 1.33 | |
| Beta-sheets | |||
| 1 | C(551ILE)- N(552SER) | 1.35 | |
| 2 | C(552SER)- N(553LEU) | 1.34 | |
| 3 | C(553LEU)- N(554ASP) | 1.36 | |
| 4 | C(705ASP)- N(706GLN) | 1.34 | |
| 5 | C(711GLU)- N(712THR) | 1.34 | |
3.1.2 Hydrogen bonds
Водородная связь — тип взаимодействия электростатической и частично ковалентной природы. Связь формируется при наличии атома электроатрицательного элемента и связанного с другим электроотрицательным элементом атома водорода, находящимися на достаточно близком расстоянии. Результатры измерений представлены в таблице:
| # | Atomic names | Bond length (Å) | N-H-O angle (°) |
|---|---|---|---|
| Alpha-helices | |||
| 1 | O(393 PHE) – N(397 GLY) | 3.13 | 149.5 |
| 2 | O(398 PHE) – N(402 THR) | 2.78 | 166.4 |
| 3 | O(406 GLY) – N(410 VAL) | 3.37 | 146.7 |
| - | Average | 3 | 155.97 |
| 310-helix | |||
| 1 | O(662 GLY)- N(625 THR) | 3.4 | 103.7 |
| 2 | O(621 PRO)-N(624 ASN) | 3.12 | 127.7 |
| - | Average | 3.267 | 115.7 |
| Beta-sheet | |||
| 1 | O(517 ILE) – N(492 LEU) | 2.91 | 163 |
| 2 | O(703 VAL) – N(710 THR) | 2.86 | 165.7 |
| 3 | O(495 LYS) – N(550 THR) | 3.02 | |
| - | Среднее значение | 3.57 | 158.53 |
Как видно из таблицы, водородные связи в бета-слое по расстоянию между донорной карбонильной и акцепторной иминогруппой являются слабыми, в то время как у играющих основную роль в поддержании структуры альфа-спиралей водородные связи достаточо сильные[5]
Посмотреть визуализацию водородных связей в структуре, а также посмотреть скрипт можно в отдельном разделе.
Интересный случай, когда формирование водородных связей (выделены циановым), в альфа-спирали (534-541 остатки) не соотвествует стандратному паттерну представлен на Изображении 1. Формирование подобных водородных связей может быть обусловлено повышенной кислотностью митохондриального матрикса (обоснование положения изложено в пункте 4).
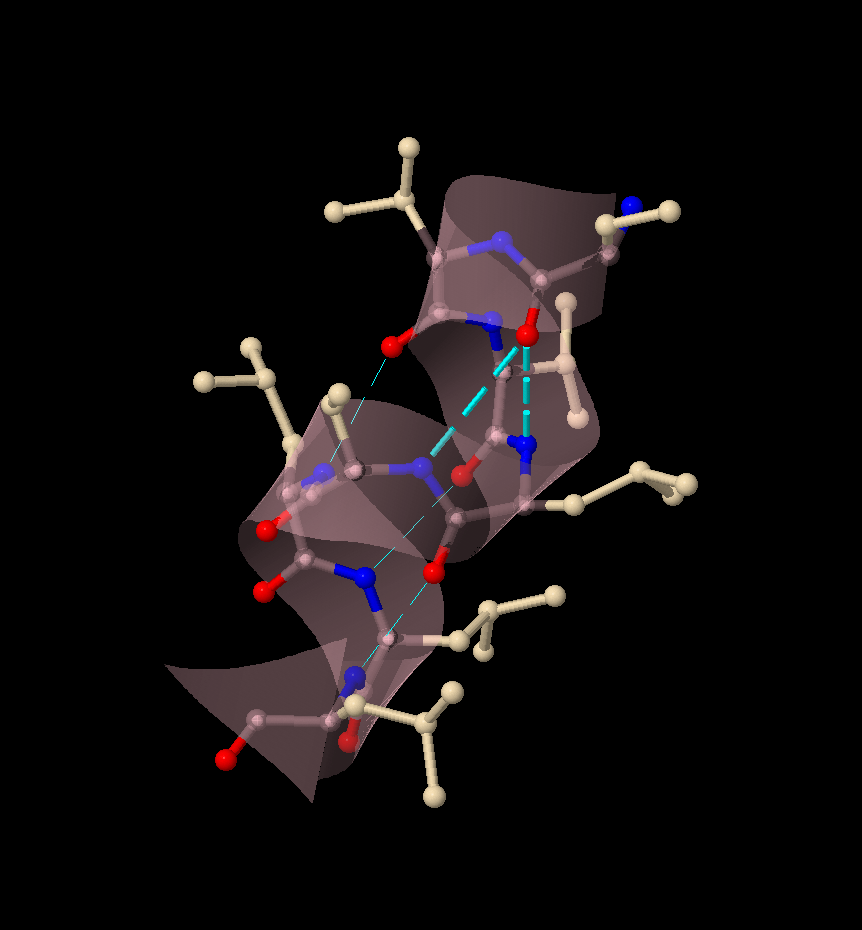
3.1.3 Salt bridges
Iogenic groups (residues of glutamic, aspartic acids; histidine, lysine and arginine) participate in ionic interactions, forming the so-called salt bridges. Negatively charged carboxyl groups of the Asp and Glu radicals and positively charged groups of the Lys, Arg, or His acids are involved in the interactions. These interactions are possible both inside the protein and between different molecules.
There are several approaches to finding salt bridges. You can that manually, to do that you’ll have to isolate the polar residues of amino acids and color them in accordance with the charge. And then visually look for those amino acid residues that most likely can form an ionic bond. You can make a different approach and use the calculator of protein interactions, ionic ones in particular.
We used both ways, but for the sake of fidelity we decided to focus on the second, which we used PIC: Protein Interactions Calculator for. According to machine calculations, there are 25 salt bridges in the protein totally.
Two of them were examined in detail: one between [GLU] 356 and [ARG] 357, the second between [LYS] 650 and [ASP] 593. You can see the result in the Image 2 and Image 3 respectively.
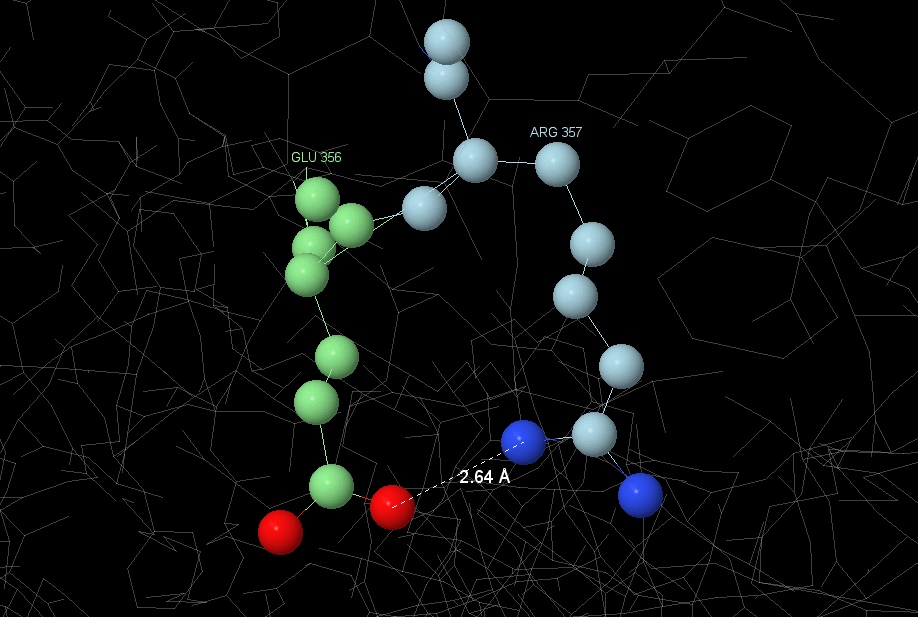
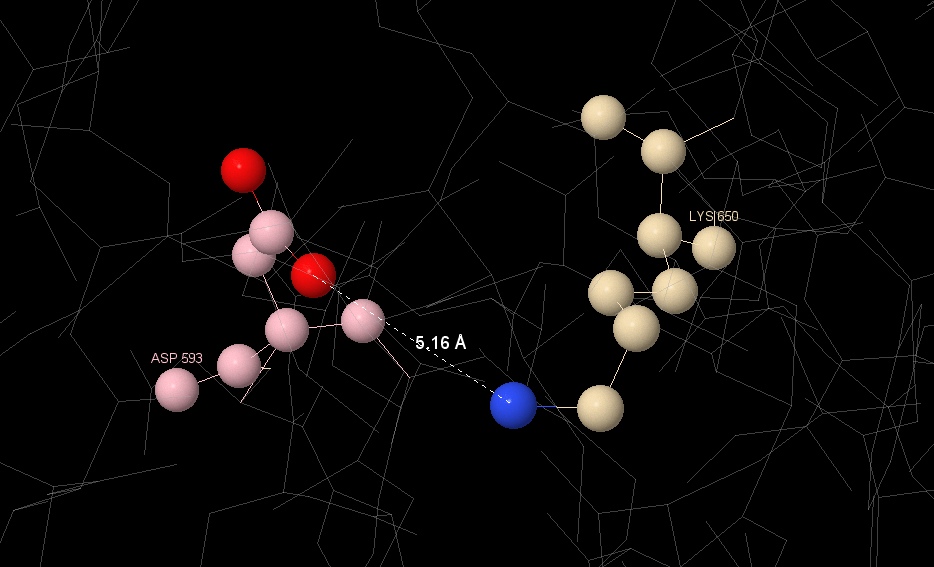
3.1.4 Hydrophobic interactions
[PHE] 398:A was selected to determine the packing density in the hydrophobic core. A hydrophobic residue was defined as myresidue. At the distance of 1 Å from the residue, no surrounding atoms are observed.
The first surrounding atoms appear at the distance of 2 Å. At the distance of 4 Å we can see that the residue is completely covered by the atoms. The approximate distance between not covalently bind atoms is 3.35 Å, which is less than the diameter of water 4.916 Å. Therefore another molecule can not fit between surroundings atoms.
Stacking interactions
Stacking interactions are non-covalent interactions between organic compounds containing aromatic systems. They are most widely represented in DNA, where they increase the stability of the molecular structure, but are also found in proteins.
In proteins, this effect can be realized with aromatic amino acids - Phe, Tyr, Trp. The so-called T-stacking is characterized by the following features: hydrogen atoms with a partial positive charge of one aromatic system are directed to the center of the other aromatic system perpendicular to the plane of the latte[3].
The search for stacking interactions in the protein was carried out with the help of the Protein Interactions Calculator[4]. Calculator output:
| # | Position | Residue | Position | Residue | Dihedral Angle |
|---|---|---|---|---|---|
| 1 | 262 | PHE | 263 | PHE | 78,72 |
| 2 | 332 | TYR | 398 | PHE | 75,4 |
| 3 | 488 | PHE | 565 | TRP | 103,49 |
| 4 | 501 | TYR | 544 | TYR | 87,13 |
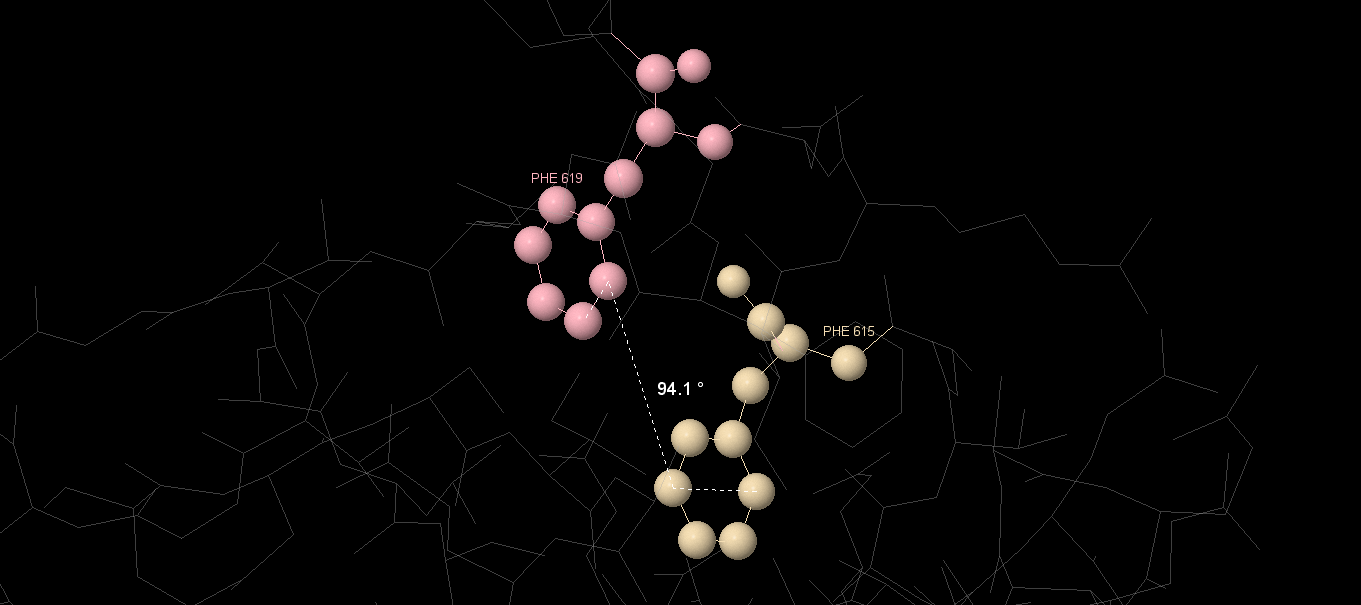
| 5 | 615 | PHE | 619 | PHE | 96,33 |
Graphical images of some of the found interactions are shown on the figures. By analyzing the angles between amino acid residues, we can suppose that these are T-stackings (angle ≈90 °)
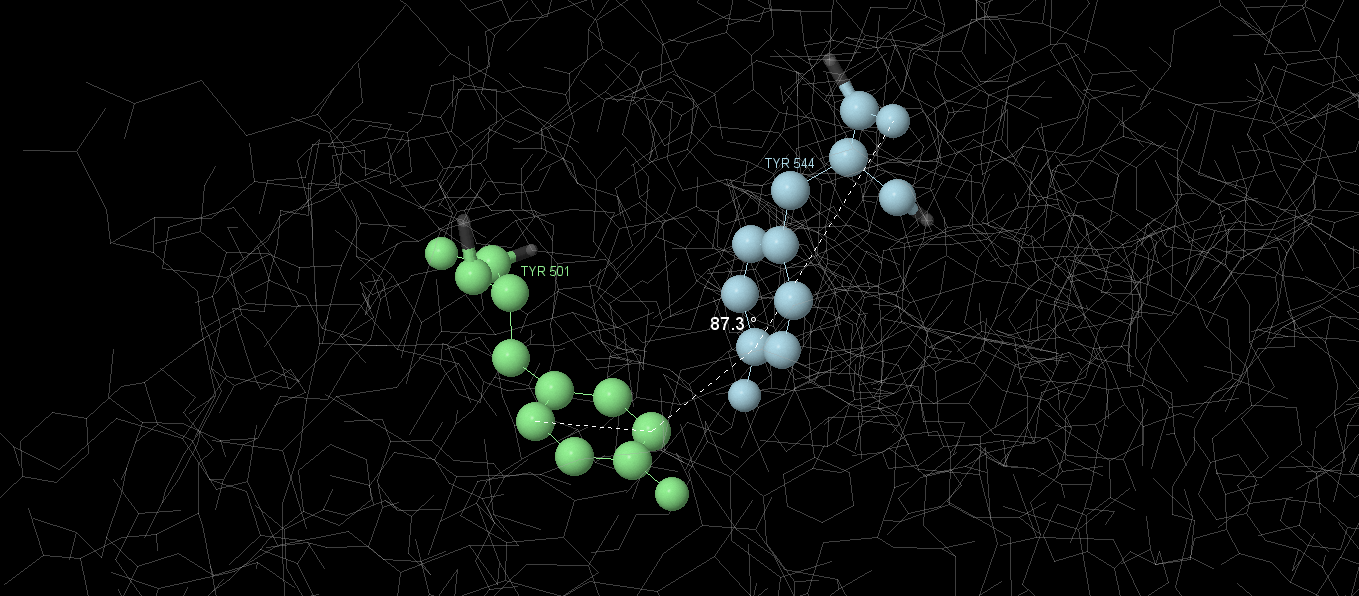
3.2 Ligand-biomolecular contacts
3.2.1 Mg

Магний координационно пришит к ACP900 (аналог ATP), а также к Ser799 и Gln802 через кислород (с остатками аминокислот он может быть связан или через кислород или через электростатические взаимодействия).
3.2.2 LMT

Возможно образование стэкинг-взаимодействий через TYR200 (расстояние между ароматическими элементами предсказывает высокую вероятность образования такого типа контакта). Также, расстояние <4Å между LYS1710 и ASP1714 предполагает наличие солевых мостиков. Водородные связи возможны, но большое расстояние ставит их существование под вопрос.

3.2.3 Gly

Возможно образование стэкинг-взаимодействий через TYR200(расстояние между ароматическими элементами предсказывает высокую вероятность образования такого типа контакта). Также, расстояние <4Å между LYS1710 и ASP1714 предполагает наличие солевых мостиков. Водородные связи возможны, но большое расстояние ставит их существование под вопрос.
3.2.4 CDL

Основной контакт с белком – через водородные связи(расстояние меньше 3Å).
3.2.5 ACP

Кроме уже описанных координационных связей с Mg1717, высока вероятность образования стэкинг-взаимодействий с TYR501, PRO502, TYR544(длина связи<12Å), а также водородных связей(длина связей в районе 3Å).
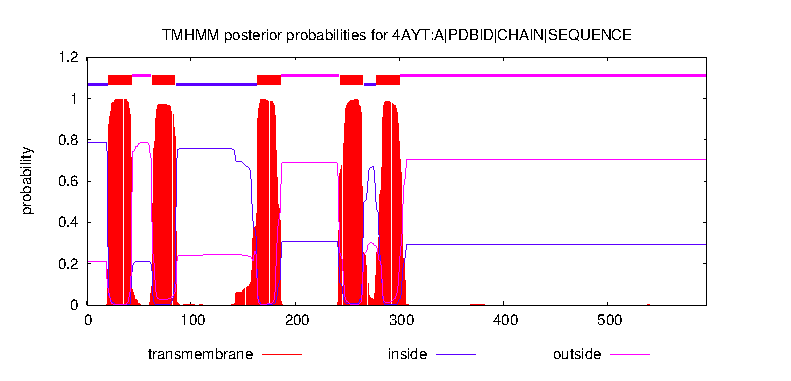
4. Положение в мембране
Ресурс TMHMM позволил определить наиболее вероятное расположение частей белка относительно мембраны. Одна субъединица, предположительно, имеет пять трансмембранных участков, представленных альфа-спиралями. Вероятностное распределение представленно на Графике 1, предполагаемое расположение частей по аминокислотным остаткам можно изучить в таблице:
| In the mitochondrial matrix | 1-20 | 86-163 | 266-277 | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| Transmembrane part | 21-43 | 63-85 | 164-186 | 243-265 | 278-300 | ||||||
| Intermembrane space | 44-62 | 187-242 | 301-595 |
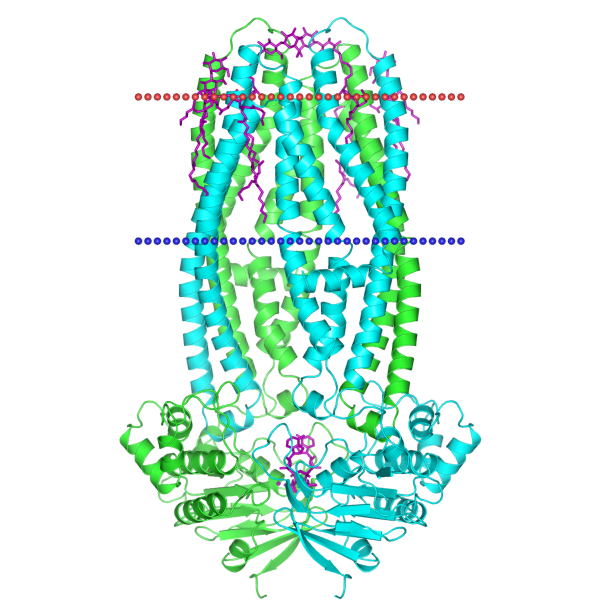
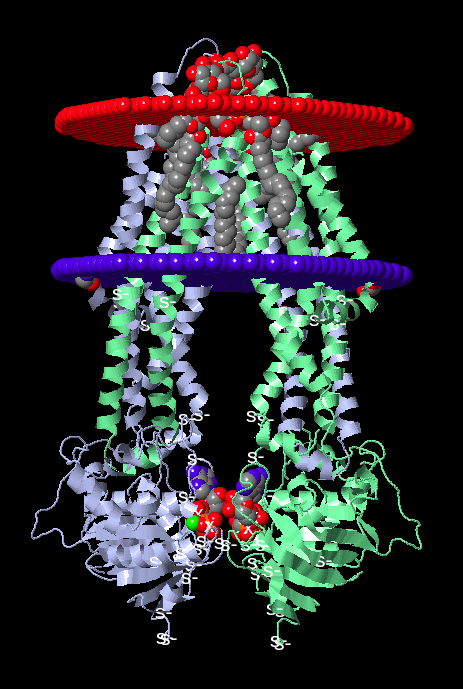
on the images 6 and 7 below представлена визуализация расположения полной структуры в мембране с использованием JSmol и OPM соответственно.
5. Autor contributions
Boltysheva A.I. studied all the questions related to ligands: the review of the ligands was carried out, the research and the measurement of ligand-protein interactions, and a script was prepared to represent the relevant types of interactions.
Vashurkina S.A. was a link between relatively independent working members of the group, analyzed the characteristics of the salt bridges and compiled the script. In addition, she made a significant contribution to the study of hydrogen and hydrophobic interactions; the latter includes the consideration of stacking interactions. Her contribution is also complemented by writing a resume.
Kamkina T.D. wrote an introduction and studied the hydrophobic interactions. She conducted a literature analysis and studied the hydrophobic core in the protein. Her work also included the description of the characteristics of peptide bonds and disulfide bridges, as well as the preparation of scripts for the corresponding applets.
Skvortsov D.P. was engaged in website design, compilation of results from other members of the research group, as well as the consideration of hydrogen bonds together with S. Vashurkina. He carried out the adaptation of graphic, textual information and scripts for the site. He was involved in editing, coordination, and consultation. He calculated the location of protein structures in the membrane, and embodied the visualization of the protein in the mitochondrial membrane.
6. Acknowlegments
Выражаем свою благодарность преподавателям практической биоинформатики Факультета биоинженерии и биоинформатики Московского государственного унивеститета им. Ломоносова за предоставленную возможность проявить свои способности, а так же наставничество и помощь в разрешении возникающих вопросов.
7. References
-
Structures of ABCB10, a human ATP-binding cassette transporter in apo- and nucleotide-bound states Chitra A. Shintre, Ashley C. W. Pike, Qiuhong Li, Jung-In Kim, Alastair J. Barr, Solenne Goubin, Leela Shrestha, Jing Yang, Georgina Berridge, Jonathan Ross, Phillip J. Stansfeld, Mark S. P. Sansom, Aled M. Edwards, Chas Bountra, Brian D. Marsden, Frank von Delft, Alex N. Bullock, Opher Gileadi, Nicola A. Burgess-Brown, and Elisabeth P. Carpenter
PNAS June 11, 2013 110 (24) 9710-9715;
https://doi.org/10.1073/pnas.1217042110
Edited* by Wayne A Hendrickson, Columbia University, New York, NY, and approved April 24, 2013 (received for review October 2, 2012)
-
Course "Химические основы биологических процессов" by A.V. Bacheva and M.E. Zvereva for the Faculty of bioingeniering and bioinfarmatics, MSU, 2019
-
R Luo, H S Gilson, M J Potter, and M K Gilson “The physical basis of nucleic acid base stacking in water.” Biophys J. 2001 Jan; 80(1): 140–148.
doi: 10.1016/S0006-3495(01)76001-8
PMCID: PMC1301220
PMID: 11159389
- P I C : Protein Interactions Calculator, Molecular Biophysics Unit, Indian Institue of Science, Bangalore.
Applet and the scripts