Для примера, было взято слово ITALY, состоящее из таких букв, которыми называются аминокислоты.
Для слова было теоретически предсказано количество случайных находок во всех записях SwissProt: 191670831*5,96*5,34*8,25*9,66*2,92*10^(-10)=141,96
Где 191670831 - это число васех аминокислотных остатков во всех последовательностях из базы данных, а остальные множители - частоты соответствующих
аминокислот.
При помощи PROSITE экспериментально было определно количество раз, которое данный мотив встречается в последовательностях: 140.
Как видно на данном примере,- экспериментально и статистически полученные значения практически не отличимы, что говорит о том,
что белковые последовательности построены случайным образом)
Альтернативое и дополнением BLAST является поиск гомологичных последовательностей по так называемм паттернам.
Паттерны как правило получают при анализе множественного выравнивания уже найденых гомологов. По такому варавниванию часто просто бывает определить
какой-нибудь консервативный, специфичный для данной группы участок, например,- активный центр. При этом, мы считаем, что это важный участок,
медленно эволюционирующий и характерный для всех потенциальных гомологов. Далее, для этого участка составляется усредненная запись,
удовлетворяющая всем последовательностям выравнивания - это и есть паттерн данного участка (или всей) последовательности.
Синтаксис составления паттернов:
Готовый паттерн является матрицей по которой алгоритм PROSITE отбирает подходящие последовательности из всех возможных в базе данных.
Для объекта исследования - белка CRH_BACSU, для которого уже было составлено множественное выравнивание, был составлен ряд
паттернов - от очень строгих, до слабых. Результаты поиска представлены в таблице 1.
Таблица 1.
| Характеристика паттерна | Паттерн | В скольких последовательностях банка Swiss-Prot найден мотив, удовлетворяющий паттерну? | Все ли последовательности из вашего выравнивания найдены? (если нет, то сколько) | Вероятность получения случайной последовательности по такому паттерну |
| Сильный | [VIMLY]-x(4)-[GKA]-x(3)-[RFK]-x(5)-[VHAT]-x(2)-[ALTI]-x(6)-[VQIA]-x-[LVIF]- x(6,9)-[VLAI]-[NDST]-x(2)-[SNK]-x(3)-[LSVI]-x(10)-[LVIF]-x-[LVIF]-x(3,8)-[GS]-x(5)- [AVLS]-x(3)-[ELIV] |
51 | 1 - собственно B. subtilis | 1/10^6 |
| Средний | {RLEN}-x(4)-[GK]-x(3)-[RFK]-x(5)-[VHAT]-x(2)-[AITVLQ]-x(6)-[VQIA]-x-[LVIF]-x(6,12)- [KGRAP]-[SNK]-x(3)-[LSVI]-x(3)-[GVANES]-x(12,17)-[GS]-x-[DQKGRE]-x(7)-[ELIV] |
117 | 3 | 29/10^5 |
| Слабый | [LIVA]-[NTSD]-x-[KPRAG]-[SNK]-x(3)-[LSVI]-[LMITQ]-X-[LIMVA]-x-[VLAIC]-x(5)-[VILF]-x- [LIVF] |
401 | 6 | 87/10^5 |
| Очень слабый | [LIVA]-[NTSD]-x(2)-[SNK]-x(3)-[LSVI]-x(2)-[LIMVA]-x-[VLAIC]-x(5)-[VILF]-x-[LIVF] | 4252 | 20 | 1029/100000 |
Для сильного паттерна.
С одной стороны, все хиты являются очень близкими гомологами к CRH_BACSU (см рис 1). C другой стороны, кроме B. subtilis
не было найдено ни одной последовательности из множественного выравнивания. Не совсем понятно, как такое могло получится: присоставлении паттерна
учитывались все последовательности, а не только CRH_BACSU. Возможно такой результат частично связан с тем, что поиск роводился в разных базах данных:
в случае BLAST (для получения множественного выравнивания - по RefSeq, а в случае PROSITE - по SwissProt.
Однако, задачу можно считать выполненной, так как по паттерну были найдены
новые близкие гомологи.
Вероятность нахождения рандомной последовательности, подходящей под данный паттерн: 1/10^6.
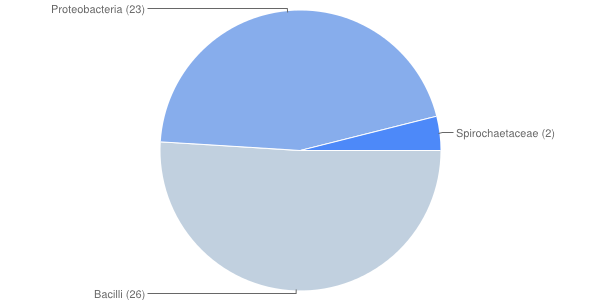
Рис 1. Распределение хитов по таксонам.
Для среднего паттерна.
Вероятность нахождения рандомной последовательности, подходящей под данный паттерн: 29/10^5.
Как и в случае сильного патерна, большую часть хитов составляли белки из близкородственных с B. subtilis организмов (см рис 2, участвующие
в передаче или транстпорте фосфата, что говорит о объективности паттерна с функциональной
стороны вопроса.
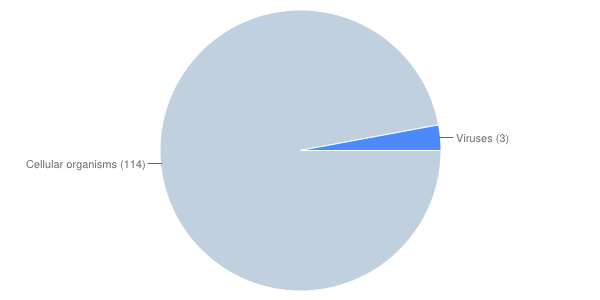
Рас 2. Распределение хитов по таксонам.
Для слабого паттерна.
Веротность нахождения рандомной последовательности, подходящей под данный паттерн: 87/10^5.
Сохраняется блок последовательностей, относящихся к транспоретрам фосфата (Phosphocarrier protein HPr).
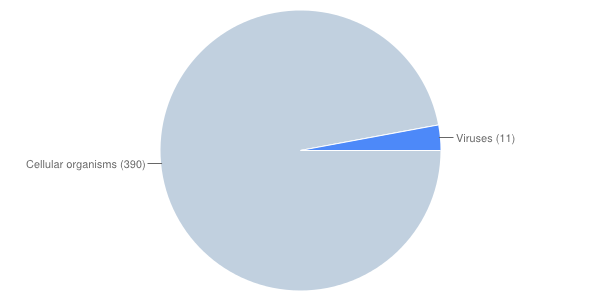
Рас 3. Распределение хитов по таксонам.
Для очень слабого паттерна.
Веротность нахождения рандомной последовательности, подходящей под данный паттерн: 1029/10^5.
Появилось гораздо больше хитов, которые использовались в множественном выравнивании.
Сохраняется четкий блок последовательностей белков-переносчиков
фосфата. Распределение хитов по таксонам представлено на рисунке 4.
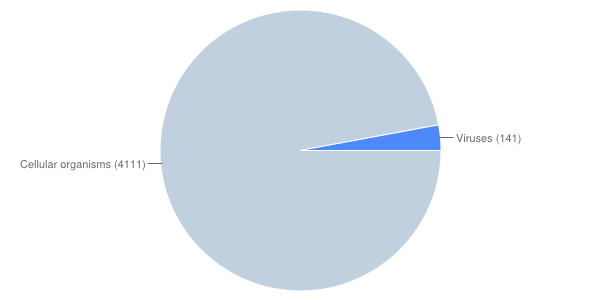
Рас 4. Распределение хитов по таксонам.
В целом, результаты поиска можно считать удовлетворительными: были найдены многие последовательности по которым составлялся паттерн,
но большую часть находок составляли бактерии филлума Firmicutes, и близких к нему, что логично, так как именно к этой группе относится B. subtilis и именно
в ней широко распространены близкие гомологи CRH_BACSU.
Среди всех находок при всех вариантах паттерна выделяется обширный кластер последовательностей из разных организмов,
в котором записи охарактеризованы как Phosphocarrier protein HPr (белок-переносчик фосфата HPr или HPr-like protein Crh),
к которым относится и СRH_BACSU. Следовательно, можно сделать вывод, что поиск прошел удачно - были выявлены новые явные функциональные
гомологи нашего белка.
| Идентификатор документа Prosite (AC) | Название мотива | Краткое описание мотива | Тип подписи (паттерн, профиль) | Паттерн (если это паттерн) | Специфична ли подпись? | Сколько мотивов нашлось в белке? |
| PS51094 | ACT_SITE | Pros-phosphohistidine intermediate; for HPr activity | сайт | нет | специфично | 1 |
| PS51350 | MOD_RES | Phosphoserine; by HPrK/P | аминокислота | нет | специфично | 1 |
| PS00589 | PTS_HPR_SER | PTS HPR domain serine phosphorylation site signature | паттерн | GKkVNaKSIMGLMsLA | специфично | 1 |
| PS00009 | AMIDATION | Amidation site | паттерн | x-G-[RK]-[RK] (x is the amidation site) | неспецифично | 1 |
| PS00006 | CK2_PHOSPHO_SITE | Casein kinase II phosphorylation site | паттерн | [ST]-x(2)-[DE] | неспецифично | 1 |
| PS00008 | MYRISTYL | N-myristoylation site | паттерн | G-{EDRKHPFYW}-x(2)-[STAGCN]-{P} | неспецифично | 1 |