Реконструкция деревьев по нуклеотидным последовательностям. Анализ деревьев, содержащих паралоги
1. Построение дерева по нуклеотидным последовательностям
Итак, сначала - правильное дерево с выбранными бактериями:
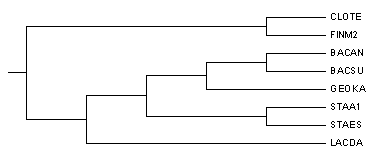
Были найдены последовательности 16S рибосомальной РНК каждой из них (c помощью команды seqret).
| Бактерия | AC | Координаты 16S rRNA | Последовательность |
|---|---|---|---|
| BACAN | AE016879 | 9335..10841 | прямая |
| BACSU | AL009126 | 30279..31872 | прямая |
| CLOTE | AE015927 | 8715..10223 | комплементарная |
| FINM2 | AP008971 | 197837..199361 | прямая |
| GEOKA | BA000043 | 10421..11873 | прямая |
| LACDA | CR954253 | 45160..46720 | прямая |
| STAA1 | AP009324 | 531922..533476 | прямая |
| STAES | AE015929 | 1598006..1599559 | комплементарная |
Все последовательности были объединены в единый fasta-файл, а потом выровнены с помощью программы muscle с параметрми по умолчанию. Результат.
С помощью MEGA (метод neighbour-joining, bootstrap 100) было построено филогенетическое дерево (стоит отметить, что другими двумя методами результат точно такой же):
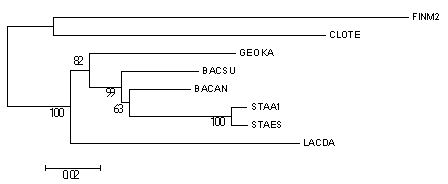
Дерево не совпадает с правильным. Как видно, сохраняется ветвь {CLOTE,FINM2} против всего остального, также верно определена ветвь {BACAN,BACSU,GEOKA,STAA1,STAES} против остального, но внутри этой ветви дерево отличается от правильного. По предыдущим заданиям, именно в этом месте и раньше возникали ошибки (если возникали).
Полученное дерево оказалось "хуже", чем дерево, построенное по белку (см. прошлое задание), там после выравниваний правильные деревья получались.
Построение и анализ дерева, содержащего паралоги
Так как у некоторых бактерий (например, BACAN) было найдено слишком много достоверных гомологов, были выбраны только некоторые из них.
Файл с последовательностями белков
Они были выровнены с помощью muscle.
Файл с выровненными последовательностями
Потом было (абсолютно теми же методами, что и в первом пункте, то есть neighbour-joining) построено дерево:
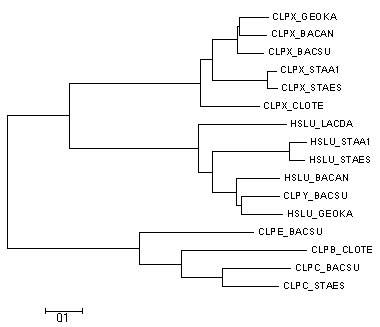
Примеры паралогов:
- CLPX_BACSU, CLPY_BACSU, CLPE_BACSU, CLPC_BACSU
- CLPX_GEOKA, HSLU_GEOKA
- CLPX_BACAN, HSLU_BACAN
- CLPX_STAA1, HSLU_STAA1
- CLPX_STAES, CLPC_STAES, HSLU_STAES
Примеры ортологов:
- CLPX_GEOKA, CLPX_BACAN, CLPX_STAA1, CLPX_STAES, CLPX_CLOTE (все они образуют одну ветвь, где каждое разветвление сопровождалось видообразованием)
- CLPC_BACU, CLPC_STAES
- HSLU_LACDA, HSLU_STAA1, HSLU_STAES, HSLU_BACAN, CLPY_BACSU, HSLU_GEOKA (СLPY как-то не вписывается в общий ряд, но мы же считаем дерево верным)