| Jmol практикум 1 |
| Jmol практикум 2 |
| Jmol практикум 3 |
| Главная страница |
Первая итерация |
|||||
| Количество находок лучше порога E-value < 0.005 | 659 | ||||
| Лучшая находка | Худшая находка | ||||
| ID | Score | E-value | ID | Score | E-value |
| Q8A015.1 | 491 | 2e-169 | B6ELS3.1 | 41.2 | 0.005 |
Вторая итерация |
|||||
| Количество находок лучше порога E-value < 0.005 | 751 | ||||
| Лучшая находка | Худшая находка | ||||
| ID | Score | E-value | ID | Score | E-value |
| Q8A015.1 | 609 | 0.0 | P57331.1 | 62.4 | 4e-09 |
Третья итерация |
|||||
| Количество находок лучше порога E-value < 0.005 | 659 | ||||
| Лучшая находка | Худшая находка | ||||
| ID | Score | E-value | ID | Score | E-value |
| Q8A015.1 | 585 | 0.0 | P57331.1 | 69.0 | 3e-11 |
Разница между E-value для последней правильной последовательности (3e-11) и первой неправильной (0.62) большая, значит, с большой вероятностью, находки действительно составляют одно семейство. Если же искать гомологи для P57331.1, то результаты следующие:
Первая итерация |
|||||
| Количество находок лучше порога E-value < 0.005 | 237 | ||||
| Лучшая находка | Худшая находка | ||||
| ID | Score | E-value | ID | Score | E-value |
| Q8K9S5.1 | 608 | 0.0 | B8GBH2.1 | 41.2 | 0.005 |
| ... | |||||
Десятая итерация |
|||||
| Количество находок лучше порога E-value < 0.005 | 597 | ||||
| Лучшая находка | Худшая находка | ||||
| ID | Score | E-value | ID | Score | E-value |
| Q8Z9A3.1 | 637 | 0.0 | Q9RC57.1 | 83.2 | 7e-17 |
То есть, если начинать с другой последовательности, можно прийти к другому результату. Белок может принадлежать не к одному семейству и 'пограничный' для одного семейства белок может оказаться типичным для другого.
При помощи команды muscle было построено множественное выравнивание гомологов G2Z3N7_FLABF.
Команда: muscle -in psiBLAST.txt -out psBL.txt, дополнительные параметры (количество итераций, максимальное время работы программы) по умолчанию.
Для облегчения дальнейшей работы из множественного выравнивания были выбраны типичные (гомологичные почти по всей длине, но и не идентичные) последовательности.
Для тех же последовательностей было построено собственное множественное выравнивание с помощью mafft.
Эти выравнивания различаются в трех местах:
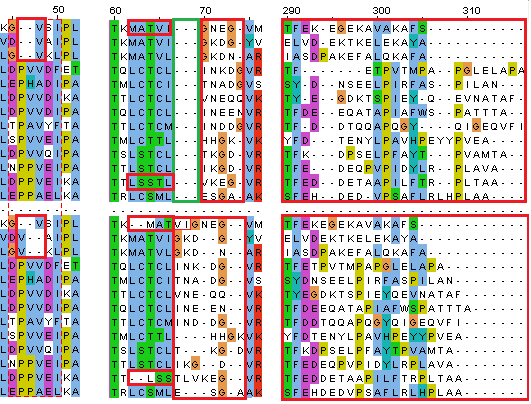
Вверху множественное выравнивание типичных последовательностей, внизу типичные последовательности множественного выравнивания.
1) Различаются положения гэпов, сверху их расстановка лучше, так как образуется колонка со сходными основаниями.
2) Для лучшего выравнивания пришлось вставить гэпы в верхнее выравнивание. В нижнем выравнивании больше гэпов и меньше хороших колонок.
3) В конце последовательности различаются сильнее всего (наверное, изменения на конце белковой цепи мало сказываются на работе белка). Здесь наоборот, больше гэпов в верхнем выравнивании, в нижнем они отсутствуют. Зато в верхнем выравнивании появляется некоторое количество сходных колонок.
В целом, множественное выравнивание, которое изначально строилось для небольшого количества последовательностей лучше. Этого следовало ожидать, второе выравнивание изначально содержало большее количество последовательностей и некоторые фрагменты были выровнены с последовательностями, не вошедшими в окончательное выравнивание. Так как концы последовательностей очень разные, то для выравнивания большого количества последовательностей наилучший результат дает выравнивание, не содержащее гэпов.
JavView project