| Jmol практикум 1 |
| Jmol практикум 2 |
| Jmol практикум 3 |
| Главная страница |
Для сортировки последовательностей по сходству было построено дерево (NeighbourJoining Using BLOSUM62).
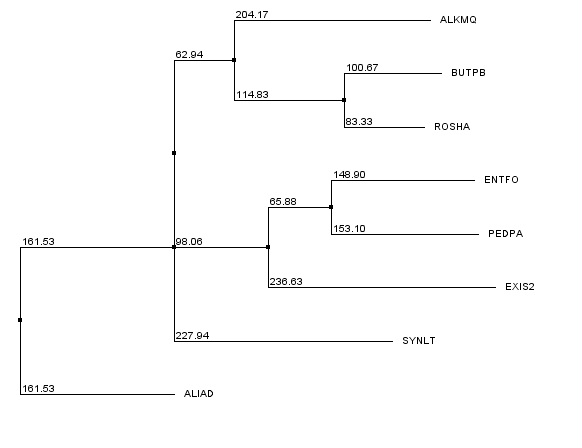
Далее было проведено выравнивание по этому дереву и расскраска двумя способами: BLOSUM62 и ClustalX.


В ходе анализа последовательностей было выделено 7 вертикальных блоков (обозначены в строке разметки blocks).
Tри последних можно объеденить в кластер, так как между ними нет гэпов (обозначены в строке разметки clusters: вертикальные блоки, входящие в кластер, отмечены символом B, участки между блоками - символом С).

Также в этой же строке разметки символом X отмечены самые длинные участки, не входящие в состав блоков и кластеров: их два и каждый из них длиной всего лишь в 8 аминокислотных остатков.
На первом участке Х аминокислоты последовательностей ROSHA/7-11 и BUTPB/7-11 гомологичны, а у остальных, скорее всего, нет.
У рассматриваемых последовательностей совпадают все аминокислоты, кроме N/K на третьей позиции.

Итого, во втором блоке из 15 аминокистот:
- абсолютно консервативных позиций 6, что составляет 40%
- абсолютно функционально консервативных 4, что составляет 26,7%
- консервативных на 70% 0
- функционально консервативных на 70% 4, что составляет 26,7%
В первом участке X из 8 аминокислот всего 1 гэп. Но это очень маленький участок, потому информация о частоте гэпов недостоверна.
К выравниванию добавлена еще одна последовательность CELLD/1-235, она выравнена с остальными путем добавления трех гэпов:

Добавлена заведомо негомологичная последовательность:

С помощью удлинения гэпов в исходном выравнивании удалось найти 52 консервативных и функционально консервативных позиции (из 239), что составляет 21,76%.
Это довольно большой процент сходства, однако, в основном, сходство имеется между единичными колонками и довольно редко между блоками.
Множественное выравнивание неродственных белков:
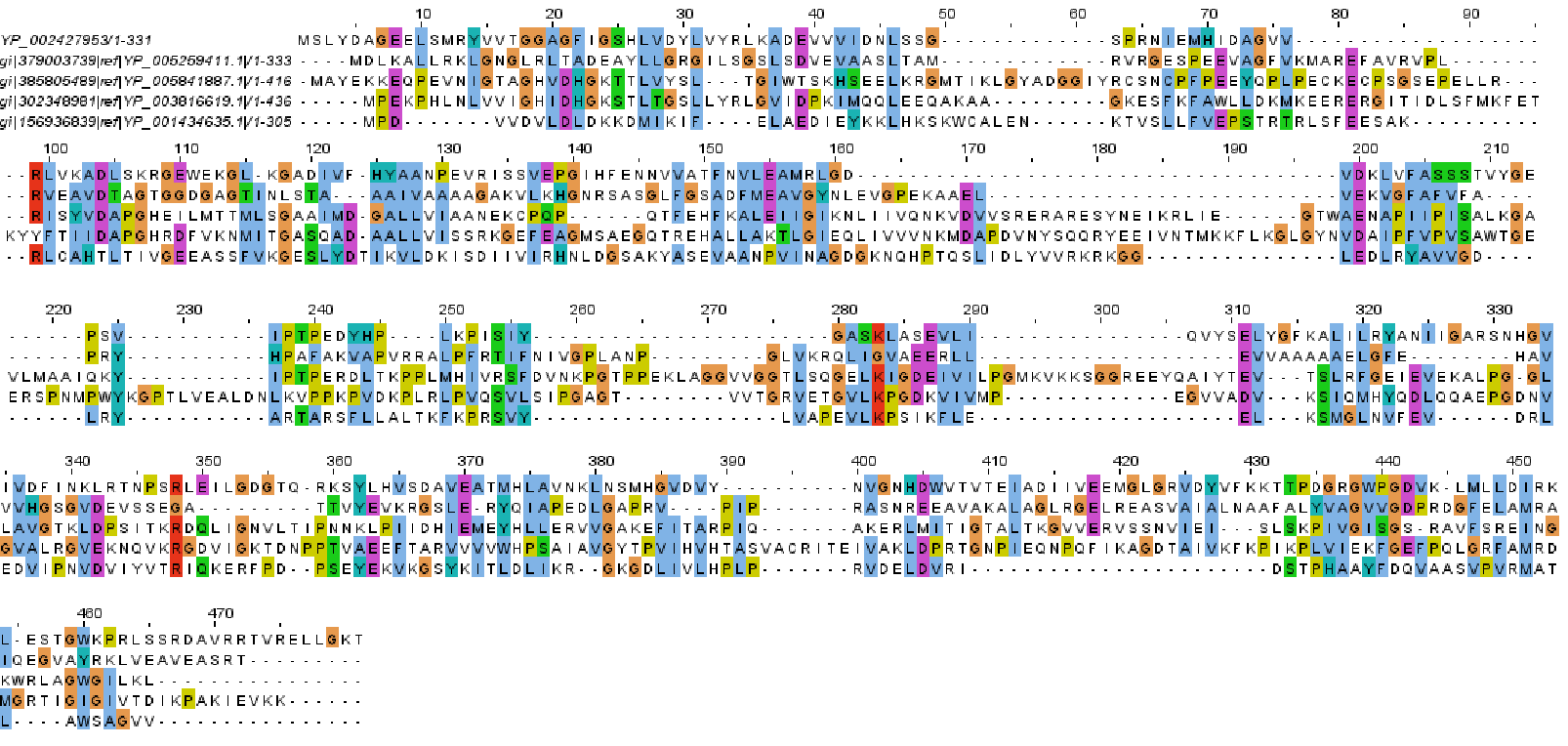
По выравниванию сразу видно, что белки непохожи и все совпадения, вероятнее всего, являются случайными.
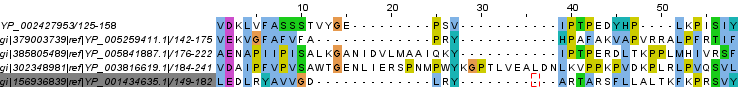
Самые лучшие блоки:
JavView project