Пространственное строение нуклеиновых кислот
Построение пространственных структур ДНК
С помощью средств пакета 3DNA были созданы три файла PDB, которые отражают структуры А,В и Z-форм ДНК. О различиях между этими формами будет подробно рассказано далее. Каждая молекула ДНК имеет длину 20 нуклеотидов и представляет собой 5 раз повторённый участок gatc. Одинаковая последовательность позволяет нам более объективно проводить сравнение структур.Работа со структурами нуклеиновых кислот средствами Jmol
- Визуализация различных групп атомов
Используя в качестве исходной структуры файл PDB для А-формы ДНК, приведу некоторые возможные примеры выделения групп атомов в структуре ДНК. Для начала продемонстрирую различные варианты того, как можно показать отдельные атомы. Так как мономером ДНК является один нуклеотид, то выделение всего участка молекулы есть выделение всех нуклеотидов.


Рис. 1 Выделение catoons. Показан условный остов молекулы, на условных связях отходят основания. Не окрашивается покраской cpk.
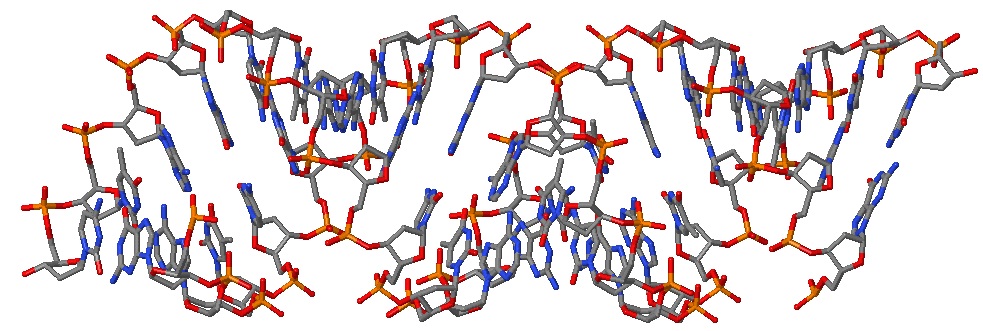
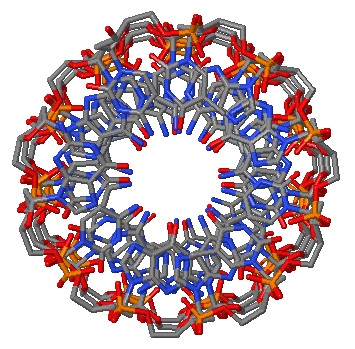
Рис. 2 Выделение wireframe. Показаны связи между атомами, но каждый из них представляет собой только точку. Окрашено по способу cpk - каждый тип атома в свой цвет.
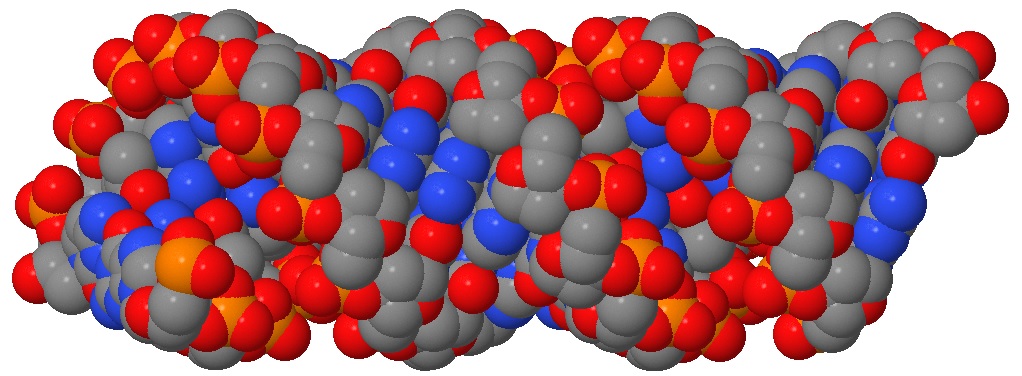
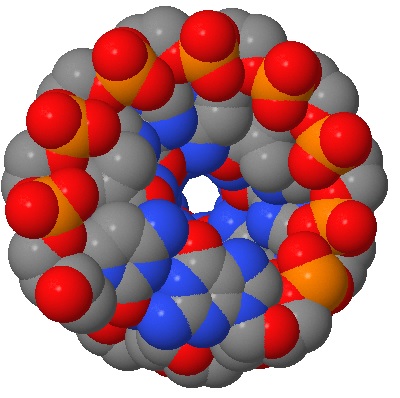
Рис. 3 Выделение cpk. Каждый атом представлен как объёмная единица со своим точны
м атомным радиусом.
Приведу несколько примеров выделения групп атомов:
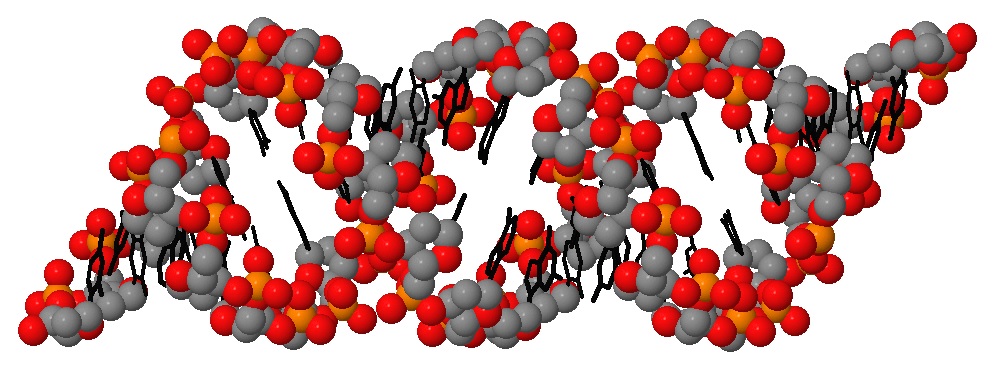
Рис. 4 Выделение cpk показывает сахаро-фосфатный остов молекулы, представленный остатками дезоксирибозы и фосфорной кислоты. Нуклеотиды показаны чёрным.
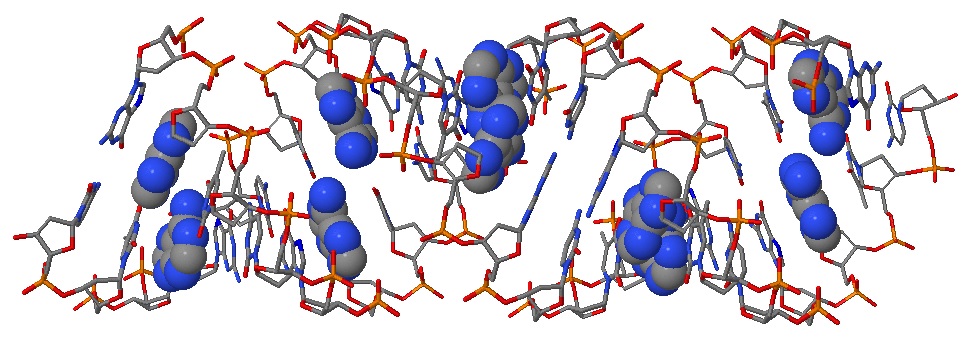
Рис. 5 Выделением cpk отмечены все остатки аденина - одного из двух пуриновых оснований, обнаруживаемых в ДНК.
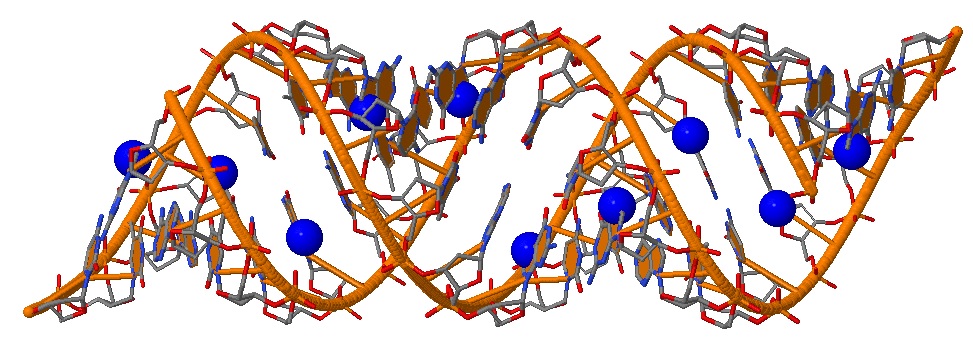
Рис. 6 Показаны атомы азота N7 во всех остатках гуанина, входящих в состав фрагмента ДНК.
- Для дальнейшей работы сайта PDB получены две структуры: одна из них содержит фрагмент ДНК, а другая РНК. Что же они представляют с биологической точки зрения?
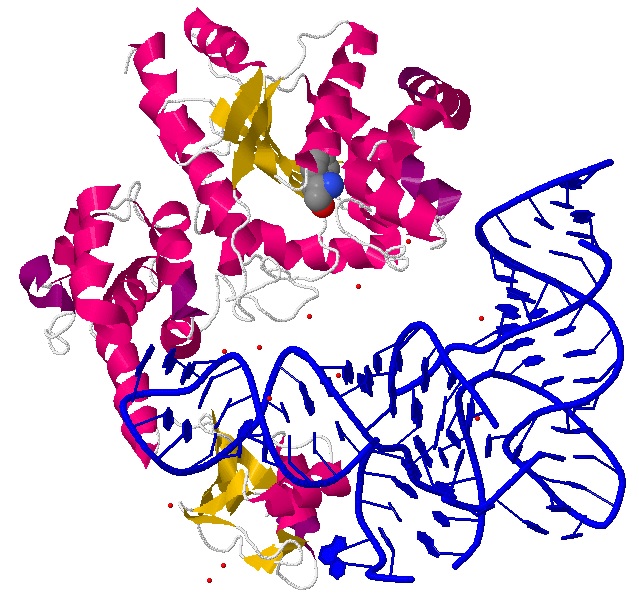

Рис. 7 Тирозил-тРНК-синтетаза из Thermus thermophilus в комплексе с тирозиновой тРНК, АТФ и тирозином.
Рис. 8 Белок из Trichomonas vaginalis, ответственный за связывание с инициаторами, в комплексе с иницаторным участком гена ферродоксина
- Проверим структуры ДНК и РНК на наличие разрывов различной природы. Для этого нуклеиновые кислоты визуализированы в проволочной модели (wireframe), затем просмотрены на неразрывность сахарофосфатного остова.
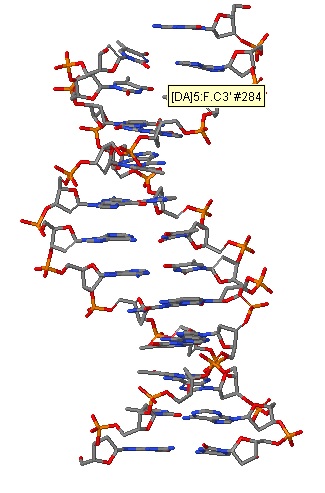
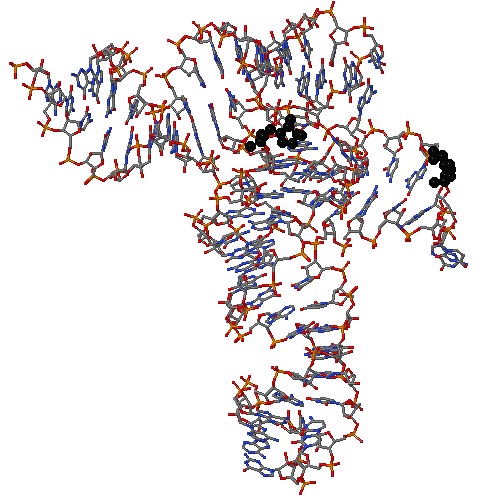
Рис. 9 В участке млекулы ДНК разрывов не содержится. Приведена проволочная модель участка.
Рис. 10 В транспортной РНК также разрывы не обнаружены. Правда, нашлись два места, где к рибозе не прикреплён никакой остаток азотистого основания. Такие сайты обозначены на рисунке чёрным.
Сравнение структур 3-х форм ДНК с помощью средств JMol
Вернёмся к работе с тремя сгенерированными участками ДНК, а не с файлами pdb, что мы делали только что. Интересно, что при формировании спиральной структуры ДНК появляются так называемые большая и малая бороздки (см. рис 11). 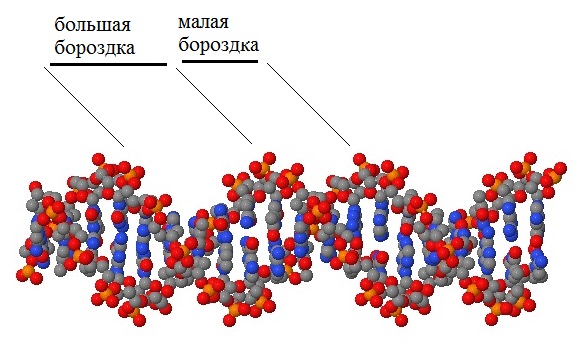 |
Рис. 11 Большая и малая бороздки в структуре фрагмента B-формы ДНК. |
Давайте попробуем понять какие атомы определённого азотистого основания обращены в малую, а какие в большую бороздку. В качестве примера будем использовать тимин, который мы рассматривали и в блоке "Химическое строение нуклеиновых кислот".
 |
Рис. 12 Распределение атомов тимина между большой и малой бороздками ДНК (B-форма). |
В нижеприведённой таблице представлены результаты измерений, проведённых на участках различных форм ДНК (A-, B- и Z-) с помощью средств измерения Jmol. Рисунки 13-15 иллюстрируют процесс измерения шага спирали как расстояния между элементами, совпадающими при взгляде с торца молекулы.
Основные спиральные характеристики форм ДНК |
A-форма |
B-форма |
Z-форма |
| Тип спирали (правая или левая) | Правая |
Правая |
Левая |
| Шаг спирали (A) | 28,03 |
33,75 |
43,5 |
| Число оснований на виток | 11 |
10 |
12 |
| Ширина большой бороздки (A) | 16,97 ([A]:6.A.P -[A]:38.B.P) |
17,21 ([G]:33.B.P - [G]:5.A.P) |
16,08 ([C]:38.B.P - [C]:5.A.P) |
| Ширина малой бороздки (A) | 7,98 ([A]:6.A.P - [G]:29.B.P) |
11,69 ([T]:15.A.P - [A]:30.B.P) |
7,2 ([G]:15.A.P - [G]:29.B.P) |
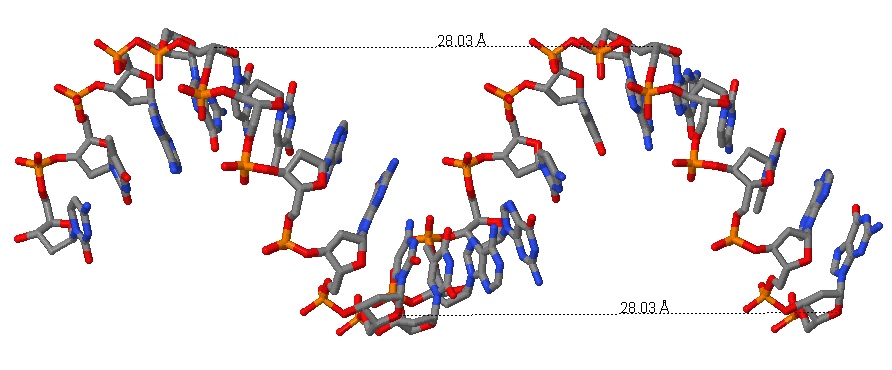 |
Рис. 13 Измерение шага спирали на молекуле ДНК в А-форме. показана одна из двух цепей. |
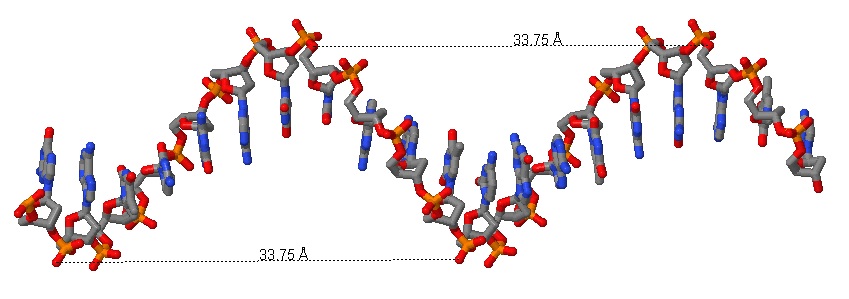 |
Рис. 14 Измерение шага спирали на молекуле ДНК в B-форме. показана одна из двух цепей. |
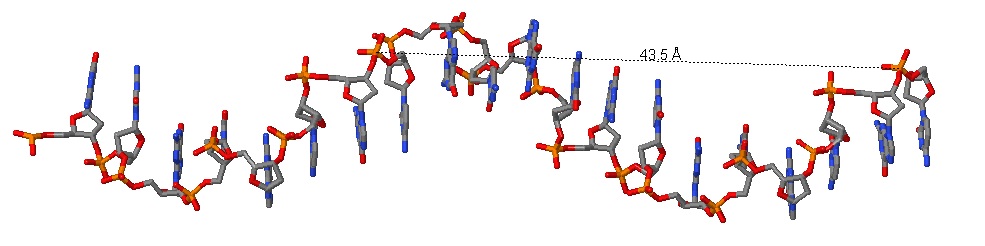 |
Рис. 15 Измерение шага спирали на молекуле ДНК в Z-форме. показана одна из двух цепей. интересно, что для таких измерений пришлось создать более длинный фрагмент (20 нуклеотидов), т.к. во фрагменте длинной в 10 не было полного шага спирали. |
 |
|
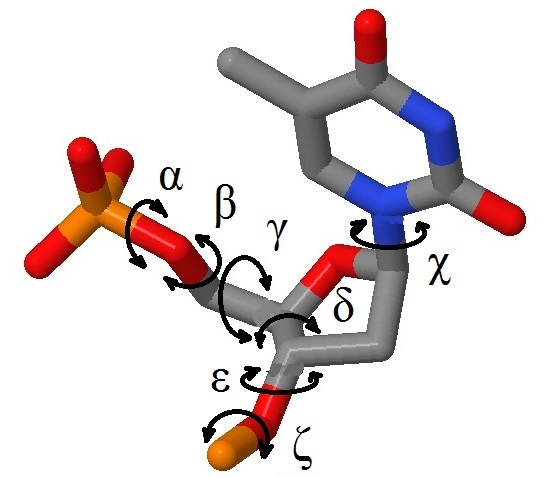
Рис. 16 Торсионные углы в тимидиловом нуклеотиде |
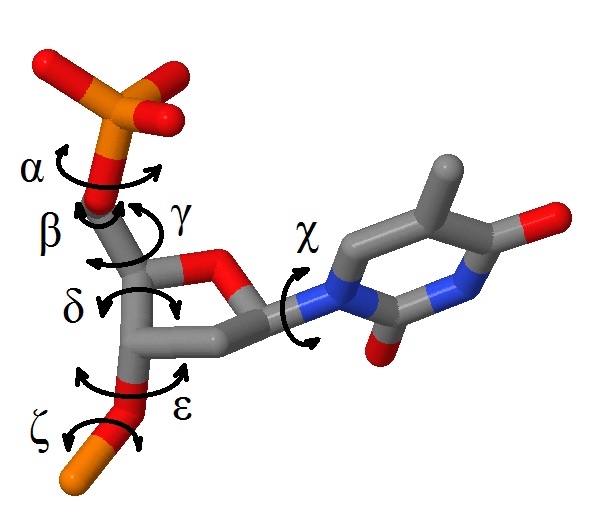
Рис. 17 Торсионные углы в тимидиловом нуклеотиде |
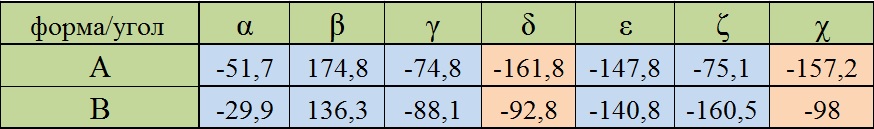 |
Рис. 18 Таблица результатов измерения торсионных углов в A и В формах ДНК. |
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
- Определение торионных углов в нуклеотидах
- Подготовим файл для дальнейшей обработки пакетом 3DNA. Для этого из него нужно убрать всё, что не касается нуклеиновых кислот, а также перевести в старый формат программой remediator --old.
- Далее используем конвейр из команд find_pair -t и analyze, первая из которых "готовит" файл .pdb для работы второй, которая, в свою очередь, проводит анализ структуры по многим параметрам. в том числе она позволяет определять торсионные углы.
- Из всех полученных в результате анализа файлов выбираем тот, что имеет расширение .out, в нём и находим информацию по торсионным углам.
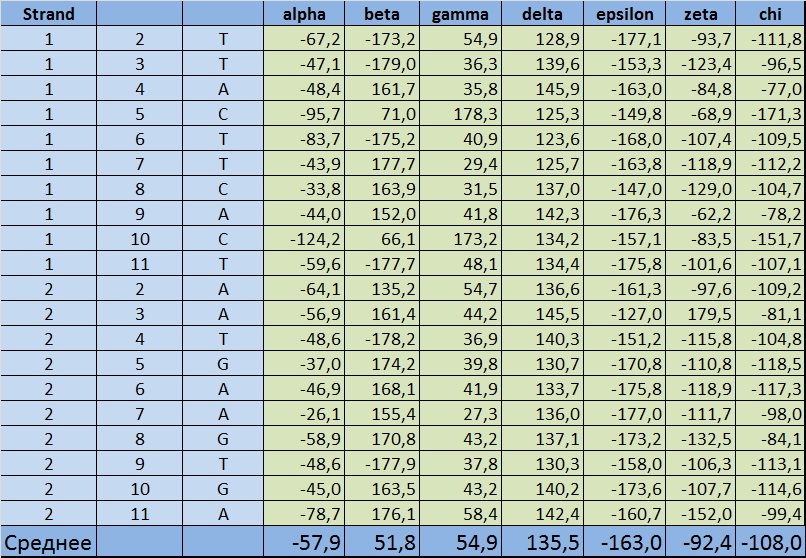
Рис. 19 Торсионные углы во фрагменте ДНК. Представлена информация по каждому изнуклеотидов кроме крйних, для которых очевидно невозможно определить два угла из восьми. приведён расчёт средних значений.
Анализируя представленную таблицу, замечаем, что наиболее деформированы (в плане наличия нестандартных торсионных углов) цитозиновые нуклеотиды в позициях 5 и 10 цепи 1. считаю необходимым сказать отдельно про угол бета, для которого среднее значение никак не отражает реальность, ведь по модулю среднее будет 158, всилу того, что угол принимает большие значения то со знаком "+", то со знаком "-".
Ссылка на аналогичную таблицу для РНКДанные по РНК α β γ δ ε ξ χ Average angle -40,28 10,02 67,20 88,83 -152,52 -70,94 -136,41
- Определение структуры водородных связей
Структура РНК весьма интересна именно с точки зрения того, как эта линейная молекула располагается в пространстве. Разумеется, водородные связи возникают и здесь, что приводит к формированию гораздо более сложных (по сравнению с ДНК) пространственных структур: шпилек, петель, стеблей, псевдоузлов и т.п. Попробуем и мы проанализировать нашу молекулу с такой точки зрения.
Используем команду find_pair -d XXXX_old.pdb XXXX_old.inp для того, чтобы описать вторичную структуру РНК. Получаем множество различных файлов. Для поиска выяснения расположения водородных связей используем файл bp_order.dat, в котором описаны координаты спиральных участков, среди которых мы и будем осуществлять поиск стеблей РНК уже визуально, используя средства Jmol.
Ниже приведены координаты найденных участков с водородными связями и их расположение в структуре тРНК:Акцепторный стебель: Стебель вариабельной петли: B:...1_:[..G]G-----C[..C]:..72_:B B:..45_:[..G]G-----C[..C]:..47G:B B:...2_:[..G]G-----C[..C]:..71_:B B:..46_:[..G]G-----C[..C]:..47F:B B:...3_:[..G]G-----C[..C]:..70_:B B:..47_:[..C]C-----G[..G]:..47E:B B:...4_:[..C]C-----G[..G]:..69_:B B:..47A:[..G]G-**--U[..U]:..47B:B B:...5_:[..A]A-----U[..U]:..68_:B B:...6_:[..G]G-----C[..C]:..67_:B T-стебель: B:...7_:[..G]G-----C[..C]:..66_:B B:..49_:[..G]G-----C[..C]:..65_:B B:..50_:[..C]C-----G[..G]:..64_:B D-стебель: B:..51_:[..U]U-----A[..A]:..63_:B B:..10_:[..C]C-----G[..G]:..25_:B B:..52_:[..G]G-----C[..C]:..62_:B B:..11_:[..C]C-----G[..G]:..24_:B B:..53_:[..G]G-----C[..C]:..61_:B B:..12_:[..C]C-----G[..G]:..23_:B Отдельные изолированные водородные связи: Антикодоновый стебель: B:..15_:[..G]G-**+-C[..C]:..48_:B B:..26_:[..G]G-*---U[..U]:..44_:B B:..19_:[..G]G-----C[..C]:..56_:B B:..27_:[..A]A-----U[..U]:..43_:B B:..20B:[..A]A-*---U[..U]:..47H:B B:..28_:[..C]C-----G[..G]:..42_:B B:..21_:[..A]A-*---U[..U]:..47I:B B:..29_:[..G]G-----C[..C]:..41_:B B:..30_:[..G]G-----C[..C]:..40_:B B:..31_:[..U]U-----A[..A]:..39_:B
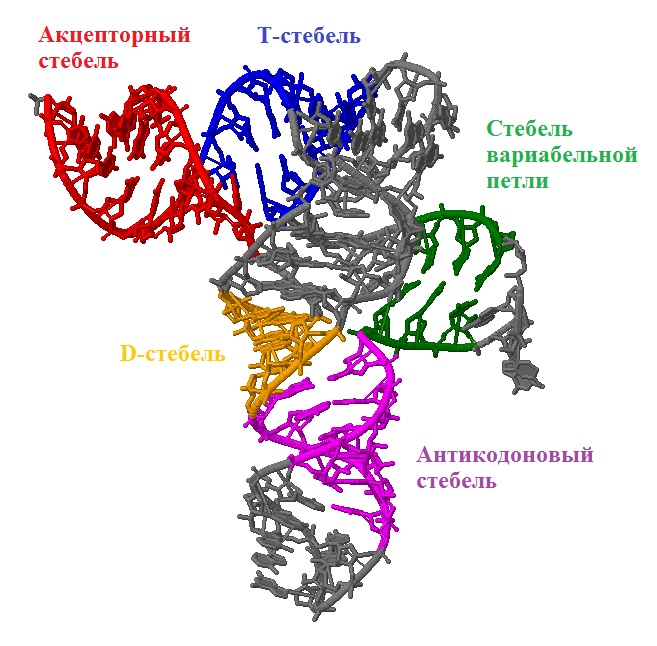
Рис. 20 Визуализация стеблей, т.е. участков, содержащих водородные связи в структуре тРНК.
-
Неканонические пары, изолированные связи:
Среди обнаруженных в структуре связей найдено 5 не Уотсон-Криковских пар. В списке всех водородных связей они могут быть найдены по содержанию символа "*". Отмечу отдельно, что много таких пар как раз в изолированных водородных связях.
Обнаружено 4 водородные связи, которые образуются между различными петлями и участвуют в оформлении третичной структуры молекулы - это изолированные связи, их список приведён выше, там же, где координаты для стеблей.
-
Поиск возможных стэкинг-взаимодействий:
В этом задании речь пойдёт о стэкинг-взаимодействиях: ситуации, когда происходит перекрывание пи-систем азотистых оснований, находящихся параллельно друг другу в структуре участка. Такие взаимодействия наряду с водородными связами придают дополнительную устойчивость нуклеиновым кислотам и не позволяют цепям расходиться.
Из файла dRNA_old.out получаем информацию о перекрывании пар в структуре РНК: ССЫЛКА
Рассмотрим две пары нуклеотидов с наиболее сильным перекрыванием (12,4 квадратного ангстрема), а следовательно с наиболее сильно проявляющимися стэкинг-взаимодействиями - это пары 3 и 4 (GC/GC). Кроме того выберем две пары нуклеотидов с минимальным ненулевым перекрыванием.step i1-i2 i1-j2 j1-i2 j1-j2 sum 3 GC/GC 6.59( 3.49) 0.00( 0.00) 0.00( 0.00) 5.81( 2.70) 12.40( 6.19) 16 UU/AA 0.00( 0.00) 0.00( 0.00) 1.80( 0.00) 0.00( 0.00) 1.80( 0.00)
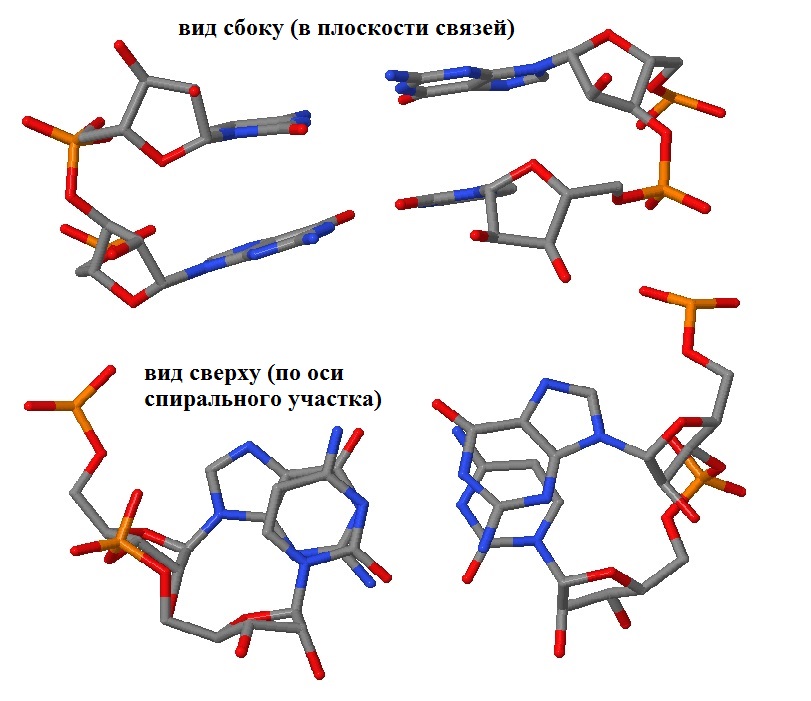
Рис. 21 Пары с наиболее сильными стэкинг-взаимодействиями, визуализация Jmol.
Кроме уже известных нам способов визуализации Jmol можно воспользоваться некоторыми командами пакета 3DNA:ex_str -3 stacking.pdb step3.pdb stack2img -cdolt step3.pdb step3.ps
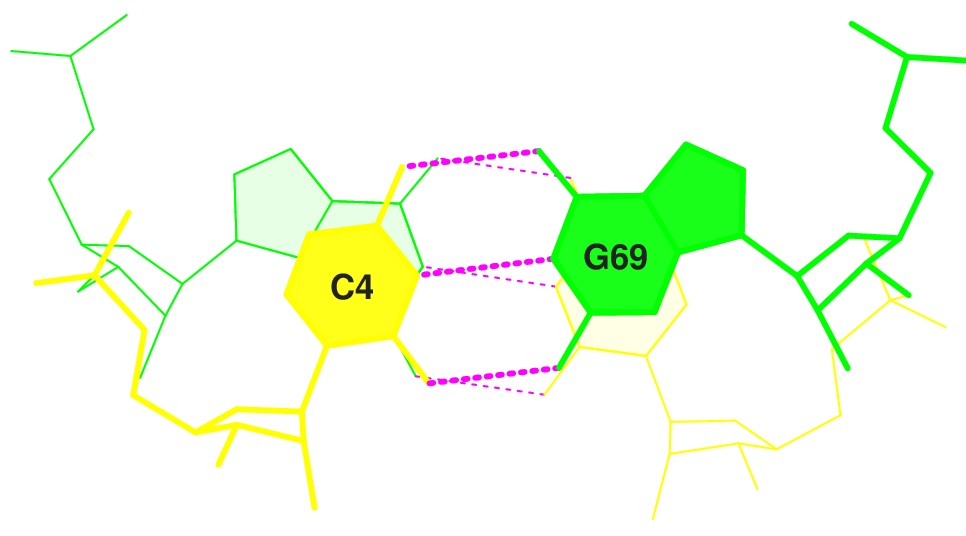
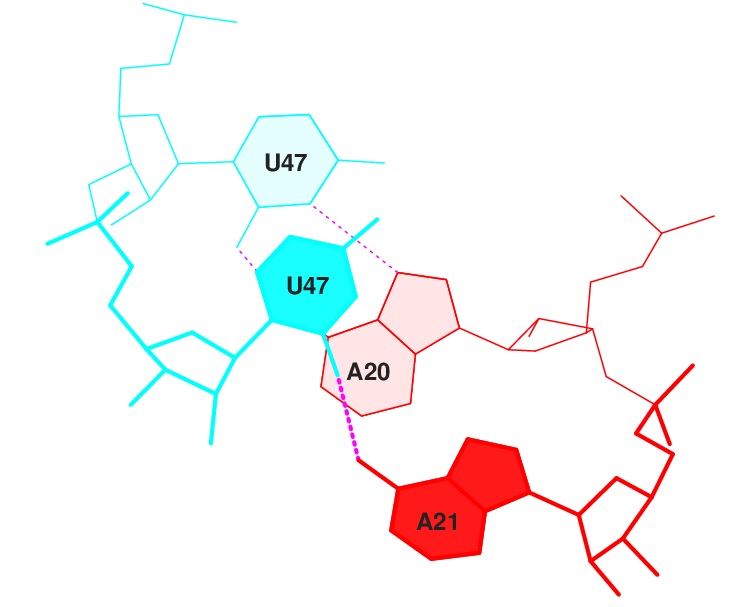
Рис. 22 Пары с наиболее сильными стэкинг-взаимодействиями, визуализация stack2img.
Рис. 23 Пары с наименее сильными стэкинг-взаимодействиями, визуализация stack2img.
Дата последнего обновления: 22.12.2013
© Dmitry Travin, 2013