Функциональная роль гена в подсистеме (GO, SEED).
Название и краткое текстовое описание системы секреции
Выданный мне белок с идентификатором Uniprot Q74XK1 является липопротеином аппарата системы секреции III у чумной палочки Yersinia pestis.Система секреции III (KEGG_Module)
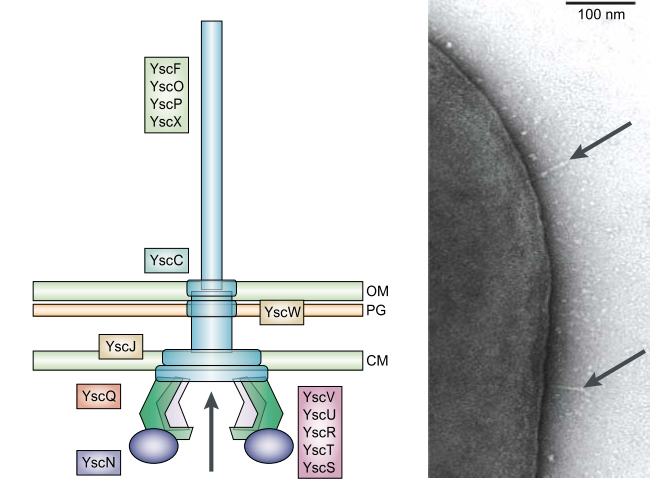
Система секреции типа III (см. Рис.1) присутствует исключительно в клетках грамотрицательных бактерий, многие из которых проявляют патогенность (Yersinia spp., Salmonella spp., Shigella spp., патогенные штаммы Escherichia coli, Pseudomonas aeruginosa, Vibrio parahaemolyticus, Bordetella spp., Chlamydia spp.). Система предназначена для импорта белков непосредственно в клетки эукариот (в отличие различных систем секреции, обеспечивающих выход белков в межклеточное пространство). С помощью TTSS (Type III Secretion System) белки преодолевают обе бактериальных мембраны, плазматическую мембрану эукариотической клетки, а в ряде случаев ещё и покровы последней (такие, как растительная клеточная стенка). Компоненты TTSS наиболее хорошо изучены у чумной палочки Yersinia pestis. Импортируемые таким образом белки называются эффекторами, так как они способны модулировать различные клеточные процессы в клетке эукариот.
 |
 |
| Рисунок 1. Схемы и микрофотография белкового комплекса системы секреции белков типа III (из PMID:12360191, PMID:15590783). | |
По материалам статей:
[1] G. R. Cornelis, The Yersinia Ysc-Yop ‘type III’ weaponry., Nat. Rev. Mol. Cell Biol., vol. 3, no. 10, pp. 742–52, Oct. 2002.
[2] P. Ghosh, “Process of protein transport by the type III secretion system.,” Microbiol. Mol. Biol. Rev., vol. 68, no. 4, pp. 771–95, Dec. 2004.
Белок имеет единственный релевантный термин GO:0009306 (cм. расшифровку в таблице 1).
Таблица 1. Термины GO, ассоциированные с белком Q74XK1
| GO-ID | Название | Онтология | Описание |
| GO:0009306 | Protein secretion (секреция белков) |
Биологический процесс | Контролируемый выход белков из клетки |
Функциональная роль белка в системе
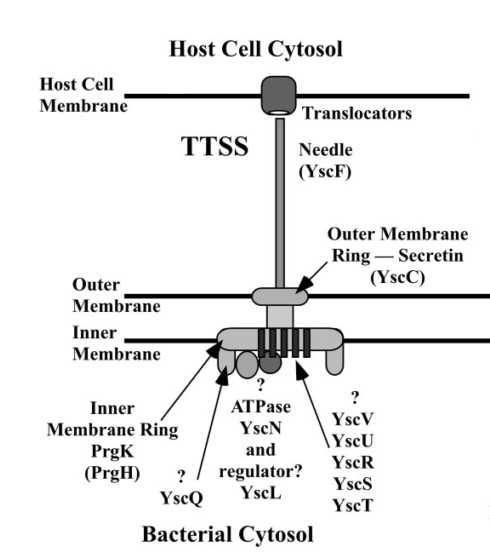
Для выяснения функциональной роли рассматриваемого белка была найдена страница KEGG для системы секреции III типа (KEGG_Module), оттуда я перешёл на страницу, где указаны все гены-ортологи для гена yscJ (KEGG_Ortology). Об ортологии гена рассматриваемого белка (ген flif1) с геном yscJ я узнал из статьи (PMID:15590783, таблица 1). В свою очередь, далее была найдена ссылка непосредственно на страницу гена flif1 KEGG_Gene_flif1). На рисунке 2 показана таблица, в которой приведены компоненты системы секреции III типа и схожие по последовательности белки, участвующие в построении жгутика различных бактерий (базальная часть жгутика, как известно, тоже является одной из систем экскреции).  |
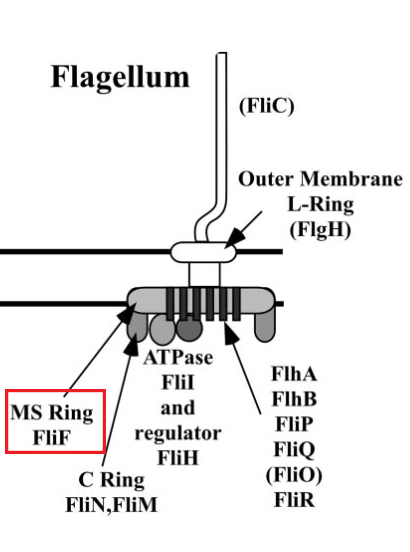 |
| Рисунок 2. Таблица соответствия между генами белков системы секреции III типа и генами белков базальной части бактериального жгутика (слева) и схема белков в системе экскреции, связанной со жгутиком. Выделены подписи, связанные с рассматриваемым белком (PMID:15590783). | |
Кроме того, из записи базы Uniprot рассматриваемого белка был осуществлён переход на страницу семейства Pfam: Family: YscJ_FliF (PF01514).
Вывод о функциональной роли белка: на основании данных статей (см. выше), страниц KEGG, SEED и Pfam было определено, что рассматриваемый белок локализуется во внутренней мембране грамотрицательных бактерий, является структурным и участвует в формировании связи между компонентами системы секреции во внутренней и внешней мембране (YscJ, HrcJ, EscJ, PscJ). Демонстрирует высокое сходство по последовательности с генами, формирующими M-кольцо бактериального жгутика.
Определение гомологов и сравнение окрестностей гена в различных организмах
При переходе на страницу гена в SEED произвели выборку гомологов, используя следующие параметры: 10 гомологов, E-value cutoff 1е-20, участок 15000 нуклеотидов. Окрестности гомологичных генов из выбранных организмов приведены на рисунке 3. 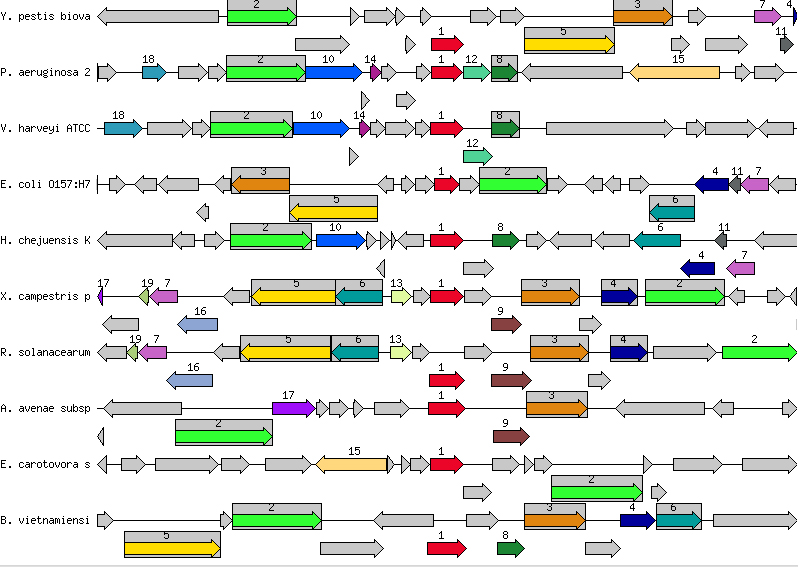 |
| Рисунок 3. Гены в окрестности рассматриваемого (обозначен как 1) для десяти гомологов |
Для выборки из десяти гомологов было получено выравнивание: ClustalW. Визуализация с помощью JalView приведена на рисунке 4, а также доступна по ссылке: Alignment_for_JalView.
 |
| Рисунок 4. Выравнивание последовательностей рассматриваемого белка из 10 отобранных гомологов. В подписи последовательностей обозначены виды бактерии, раскраска по консервативности с порогом 15%. |
Была получена таблица, содержащая информацию о найденных ортологах, на её основе была сформирована сводная таблица о генах, встреченных в окрестности рассматриваемого гена в выбранных 10-ти организмах. Таблицы доступны на листах "Исходные данные" и "Сводная" по ссылке: Table of homologs. Кроме того, приведён список всех белков, встречающихся в подсистеме, для каждого указана частота встречаемости на 10 геномов.
Выводы:
- Большинство генов, обрамляющих данный, в пределах 15000 нуклеотидов относятся, согласно базе данных SEED, к той же подсистеме, что и рассматриваемый ген. К наиболее часто ко-локолизуемым (более чем в половине случаев) генам относятся гены белков: YscC, YscL, YscN, YscS, YscT, YscD, YscR, YscU - компоненты системы секреции III типа (см. схему на Рис. 1).
- Сложно что-либо сказать о ко-локализации генов из других подсистем, так как для каждой из 9 отличных от нашей подсистем нашлось только по одному гену в каком-либо одном геноме (это может объясняться исключительно случайными факторами).
- Гены белка YscC (формирование поры во внешней мембране) и белков YscS, YscT, YscD, YscR, YscU (во внутренней мембране) колокализуются с рассматриваемым геном во многих отобранных геномах. Это косвенно подтверждает то, что функция изучаемого гена в подсистеме состоит в образовании мостика между компонентами секреторного комплекса во внутренней и внешней мембранах.
- Из рисунка 3 становится понятно, что в принципиальном большинстве случаев гены идут друг за другом "голова к хвосту", что, очевидно, значительно удобнее для совместной транскрипции и регуляции генов секреторной системы.
- С удалением от рассматриваемого гена падает ортологичность расположения генов, появляется большее число генов, которые не относятся к секреторной системе III типа (на основе визуальных наблюдений в SEED с участком в 30000 нуклеотидов).
Дата последнего обновления: 22.05.2014
© Dmitry Travin, 2014