Практикум 10
Гомологи ДНК-зависимой РНК-полимеразы
Для выполнения данного практикума я использовала последовательность DNA-directed RNA polymerase subunit beta(A0A0A7RBW6_9EURY), принадлежащую Natribaculum luteum. Последовательность в формате fasta была скачана с UniProt и загружена в BLAST, при этом в качестве банка данных был выбран Swiss-Prot. Был получен файл с текстовой выдачей программы:
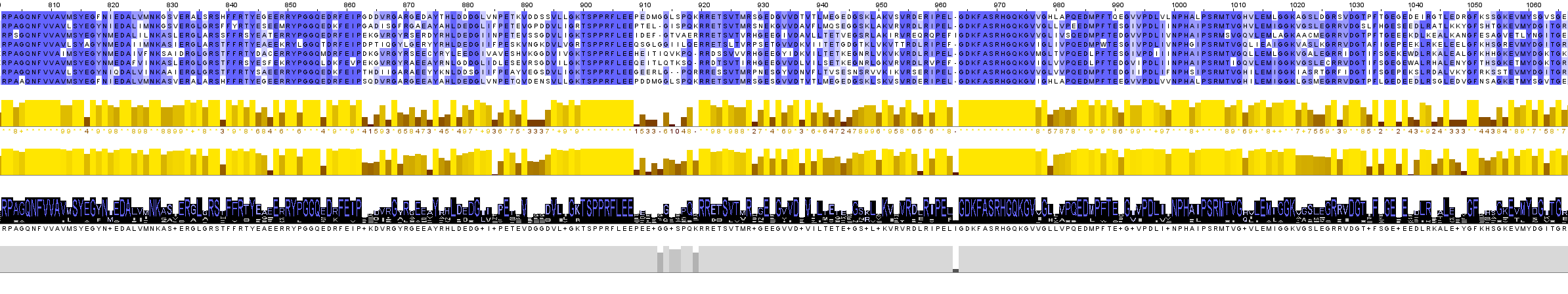
Далее из полученного с помощью BLAST списка гомологов к ДНК-зависимой РНК-полимеразе Natribaculum luteum были выбраны 6 последовательностей белка DNA-directed RNA polymerase subunit Rpo2C и subunit Rpo2 с E-value=0.0, принадлежащих разным видам: Halobacterium salinarum R1, Archaeoglobus fulgidus DSM 4304, Methanothermobacter thermautotrophicus str. Winter, Thermococcus celer, Methanocaldococcus jannaschii DSM 2661, Methanococcus vannielii SB, Thermoplasma acidophilum DSM 1728. Там же в Blast с ними было построено множественное выравнивание, которое я затем загрузила в Jalview. Получившееся выравнивание раскрасила по проценту идентичности. Так как некоторые белки очень короткие по сравнению с другими, программа добавила к ним длинные инделы. Но все равно все последовательности сдержат одинаковые консервативные блоки, поэтому я никакие из них за негомологичностью не удаляла.
Ссылка на выравнивание: Проект Jalview
Фрагмент множественного выравнивания, показывающий один из консервативных участков
Гомологи зрелого вирусного белка, вырезанного из полипротеина
В UniProt через расширенный поиск была найдена запись полипротеина P1234 - POLN_AURAV(ID:P1234, AC:Q86924), принадлежащего вирусу ауры(Aura virus)
В его записи Swiss-Prot в поле FT были найдены ключи CHAIN. Из низ я выбрала четвертый ключ, то есть фермент, блокирующий мРНК nsP1 (mRNA-capping enzyme nsP1) с координатами 1..539.
Информация о ферменте, блокирующем мРНК nsP1
Фермент катализирует две вирусспецифические реакции: метилтрансферазировние и гуанилилтрансферазиование nsP1. Блокирование мРНК необходимо, поскольку все вирусные РНК синтезируются в цитоплазме, а ферменты, блокирующие мРНК хозяина, ограничены ядром. Ферментативная реакция включает ковалентную связь между 7-метил-GMP и nsP1, тогда как эукариотические каппирующие ферменты образуют ковалентный комплекс только с GMP. Кэпирование nsP1 заключается в следующих реакциях: GTP сначала метилируется в 7-метил-GMP, а затем ковалентно связывается с nsP1 с образованием комплекса m7GMp-nsP1, из которого комплекс 7-метил-GMP переносится на мРНК для создания кэп-структуры. NsP1 необходим для инициации синтеза минус-цепочечных РНК. Вероятно, служит мембранным якорем для репликационного комплекса, состоящего из nsP1-nsP4. Пальмитоилированный nsP1 реконструирует цитоскелет клетки-хозяина и индуцирует образование структуры, подобной филоподию, на поверхности клетки-хозяина.
Вырезанная последовательность зрелого белка
Далее были проделаны те же действия, что в первом пункте домашнего задания.
Для множественного выравнивания помимо вырезанной последовательности mRNA-capping enzyme nsP1 Aura virus я выбрала Polyprotein P1234 следующих вирусов, чьи последовательности имели E-value 0.0:Ockelbo virus, Sindbis virus, Semliki Forest virus, Getah virus, Sagiyama virus, Ross river virus (STRAIN NB5092), Mayaro virus (strain Brazil). При редактировании выравнивания в Jalview были удалены все буквы находок, которые в выравнивании оказались правее последней буквы, выровненной с какой-либо буквой исходного зрелого белка, так как нам не нужно сравнивать полипротеины целиком.
Одна из последовательностей в выравнивании значительно короче других, но тем не менее все они имеют большое количество длинных консервативных блоков, так что можно сказать, что гомологичнсть последовательностей налицо.
Ссылка на выравнивание: Проект 2 в Jalview
Исследование зависимости E-value от объёма банка
При повторении последнего поиска в Blast, но уже с фильтром по вирусам, список находок не изменился. Однако у тех полипротеинов, у кого значение E-value отличалось от нулевого, поменялось это самое значение. Например, у полипротеина P1234 до добавления фильтра значение составляло 5e-125, а после - 2e-126. Путём сравнения значений E-value этой находки в двух поисках можно оценить долю вирусных белков в Swiss-Prot, для этого рассмотрим формулу для вычисления E-value.
E-value=mn·2^(–B), где:
m — длина исходной последовательности
n — размер базы данных
B — константа
Так как константа B и длина исходной последовательности m не меняются в нашем сравнении, то долю вирусных белков, то есть ее содержание в базе данных(n), определяем по отношению второго полученного значения E-value к первому. Путем бесхитросной работы с калькулятором получаем, что эта самая доля равна 0.04, или же 4%.
E-value – ожидаемое количество случайных находок с таким же и лучшим весом выравнивания, при поиске в той же базе данных, со случайным запросом той же длины и состава, с теми же параметрами на вычисление веса выравнивания. Так как E-value отражает математическое ожидание случайной величины, а Blast - это эвристический алгоритм, то полученное значение в 4% нельзя назвать абсолютно точным, в нем заложена значительная погрешность.