Подсказки по Jmol для начинающих
Подробное описание команд скриптового языка Jmol (на английском).
Обратите внимание: все команды, которые указаны ниже, можно как вводить по одной в командную строку Jmol, так и записать в отдельный файл и потом копировать-вставлять его содержимое в командную строку. Такой файл с командами называется скриптом. Рекомендую всегда, как только освоитесь с синтаксисом, писать скрипты, так как картинку часто бывает нужно немного исправить, а сделать это со скриптом куда проще, чем заново набивать все команды!
0) Загрузка и установка Jmol
Если на Вашем компьютере не стоит так называемая "Java-машина", то перед установкой Jmol, надо установить ее. Для этого выполните следующее:
Зайдите на официальный сайт Java.
- Перейдите на страницу загрузки JRE (Download JRE). Это Java Runtime Environment, то есть собственно Java-машина, нужная для запуска Java-приложений.
- Примите лицензионное соглашение (Accept License Agreement)
- Выберите для скачивания файл, соответствующий Вашей операционной системе (для 32-битной версии Windows выберите "Windows x86 Offline", для 64-битной версии - "Windows x64" и exe-файл, а не tar.gz).
- Запустите загруженный инсталлятор и следуйте указаниям мастера установки!
Для установки самого Jmol выполните следующее:
Зайдите на официальный сайт Jmol.
- В правом верхнем углу странички есть ссылка на страницу загрузки (Download).
- Разархивируйте zip-файл
Программа Jmol не требует специальной установки, так как является Java-приложением и может запускаться при наличии установленной на компьютере версии Java-машины (скорее всего, она уже есть у Вас на домашнем компьютере/ноутбуке, если нет - выполните действия выше). Для запуска программы используйте файл Jmol.jar в папке Jmol.
1) Базовые действия
Действие |
Результат |
Примечание |
Системные команды |
||
File ⇒ Open |
Открывает файл в формате PDB |
|
Ctrl + ЛКМ или ПКМ |
Вызов меню |
Консоль вызывается через меню (пункт Console) |
Движение изображения |
||
Alt + движение мыши или Shift + мышь влево-вправо |
Вращение в плоскости изображения |
|
Shift + мышь вверх-вниз или Колесико мыши |
Увеличение-уменьшение |
|
ЛКМ + Движение мыши |
Свободное вращение |
Вокруг центра |
Ctrl + ПКМ + движение мыши |
Перемещение в плоскости изображения |
|
2) Наборы атомов (SET) – то, с чем совершается большинство команд Jmol
Набор атомов |
Описание |
Комментарий |
none |
Ничего |
Пустое множество |
carbon |
Все атомы углерода |
|
nitrogen |
Все атомы азота |
|
oxygen |
Все атомы кислорода |
|
protein |
Все атомы белка |
|
DNA |
Все атомы, принадлежащие основаниям ДНК (A, C, G, T) |
|
RNA |
Все атомы, принадлежащие основаниям РНК (A, C, G, U). Также нуклеотидные с рибозой кофакторы. |
|
water |
Все атомы воды (по сути, кислороды) |
Синоним “HOH” |
ligand |
Не-белок, не-вода и не-нуклеотиды |
Чтобы выделить один конкретный лиганд, надо знать его название |
K, Na, Mg, Zn и т.п. |
Химические символы металлов узнаются |
|
*/25 |
Все атомы модели с номером 25 |
Модели (models) часто появляются в PDB-файлах, полученным методом ядерно-магнитного резонанса или в результатах трехмерных выравниваний. Также с помощью моделей иногда отмечают элементы биологически адекватной структуры ("biological assembly") в PDB. |
*S |
Все атомы, принадлежащие цепи, отмеченной “S” |
Цепи обычно используются, если в одном PDB-файле находится несколько молекул белка (например, это белковый комплекс, гомодимер, комплекс с ДНК и т.п.) |
*:2 |
Все атомы, принадлежащие цепи с названием 2 |
Используется если имя цепи – не буква, а число |
25 |
Все атомы, принадлежащие остатку с порядковым номером 25 |
Если несколько цепей имеют такой остаток, будут выделены все |
25-50 |
Все атомы, принадлежащие остаткам с порядковыми номерами от 25 до 50 |
|
25, 27, 29 |
Все атомы, принадлежащие остаткам с порядковыми номерами 25, 27 и 29 |
Аналогично записи 25 OR 27 OR 29 |
Asp21 |
Все остатки аспарагиновой кислоты с порядковым номером 21 |
|
*.CA |
Все Cα-атомы |
Если нужно выбрать атомы из одной цепи, используется синтаксис *А.CA |
*.N |
Все атомы азота главной цепи белка |
|
*.O |
Все атомы кислорода главной цепи белка |
|
*.C |
Углероды группы COOH главной цепи белка |
|
167:A |
Остаток с номером 167 в цепи A |
Аналогично записи 167 AND :A |
167:A.N |
Атом азота основной цепи остатка 167 из цепи A |
Аналогично записи 167 AND *.N AND :A |
167.N |
Атомы азота основной цепи всех остатков с номерами 167 (из всех цепей) |
Аналогично записи 167 AND *.N |
:A.N |
Атомы азота основной цепи всех остатков из цепи :A |
|
within (5.0, SET) |
Все атомы, которые находятся на расстоянии 5 Å от любого атома из SET |
Расстояние можно задавать любое, но обязательно использовать точку, а не запятую, при отделении дробной части |
within(GROUP, SET) |
Все атомы, входящие в группу с заданным набором атомов. Например, в одну группу входят атомы одного аминокислотного остатка |
Часто используется, когда SET в свою очередь тоже определен через within (например, чтобы получить изображение остатков, некоторые атомы которых взаимодействуют с заданным множеством) |
helix |
α-спиральные структуры |
Определяются в PDB-файле, т.е. могут не быть заданы |
sheet, sheets |
β-листы |
Определяются в PDB-файле, т.е. могут не быть заданы |
3) Основные команды визуализации для наборов атомов
Команда |
Описание |
Пример использования |
Комментарий |
Выделение |
|||
select SET |
Выбрать набор атомов на основании простых команд синтаксиса и логических операторов AND, OR, NOT |
|
|
restrict SET |
Аналогично команде select, но все кроме этого набора будет убрано с экрана |
|
|
Окрашивание |
|||
color COLOR |
Окрашивает выделенный в настоящее время набор атомов в выбранный цвет |
|
Стандартные английские названия цветов можно сразу использовать: pink, yellow, blue, red, magenta, violet, purple, cyan, orange, green, white, black |
color cpk |
Атомы красятся в соответствии с химической природой. Например, атомы углерода будут серые, азота голубые, кислорода красными, фосфора оранжевыми и т.п. |
|
Можно использовать и другую окраску, но не рекомендуется изменять базовый цвет азота и кислорода, так как по умолчанию обычно считается, что отрицательный заряд красный, а положительный – синий. |
color chain |
Все атомы будут покрашены в соответствии с принадлежностью к той или иной цепи |
|
Очень удобно, чтобы быстро увидеть, где какая цепь в мультиферментном комплексе |
color structure |
Цвета устанавливаются в соответствии со вторичной структурой |
|
|
color group |
Окрашивает в цвета обратной радуги (переход от фиолетового к красному) выделенный объект (каждая цепь красится отдельно) |
|
Лучше использовать отдельно для цепей белка и ДНК. N-конец будет синим, C-конец - красным |
color translucent VALUE |
Устанавливает меру прозрачности выделенного множества (0 - непрозрачное, 0.99 - самое прозрачное) |
|
|
background COLOR |
Устанавливает цвет фона |
|
Эта команда глобальная, то есть применяется не к выделенному сету, а вообще |
Отображение атомов |
|||
cpk VALUE |
Показывает все выбранные атомы в виде сфер |
|
Величина VALUE определяет размер атомов. Если ее не задать, каждый атом будет иметь свой Ван-дер-Ваальсовый радиус. Параметр "off" отключает это представление |
wireframe VALUE |
Показывает все химические связи в выбранном наборе атомов |
|
Величина VALUE определяет толщину линий. Если ее не задать, толщина будет минимальной. Параметр "off" отключает это представление |
backbone VALUE |
Рисует линии между всеми Cα-атомами (внимание, это не химические связи) |
|
См. описание для wireframe |
ribbons VALUE |
Остов белка будет представлен в виде ленточной модели. Подходит для рассмотрения топологии |
|
VALUE точно так же определяет толщину линий, но часто его опускают, так как дефолтная толщина выглядит лучше всего |
cartoons VALUE |
Еще один вариант "симпатичного" модельного изображения белка |
|
VALUE точно так же определяет толщину линий, но часто его опускают, так как дефолтная толщина выглядит лучше всего |
Добавление подписей к выбранному множеству атомов |
|||
label PARAMETER |
Каждый из выделенных атомов будет подписан в соответствии с параметром, поданным на вход команде (см. ниже подробности) |
|
Иногда бывает удобнее добавить подпись на уже готовый рисунок вручную |
PARAMETER |
Результат |
||
%a |
Условное имя атома (N, O, CA, CB и т.п.) |
||
%c |
К какой цепи принадлежит атом |
||
%e |
Элемент, к которому принадлежит атом (N, O, C, P, S и т.п.) |
||
%i |
Порядковый номер атома (не остатка) |
||
%m |
Однобуквенный код аминокислоты, к которой принадлежит атом |
||
%n |
Трехбуквенный код аминокислоты, к которой принадлежит атом |
||
%r |
Номер аминокислотного остатка, к которому принадлежит атом |
||
ON |
Включить подписи |
||
OFF |
Выключить подписи |
||
<любая строка> |
Подписать атом этой строкой |
||
hover PARAMETER |
Работает аналогично label, но выбранные подписи будут показываться не всегда, а только при наведении мыши на атом |
|
|
set fontsize VALUE |
Задает размер шрифта для подписей |
|
Размер по умолчанию – 8 |
set labeloffset X Y |
Задает отступ подписи от атома |
|
|
Создание собственных имен для наборов |
|||
define NAME SET |
Создает соответствие между выбранным именем NAME и набором атомов SET |
|
Обычно используется если нужно определить как единый элемент сложную совокупность атомов |
Разное (обычно используется при написании скриптов) |
|||
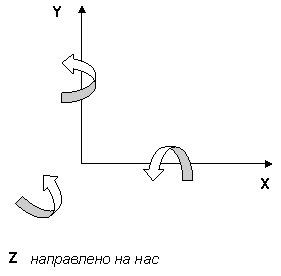
rotate AXIS ANGLE |
Вращает молекулу вдоль указанной оси AXIS на угол ANGLE (угол от -180? до 180?) |
|
Названия осей – X, Y и Z. Их расположение относительно экрана приведено на рисунке ниже |
center SET |
Указывает центр, относительно которого будет происходить вращение. |
|
Если SET указывает не на конкретный атом, а на любое множество атомов (например, цепь) – будет выбрана точка с усредненными по осям координатами (очень грубо – "центр масс") |
echo WHAT |
Выписывает выражение WHAT в консоль |
|
|
#comment/whatever |
Строчка после "#" игнорируется; можно использовать ее в качестве комментария к скрипту |
|
|
script PATH |
Вызывает скрипт с именем PATH |
script myscript.txt |
Должен лежать в директории с Jmol или нужно указывать полный путь |
|
Рисунок 1. Оси координат. |
4) Визуализация водородных связей
Команда |
Описание |
Пример использования |
Комментарий |
По умолчанию, в Jmol водородные связи вычисляются только между остовными азотами и кислородами (в белках) и комплементарными основаниями (в ДНК/РНК) |
|||
Для того, чтобы вычислить ВСЕ водородные связи, надо предварительно выполнить команду set hbondsRASMOL FALSE |
|||
calculate hbonds |
Вычислить пары атомов, связанных водородными связями |
|
Вычисляет и показывает |
calculate hbonds structure |
Применить другой метод (dssp)- вычисления водородных связей |
|
|
hbonds off или hbonds on |
Скрыть/показать водородные связи |
|
Результат - только для выбранного множества |
hbonds <число> |
Установить толщину черточек, изображающих водородную связь |
hbonds 50 |
толщина 50 у.е. |
|
|
hbonds 1.0 |
толщина 1 ангстрем |
color hbonds COLOR |
Изобразить водородные связи цветом COLOR |
color hbonds green |
|
5) Всякие сложные штуки!!!
Команда |
Описание |
Пример использования |
Комментарий |
load |
Загрузить координатный файл |
load "D:\\Bioinformatics\\udavdasha\\my.pdb" |
Не забывайте защищать слэши! Если писать команду без полного пути, файл my.pdb будет искаться в той папке, где стоит Jmol |
load append |
Загрузить координатный файл |
load append "D:\\Bioinformatics\\udavdasha\\my_second.pdb" |
Второй файл будет загружен в интерфейс модели (model) с номером 2 |
В Jmol можно применять к выбранному множеству матрицы поворота и векторы трансляции! Их, например, выдаст программа структурного выравнивания (например, DALI). |
|||
Матрица поворота: r = [[ 0.897551, -0.320665, -0.302617 ], [-0.405017, -0.328377, -0.853305], [0.174252, 0.888450, -0.424610 ]] |
|||
Преобразовать ее |
rq = quaternion(r) |
|
|
Задать вектор трансляции |
t = { 42.052868, 72.133873, -14.334292 } |
||
Повернуть |
rotateSelected MOLECULAR @rq |
||
Транслировать |
translateSelected @t |
||