Практикум 1. Филогенетическое дерево.
Цель занятия: поработать с филогенетическим деревом нескольких бактерий. Позже вы будете сравнивать это дерево с реконструкциями филогении тех же бактерий по последовательностям их белков. Отчёт по занятию должен появиться на сайте к утру дня следующего занятия.
Из нижеследующего списка протеобактерий выберите 7–8 названий.
Название |
Мнемоника |
Acidiphilium cryptum |
ACICJ |
Agrobacterium fabrum |
AGRFC |
Aromatoleum aromaticum |
AROAE |
Bartonella henselae |
BARHE |
Bordetella pertussis |
BORPE |
Brucella suis |
BRUSU |
Burkholderia cenocepacia |
BURCA |
Escherichia coli |
ECOLI |
Haemophilus influenzae |
HAEIN |
Neisseria meningitidis |
NEIMA |
Paracoccus denitrificans |
PARDP |
Pasteurella multocida |
PASMU |
Polynucleobacter asymbioticus |
POLAQ |
Proteus mirabilis |
PROMH |
Pseudomonas aeruginosa |
PSEAE |
Pseudomonas mendocina |
PSEMY |
Rhizobium meliloti |
RHIME |
Roseobacter denitrificans |
ROSDO |
Saccharophagus degradans |
SACD2 |
Serratia proteamaculans |
SERP5 |
Shewanella denitrificans |
SHEDO |
Thiobacillus denitrificans |
THIDA |
Yersinia pestis |
YERPE |
Руководствуясь приведённым ниже филогенетическим деревом, нарисуйте дерево выбранных вами организмов (для наглядности будем везде использовать мнемоники организмов вместо их полных видовых названий):
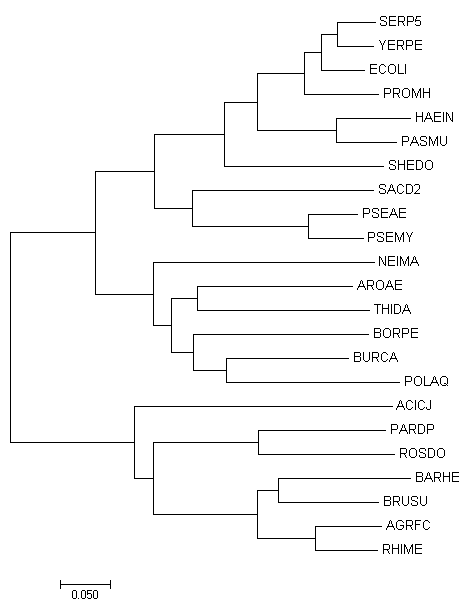
Приведите в отчёте:
а) скобочную формулу дерева;
б) изображение дерева;
в) список нетривиальных ветвей как разбиений множества листьев.
Напоминание: мы называем ветвь нетривиальной, если она разбивает множество листьев на подмножества, в каждом из которых более одного элемента.
В представлениях дерева удобно пользоваться краткими мнемоническими обозначениями видов (правая колонка таблицы). Но если вы будете их использовать, в отчёте обязательно должна присутствовать их расшифровка, то есть таблица отобранных видов с указанием мнемонического обозначения каждого вида.
Совет по созданию изображения. Можно, конечно, нарисовать дерево в каком-нибудь графическом редакторе. Но рисунок получится красивее, а вы сэкономите усилия, если поступите следующим образом. Нарисуйте дерево ручкой на бумаге. Затем создайте файл со скобочной формулой (имя файла должно иметь расширение .tre) и откройте его программой MEGA.
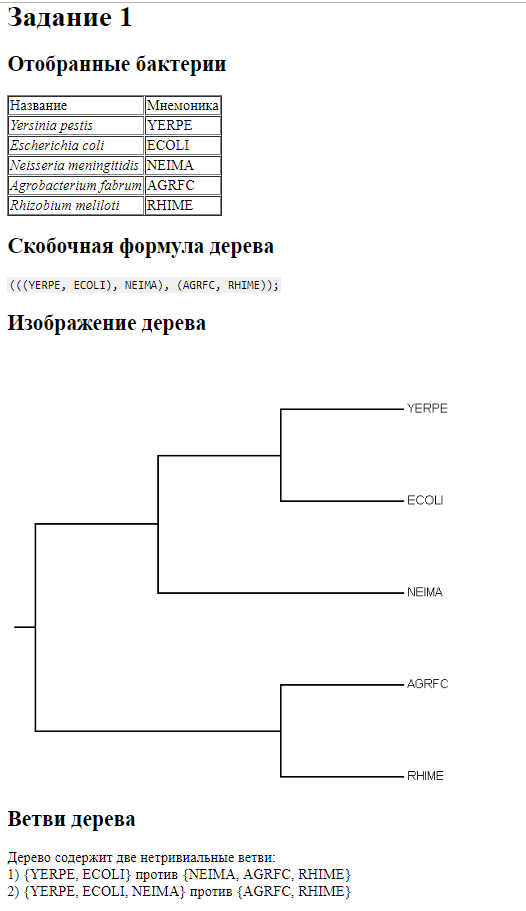
Пример отчёта
(здесь пять видов, у вас же их должно быть не менее семи).
2* Дополнительное задание от AAl
Это задание на умение написать короткий понятный текст на русском языке
Выполнение задания не обязательно
Задание проверяется и оценивается баллами. "Мёртвая черта":))) - начало блока 2, но лучше выполнить до 19 февраля.
- Найдите статью, в которой обсуждается филогения семейства белков.
- Используйте белки, с которыми вы раньше или сейчас имели дело. Но можно любые, ограничений нет.
- Используйте Pubmed или Google Scholar для поиска статьи. Желательно чтобы статья была написана на английском или русском языках.
- Важно, чтобы в статье обсуждалась филогения, но могут быть и другие результаты.
- Задание — только про филогению!
- Опишите коротко (или очень коротко) по-русски, каков результат анализа филогении.
- Ваш текст может ссылаться на данные из статьи — рисунки, таблицу, сопроводительные материалы
В отчёте приведите
- Ссылку на статью в таком виде, чтобы проверяющий мог её найти. Минимум-миниморум - первый автор, год, журнал; или первый автор, год, название. Если есть — приведите веб-адрес, по которому доступен полный текст.
- Название семейства белков, для которого реконструирована филогения. Желательно — что делают белки.
- Ссылка на рисунок с деревом в статье или копия этого рисунка с подписью в отчете.
- Несколько фраз о том, каков результат или вывод авторов по результатам реконструкции филогении.
Пример отчёта
1. Статья Wei Liang et al., "KlcAHS genes are ubiquitous in clinical, blaKPC-2-positive, Klebsiella pneumoniae isolates", 2019, Infection, Genetics and Evolution, https://www.sciencedirect.com/science/article/pii/S1567134818306245?via%3Dihub
2. В статье реконструируется филогения семейства белков KlcA из патогенной бактерии Klebsiella pneumoniae. Белки KlcA являются анти-рестрикционными белками. Это значит, что они ингибируют эндонуклеазы рестрикции, которые расщепляют чужеродную ДНК (фагов или плазмид)
3. Авторы построили филогенетическое дерево неизбыточной выборки представителей KlcA, см. Fig.2. Неизбыточная выборка значит, что из каждой группы высокосходных последовательностей берётся одна.
4. В статье изучаются белки KlcAHS, закодированные в некоторых плазмидах, выделенных из клинических изолятов K. pneumoniae. Эти белки принадлежат семейству белков KlcA. Филогения KlcA реконструирована для выяснения эволюционных взаимоотношений белков семейства KlcA. Авторы выводят, что интересующие их белки KlcAHS принадлежат одной кладе, удаленной от остальных представителей семейства. На рисунке эта клада изображена листом №16, за которым скрыты 20 высокосходных KlcAHS.
КОНЕЦ
Замечания по результатом выполнения задания.
Статья трудно написана. Из аннотации я догадался, что KlcAHS — это те белки анти-рестрикции, которые найдены на плазмидах K. pneumoniae, несущих ген blaKPC-2 бета-лактамазы.
Описание получилось длинное. Взял первую попавшуюся статью с рисунком филогенетического дерева. Оказалось, что она написана плохо. Потратил час чтобы разобраться что к чему. Студентам советую не зацикливаться на плохо написанной статье, а найти другую, более понятную!
Для ликбеза. Белки KlcA (как и другие, им гомологичные, с невысоким сходством последовательностей) мимикрируют под ДНК по форме и по распределению положительных зарядов на поверхности! Эндонуклеазы рестрикции, вместо расщепления чужеродной ДНК, связываются с этими белками и тем самым ингибируются. Поэтому плазмида с KlcA легко переносится в другие штаммы Klebsiella pneumoniae.


 2025
2025 2024
2024 2023
2023 2022
2022 2021
2021 2020
2020
 2018
2018 2017
2017