Построение дерева по нуклеотидным последовательностям
В данном задании требовалось построить филогенетическое дерево 8 представителей протеобактерий, используя нуклеотидную последовательность РНК малой субъединицы рибосомы (16S rRNA). Для работы были взяты следующие протеобактерии: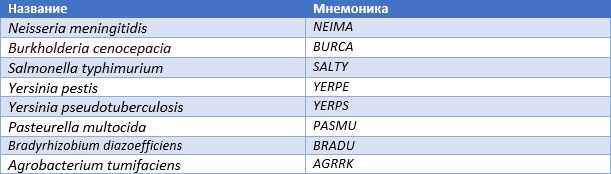
Таблица 1 Выранные мною протеобактерии
Последовательности 16S rRNA бактерий нужных видов были взяты из файлов с расширением .frn в записи, принадлежащей хромосоме каждой из бактерий, на сайте NCBI.
Ссылка на fasta-файл с последовательностями всех требуемых 16S rRNA: 16S rRNA
Я открыла последовательности в JalView и выровняла программой Muscle.
Выравнивание в fasta-формате я импортировала в программу MEGA, где реконструировала дерево, воспользовавшись методом Neighbor-Joining.
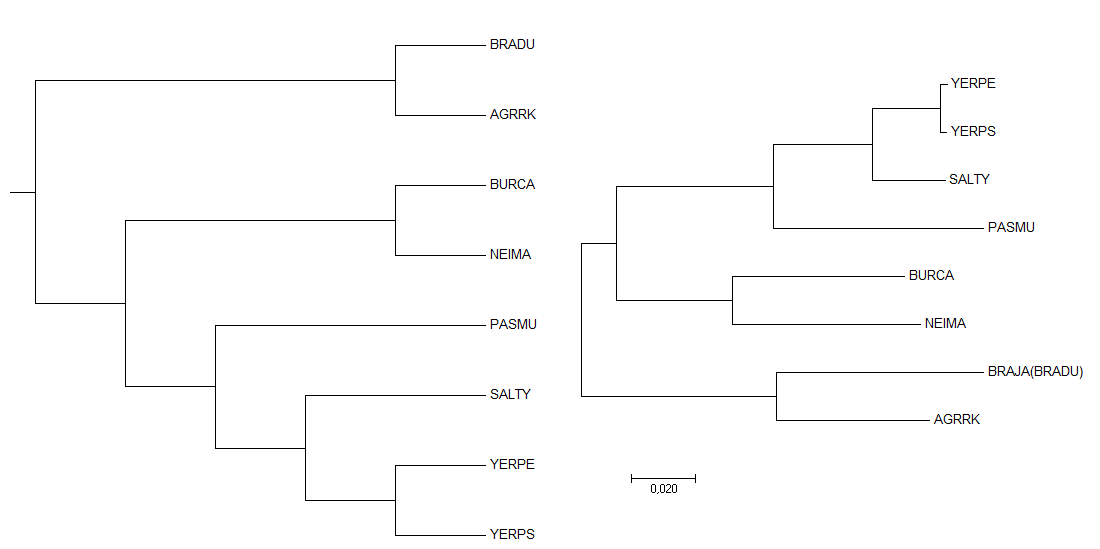
Рисунок 1 Филогенетическое дерево последовательностей 16S rRNA выбранных протеобактерий (справа); реконструировано методом Neighbor-Joining в программе MEGA. Исходное дерево слева.
Дерево, построенное по последовательностям 16S rRNA, полностью совпадает с исходным, в отличие от деревьев, построенных по последовательности белка пептидил-тРНК гидролазы.
В качестве напоминания представлено одно из деревьев, построенных по последовательности белка, реконструированное методом Neighbor-Joining и укорененное в среднюю точку.
Оно отличается от правильного тем, что в нем листья BURCA и NEIMA не составляют отдельную кладу.
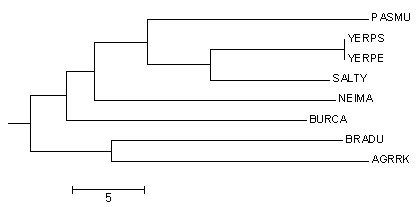
Рисунок 2 Филогенетическое дерево последовательностей белка пептидил-тРНК гидролазы выбранных протеобактерий; реконструировано методом Neighbor-Joining в программе MEGA и укоренено в среднюю точку программой retree пакета PHYLIP.
Таким образом, дерево, построенное по последовательностям РНК получилось правильным в отличие от дерева, построенного по последовательностям белка.
Построение и анализ дерева, содержащего паралоги
В данном задании было необходимо построить и проанализировать дерево гомологов белка CLPX_ECOLI из выбранных бактерий.
Чтобы найти гомологов, я взяла из файлы из директории P:\y14\term4\Proteomes, содержащие полные протеомы нужных мне бактерий, скачанные из базы UniProt, и командой "cat file1 >> file2" сложила их в один файл.
Затем я произвела поиск гомологов программой blastp с входной последовательностью белка CLPX_ECOLI по базе данных - файлу с нужными протеомами. Порог по E-value был взят 0.001.
В выдаче всего 34 находки, из них 14 - по 2 находки, принадлежащие белку HSLU, на каждую из 7 бактерий
Остальные находки представлены в одном экземпляре для каждого белка.
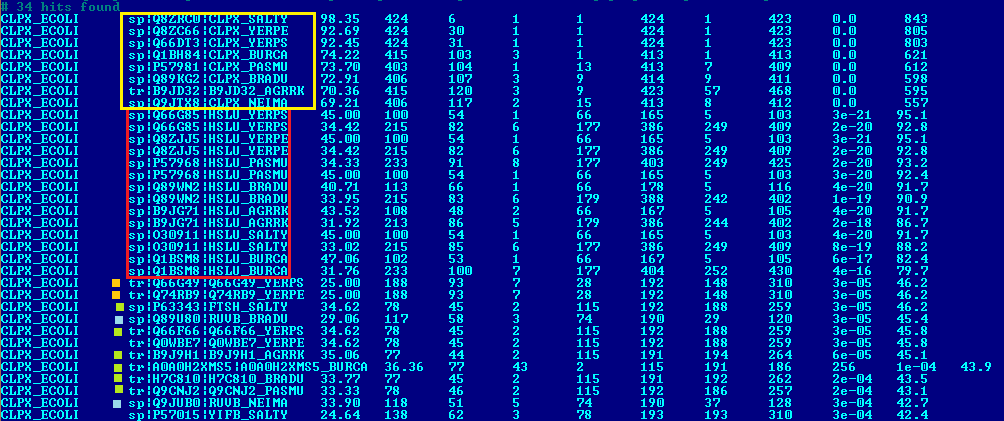
Рисунок 3 Выдача программы blastp
Затем последовательности были выровнены программой Muscle, по выравниванию было реконструировано дерево методом Neighbor-Joining в программе MEGA.
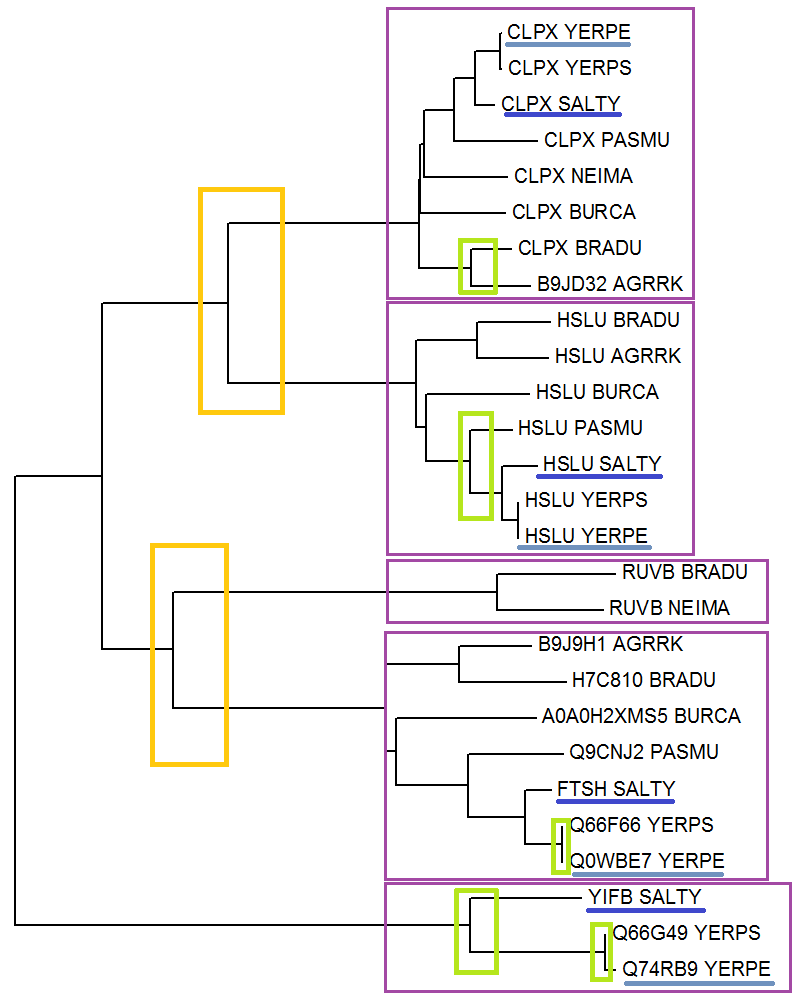
Рисунок 4 Филогенетическое дерево последовательностей гомологов белка CLPX_ECOLI; реконструировано методом Neighbor-Joining в программе MEGA.
На полученном дереве указаны следующие эволюционные события:
два белка выполняют разные функции и просто являются разными, а присутствуют у всех/почти всех видов: белки CLPX и HSLU представляют собой разные субъединицы АТФ-зависимой протеазы и оба встречаются почти у всех видов (нет белка HSLU у вида NEIMA); о дупликации, приведшей к появлению белков RUVB (хеликаза) и FtsH (АТФ-зависимой цинк металлопротеазе - разные мнемоники), свидетельствует то, что у BRADU есть оба белка