В рамках выполнения данного практикума я продолжила работать с белком lpxA (PDB ID: 2aq9). Одна субъединица белка показана на рис.1 и покрашена в соответствие со вторичными структурами.
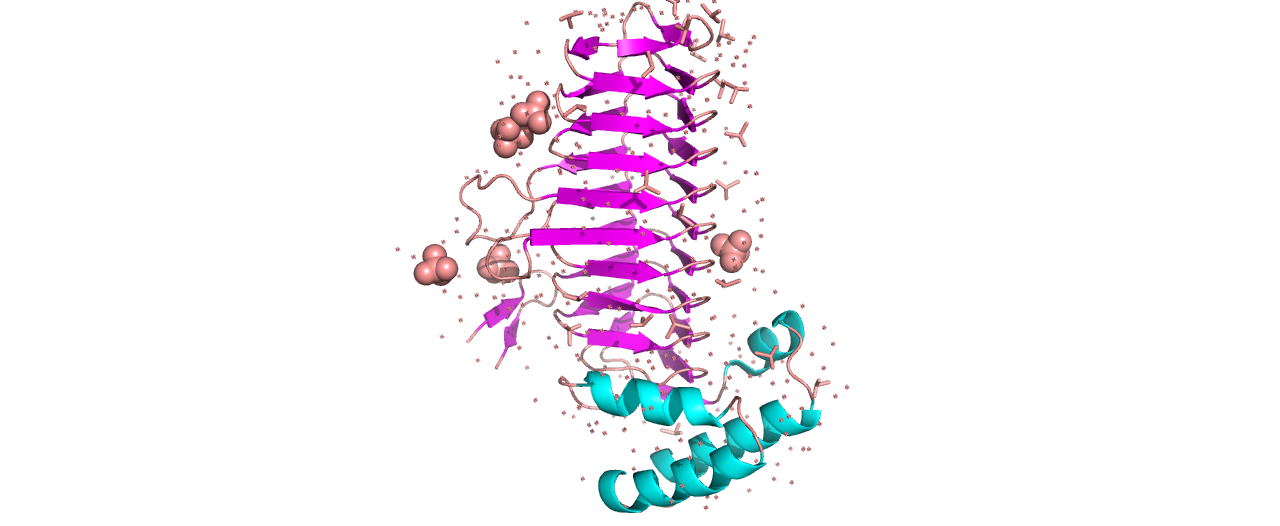
Рисунок 1. Вторичные структуры в составе белка lpxA
Вторичные структуры определялись с помощью программ DSSP и Stride, установленных на kodomo.
Здесь я бы отметила, что Stride не очень удобен для определения бета-листов. В выдаче указаны только бета-тяжи и нет информации о том, как они организуются между собой в бета-лист.
Зато Stride предоставляет визуально удобную выдачу

Рисунок 2. Stride visual assignment
Полученные файлы с результатами: 2aq9.stride, 2aq9.dssp. Эти файлы сравнивались с файлом pdb: 2aq9.pdb.
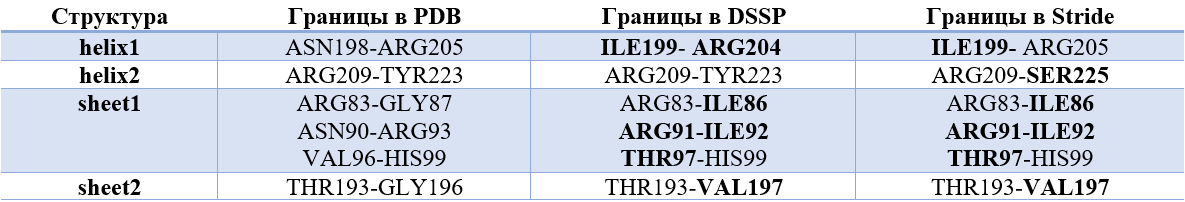
Для анализа я выбрала 4 структуры: 2 альфа-спирали и 2 бета-листа
На рис. 3 показано расположение выбранных структур в субъединице белка lpxA
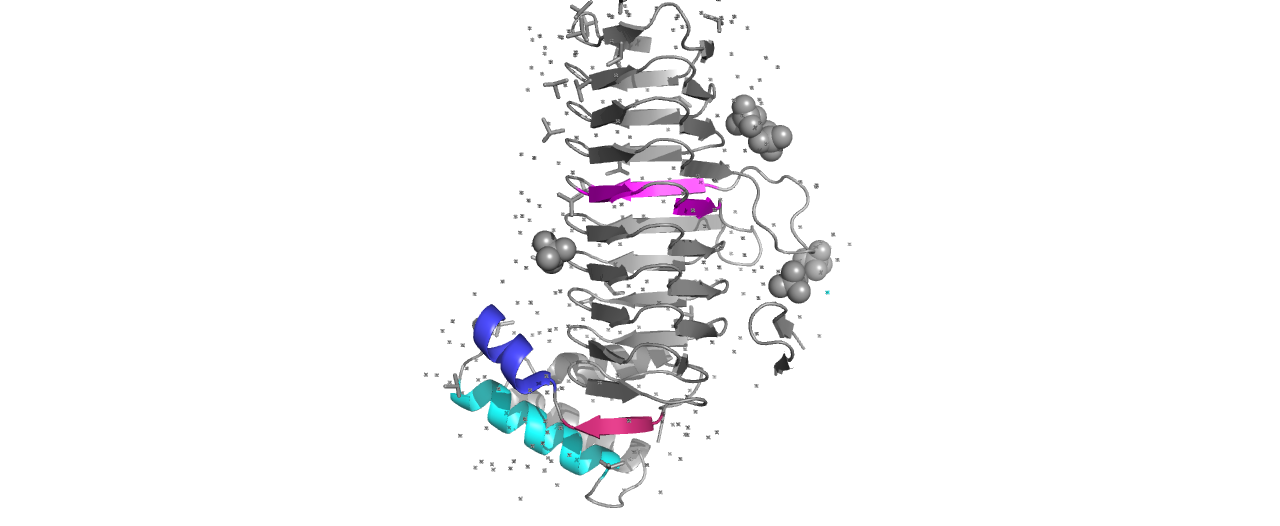
Рисунок 3. Положение выбранных элементов вторичной структуры, окрашенных в разные цвета (helix1 - синий, helix2 - бирюзовый, sheet1 - фиолетовый, sheet2 - розовый)
В таблице 1 приведено сравнение границ данных структур для pdb файла и предсказаний программами DSSP и Stride
Таблица 1. Таблица 1. Сравнение границ выбранных структур в исходном pdb-файле и в файлах с предсказаниями программ DSSP и Stride
Можно отметить, что DSSP и Stride определили бета-листы одинаково, при этом от pdb разметка отличается на 1-2 а.о Я склонна верить разметке DSSP и Stride
В случае альфа спиралей разметка также отличается на 1-2 а.о
Относительно спирали helix1 мне кажется корректной разметка PDB
Для спирали helix2 мне больше нравится разметка PDB и DSSP
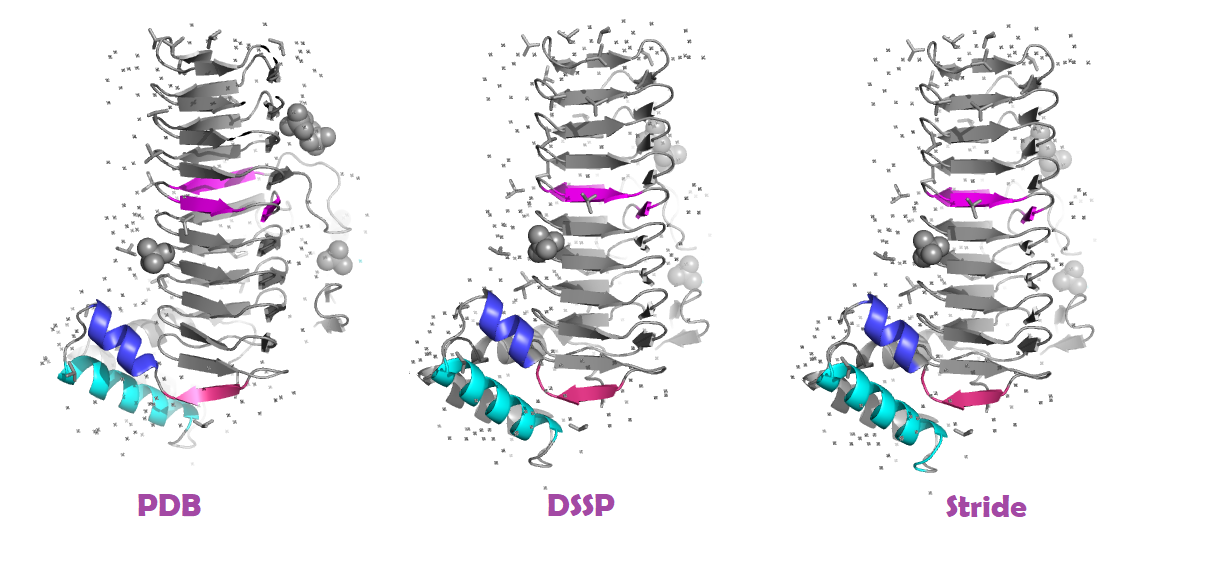
Рисунок 4. Положение выбранных элементов вторичной структуры, окрашенных в разные цвета, на основании pdb и предсказаний DSSP и Stride.
Мне показался интересным тот факт, что Stride выделил мостик (bridge): Asp180-Val181. Однако ни pdb, ни DSSP это не подтвердили