
|
Учебный сайт Алены Корягиной |

|
Главная |
Oбо мне |
Cеместры |
Cсылки |
Анализ качества и очистка чтений
Работа по анализу качества и очистке чтений проводилась для файла Ath_tae_CTTGTA_L003_R2_007.fastq, в котором находятся чтения генома резуховидки.
Анализ качества чтений
Анализ качества чтений проводился с помощью программы FastQC. Для запуска данной программы использовалась команда
fastqc Ath_tae_CTTGTA_L003_R2_007.fastq
Отчет работы этой программы смотрите здесь. В полученном отчете можно найти следующую информацию об исследуемых чтениях:
- Файл содержит 4 млн. чтений;
- Длина последовательности каждого чтения изменяется от 100 до 102 нуклеотидов, средним значением является 101 нуклеотид;
- Содержание G/C в чтениях равно 35%.
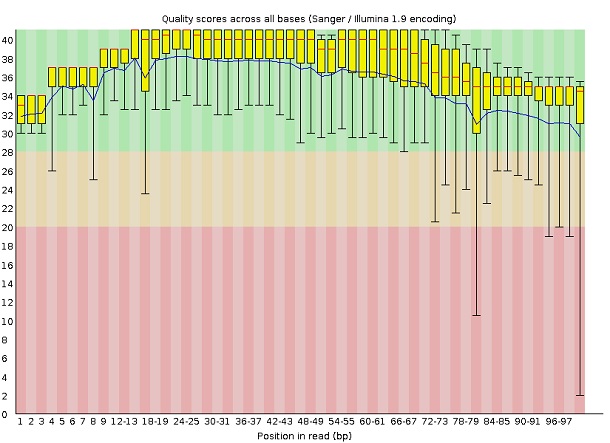
Также в отчете содержаться различные графические представления оценки качества чтений. Одно из представлений представлены ниже на рисунке 1А. На нем можно заметить, что качество чтений в середине очень хорошее, а на концах немного хуже.
Очистка чтений
Далее была проведена очистка чтений с помощью программы Trimmomatiс. Были удалены последовательности адаптеров (опция ILLUMINACLIP:adapters.fa:2:7:7), нуклеотиды с качеством ниже 20 с конца каждого прочтения (опция TRAILING:20), а также все прочтения длинной меньше 50 нуклеотидов (опция MINLEN:50). В исходном файле использовалась кодировка Sanger / Illumina 1.9, поэтому при запуске программы был выбран phred33 формат. В итоге использовались команда:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 Ath_tae_CTTGTA_L003_R2_007.fastq Ath_tae_CTTGTA_L003_R2_007_out.fastq ILLUMINACLIP:adapters.fasta:2:7:7 TRAILING:20 MINLEN:50
В результате был получен новый файл с чтениями - Ath_tae_CTTGTA_L003_R2_007_out.fastq, для которого снова был проведен анализ качества с помощью FastQC. Отчёт по очищенным чтениям можно увидеть здесь.
Теперь сравним два полученных отчета с неочищенными чтениями и с очищенными. Количество чтений среди очищенных уменьшилось до 3 872 176, такое уменьшение можно считать незначительным. Содержание гуанина и цитозина в ридах не изменилось. Также несмотря на то, что теперь длина чтений варьируется от 50 до 101 нуклеотида, среднее значение осталось тем же: 101 нуклеотид (см. график Sequence Length Distribution).
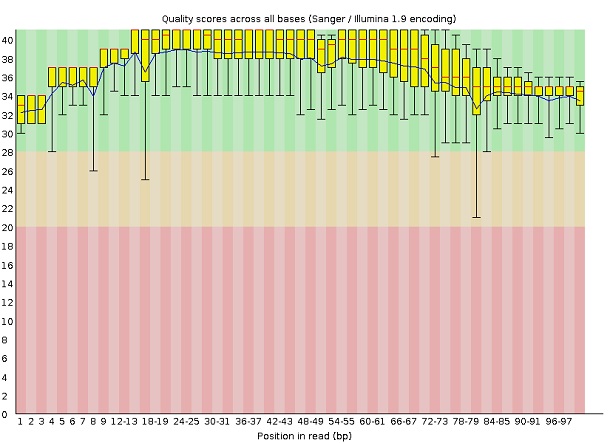
На рисунке 1В представлен график качества нуклеотидов (т.е. точность определения нуклеотида в каждой позиции) – это график Per base sequence quality. По сравнению тем же графиком для неочищенных чтений качество нуклеотидов для очищенных сильно не возросло.
|
|
Рис.1A. График, отображающий качество каждого нуклеотида в исходных чтениях. График получен с помощью программы FastQC. |
Рис.1B. График, отображающий качество каждого нуклеотида в чтениях, очищенных с помощью программы Timmomatic. График получен с помощью программы FastQC. |
В конце концов, из сравнения отчета о качестве неочищенных чтений с отчетом о качестве очищенных чтений можно сделать вывод, что исходные чтения были достаточно качественными.
Дата последнего изменения: 29.12.2014