 |
 |
|
|
Эволюционные домены Выбор семейства доменов Для работы было выбрано семейство доменов TIMELESS. Домен входит в 11 архитектур. AC: PF04821. ID: TIMELESS. Функция: участвует в контроле циркадного ритма. Ссылка на страницу домена в Pfam. В JalView было получено выравнивание (jar, fasta) из всех последовательностей выбранного семейства доменов. Была сделана окраска ClustalX с порогом консервативности, равным 10%. Командой в рабочей директории "python swisspfam-to-xls.py -p PF04821 -i /srv/databases/pfam/swisspfam.gz -z -o task9.txt" получили таблицу, которую затем импортировали в Excel. В Excel получили сводную таблицу, получив список последовательностей с указанием доменной архитектуры. Получили из Uniprot полные записи всех последовательностей, а затем, используя в рабочей директории команду "python uniprot_to_taxonomy.py -i task9_uniprot.txt -o task9_tax.txt", получили таксономию, которую добавили на отдельный лист в таблице. Выбор архитектур Были выбраны две архитектуры: 1) TIMELESS (173 последовательностей). 2) TIMELESS, TIMELESS_C (167 последовательностей). TIMELESS_C AC: PF05029. Тоже участвует в контроле циркадного ритма. Выбор таксона и подтаксона В качестве таксона были выбраны Metazoa, а в качестве подтаксонов - Chordata (C) и Arthropoda (A). Выбор представителей архитектур При помощи листа sort в таблице была получена выборка (перемещена на одноименный лист) из последовательностей двух выбранных архитектур. Используя команду "filter-alignment.py -i "task9.fasta" -m filename.fasta -o filename_align.fasta -a /", где filename имеет значения или task9_t, или task9_t_t_c, получили файлы с выравниваниями последовательностей домена, входящего в архитектуры 1 и 2. Выравнивание представителей архитектур На основе отобранных представителей было сделано "профильтрованное" выравнивание, с окраской по группам ClustalX с порогом консервативности, равным 10%. Выступающие C-концы были срезаны. К сожалению, по каким-то причинам JalView начал "барахлить" и неправильно сохранять файлы в формате "jar" (увеличивает порог консервативности). Рисунок 1. "Профильтрованное" выравнивание, разбитое на группы. Верхняя - T (архитектура: TIMELESS). Нижняя - T+T_C (архитектура: TIMELESS+TIMELESS_C). Окраска по группам CrustalX с 10%-ым порогом консервативности. Выступающие С-концы срезаны. Как можно заметить, C-конец менее консервативен, чем на N-конце. Это совпадает с изображением домена в обеих архитектурах:
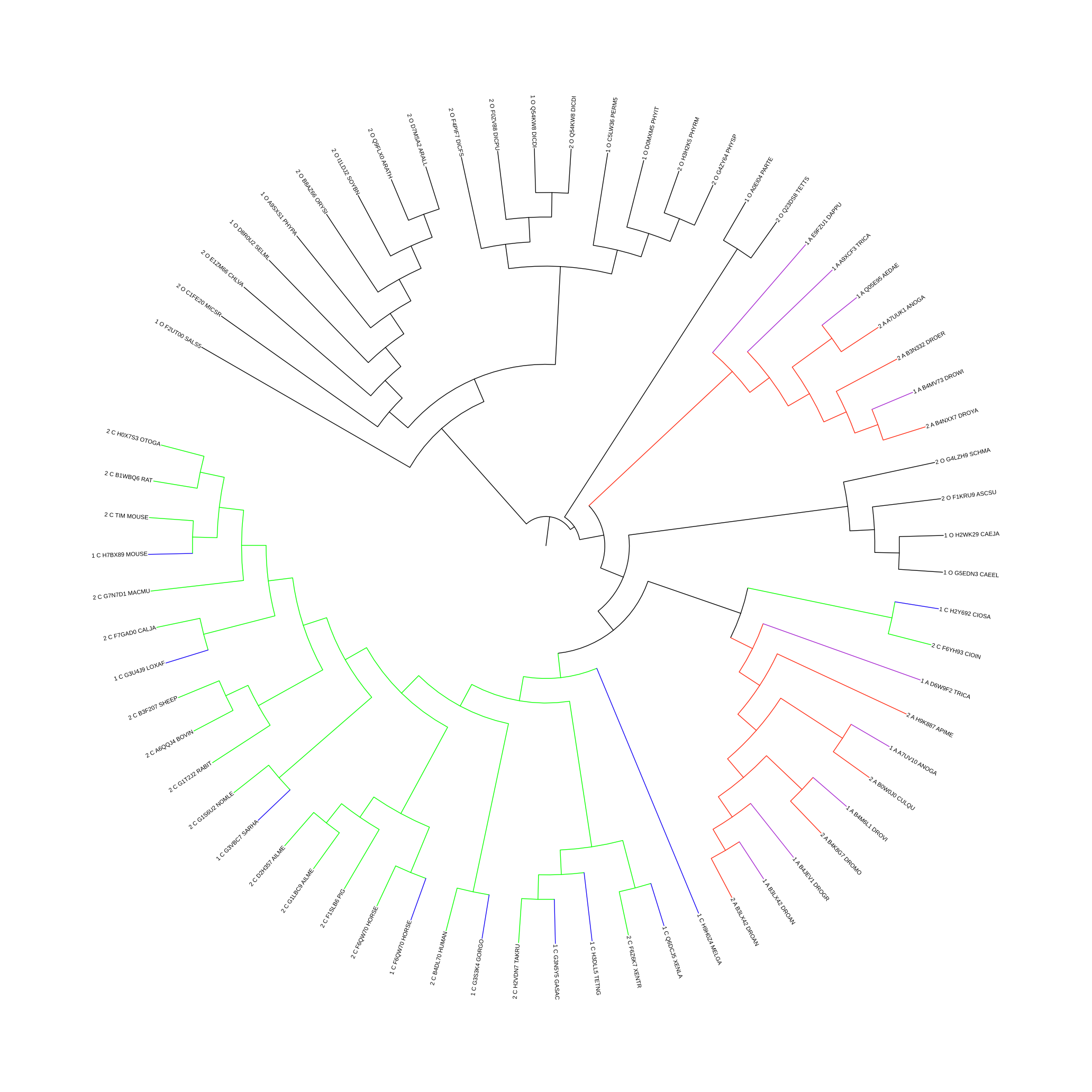
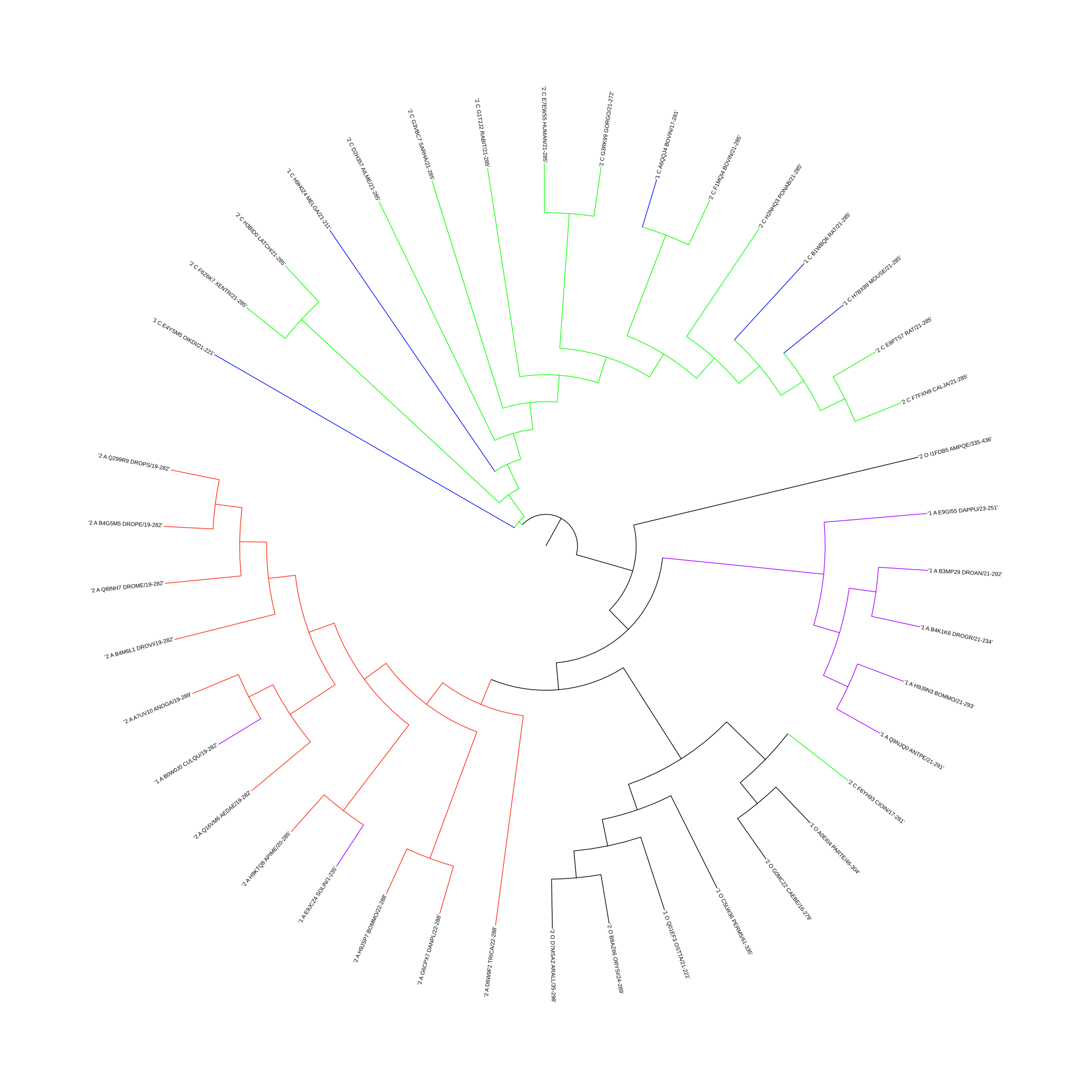
Рисунок 2. Изображение архитектур в Pfam. Эволюционные домены (продолжение) Построение филогенетического дерева Для того, чтобы построить филогенетическое дерево из такого большого количества последовательностей, было решено сделать имена говорящими. Однодоменная архитектура будет обозначаться цифрой 1. Двудоменная - цифрой 2. Представители Chordata обозначаются буквой T. А Arthropoda - буквой A. Остальные будут обозначаться буквой O. Ссылка на измененное выравнивание. Филогенетическое дерево было построено по выравниванию методом "Maximum Likelihood" с бутстрэп-анализом (500 реплик). Рисунок 3. Дерево, построенное методом "Maximum Likelihood" с бутстрэп-анализом (500 реплик). Зеленым отмечены представители Chordata с архитектурой 2, а синим - с архитектурой 1. Красным - Arthropoda с архитектурой 2, а фиолетовым - с архитектурой 1. Черным - все остальные. Для визуализации результатов использовался сайт itol.embl.de Как можно заметить, доменные архитектуры смешались в данном филогенетическом дереве. Возможно, это означает, что доменная архитектура эволюционировала с некоторым непостоянством, что можно объяснить тем, что второй домен (TIMELESS_C) ответственен за то же, что и первый. Таким образом, развод доменов в белке не сильно влиял на жизнеспособность организмов. При этом, если взглянуть на таблицу, то можно заметить, что количество организмов, не имеющих домен Timeless_C (~180), не сильно меньше количества организма, имеющих этот домен (~200). Еще одним вариантом, весьма печальным, может быть наличие ошибок при составлении выборки (наличие среди архитектур 1 и 2 одного и того же белка лошади может на это намекать). В таблице была обнаружена ошибка в формуле, которая влияла на выборку. Поэтому было решено выборку составить снова. Новое выравнивание и дерево. Рисунок 4. Дерево, построенное методом "Maximum Likelihood". Зеленым отмечены представители Chordata с архитектурой 2, а синим - с архитектурой 1. Красным - Arthropoda с архитектурой 2, а фиолетовым - с архитектурой 1. Черным - все остальные. Для визуализации результатов использовался сайт itol.embl.de Как можно заметить, дерево немного изменилось. Если доменные архитектуры все еще смешаны у Chordata, то у Arthropoda заметно разделение путей эволюции. Возможно, у общего предка Arthropoda изначально существовал ген, который затем дуплицировался, дав начало двум белкам, архитектура одного из которых была однодоменной, а другого - двухдоменной. Данное предположение основывается на том, что: А) Есть четкое разделение на филогенетическом дереве двух путей эволюции. Б) Есть два белка с различными архитектурами, но при этом из одного организма (1_A_H9J9N3_BOMMO и 2_A_H9JSP7_BOMMO). Затем, у некоторых организмов из ветки с двухдоменными белками (1_A_B0W0J0_CULQU и 1_A_E9JCZ4_SOLIN) произошла редукция до однодоменного белка. Смущение вызывает факт наличия листа с двухдоменным белком из Chordata (F9YH93_CIOIN) где-то вдали от самих Chordata. Возможно, здесь имеет место ошибка построения дерева. Интересно, что у Chordata существует два организма (BOVIN и RAT) с двумя белками в каждом, имеющими разную архитектуру. Учитывая то, что ген дуплицируется не очень часто, то можно предположить, что или у Chordata белки с данными доменными архитектурами очень консервативны, что не дает найти особых отличий в эволюции двух разных доменных архитектур, или же существуют различные изоформы белка (похоже на изоформы по выравниванию методом Clustal у белков крысы, но не похоже по выравниванию методом Clustal белков у быка). Выводы Вероятно, у нас имеется дупликация гена в Arthrapoda, что привело к независимой эволюции двух доменных архитектур, в одной из которых у двух организмов произошло независимое упрощение двудоменной архитектуры до однодоменной. У Chordata же наблюдается непостоянство доменной архитектуры. Наличие двух белков в одном организме (RAT и BOVIN) можно объяснить или дупликацией гена у общего предка Chordata и Arthropoda с крайней консервативностью домена TIMELESS у Chordata, или же наличием двух изоформ белка, один из которых обладает доменом TIMELESS_C, а другой - нет. Но тут возникает сложность в виде независимой эволюции двух доменных архитектур у Arthropoda. Возможно, что у Chordata эволюция пошла путем создания двух изоформ, а у Arthropoda - путем дупликации гена. |
 |
 |
|