Восстановление предкового состояния доменной архитектуры
Задание 1. Построение выравнивание представителей домена Pfam белков с разной доменной архитектурой
Для выполнения данного задания был выбран С-концевой домен белка G кобаламинового синтеза.
ID: CbiG_C
AC: PF01890
Функция домена: участие в анаэробном биосинтезе кобаламина, а именно в превращении кобальт-прекоррина 5 в кобальт-прекоррин 6. Он отвечает за открытие дельта-лактонового кольца и вытемнение С2-единицы.
Данный домен присутствует в 17 архитектурах (подробнее по ссылке).
Топ-5 самых распространенных архитектур:
- CbiG_N, CbiG_C - 177 последовательностей
- CbiG_C - 160
- CbiG_N, CbiG_mid, CbiG_C - 156
- CbiG_N, CbiG_mid, CbiG_C, TP_methylase - 57
- CbiG_N, CbiG_C, TP_methylase - 42
Проект JalView содержит выравнивание всех последовательностей, содержащих данный домен.
Для дальнейшей работы были выбраны 2 доменные архитектуры:
- CbiG_N, CbiG_mid, CbiG_C (156 последовательностей) (Рис. 1а). Помимо С-концевого домена здесь присутствуют также N-концевой домен и центральный регион белка Cbi.
В дальнейшем последовательности с этой архитектурой будут нести префикс 3_.

Рис. 1а. Доменная архитектура CbiG_N, CbiG_mid, CbiG_C.
- CbiG_N, CbiG_C (177 последовательностей) (Рис. 1b). Здесь добавлен только N-концевой домен. В дальнейшем последовательности с этой архитектурой будут нести префикс 2_.

Рис. 1b. Доменная архитектура CbiG_N, CbiG_C.
Файл Excel содержит сводную таблицу с информацией о доменной архитектуре и таксономии (лист "Сводная таблица").
Затем нужно было выбрать около 20 представителей каждого подтаксона с каждой доменной архитектурой. Результат: лист "CUT" файла Excel и проект JalView, содержащий выравнивания 4 групп последовательностей исходного домена (группы по подтаксонам и доменным архитектурам). В выравниваниях - раскраска ClustalX с порогом консервативности 20 % во всех группах. Предварительно из выравнивания были удалены пара последовательностей, которые были выровнены сильно хуже других. В остальном выравнивание можно считать правильным, так как в нем нет разделений на фрагменты и, как мне кажется, нет неправильно выровненных последовательностей.
Задание 2. Построение дерева.
По полученному выравниванию в программе MEGA было построено филогенетическое дерево последовательностей методом максимального правдоподобия. На Рис. 2 - изображение данного дерева с выделенными группами. Скобочная формула по ссылке.
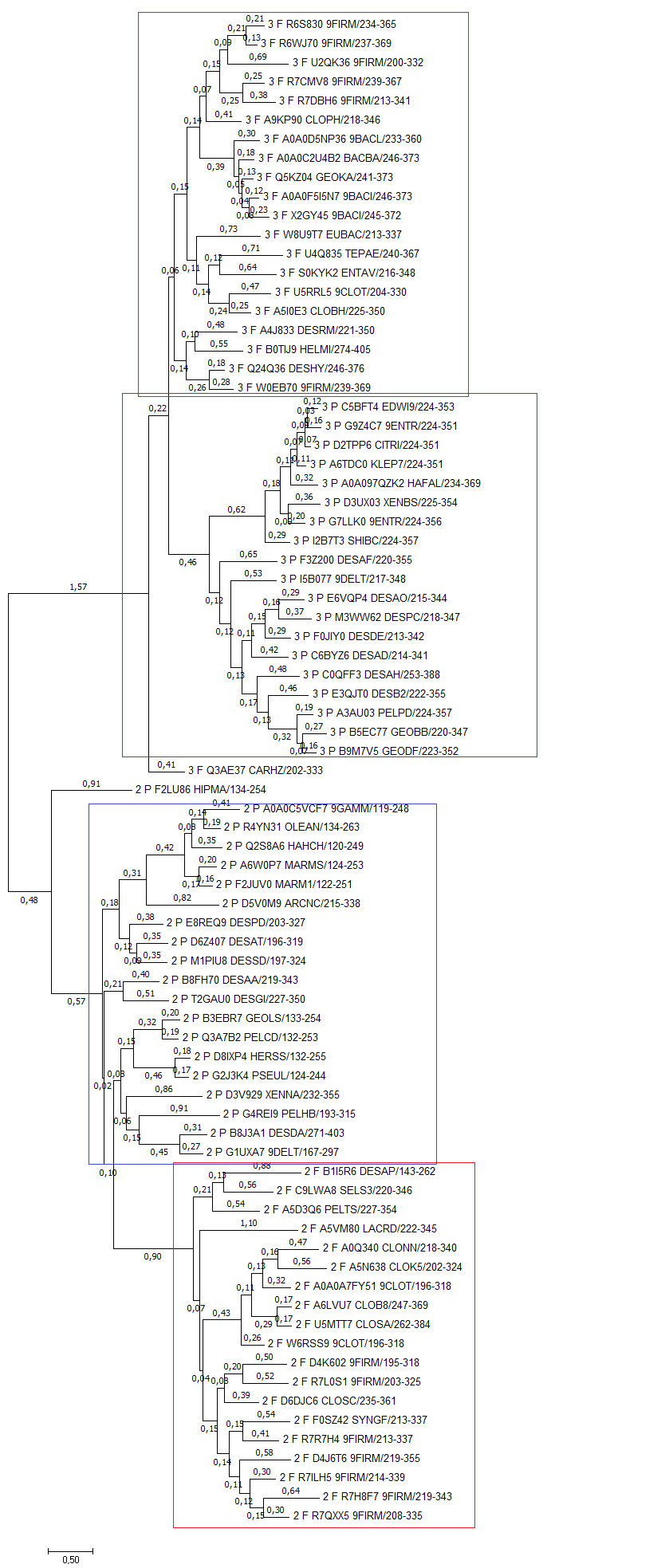
Из полученного дерева можно сделать следующие выводы об эволюции:
- Утрата центрального домена произошла раньше, чем произошло разделение бактерий на выбранные филлумы.
- Внутри разделения по доменной архитектуре дерево хорошо разбивается на клады, соответствующие филлумам, за исключением одной последовательности в каждом случае:
2_P_F2LU86_HIPMA и 3_F_Q3AE37_CARHZ. Если взглянуть на эти последовательности, то действительно можно заключить, что они выбиваются среди остальных.
Задание 3. Построить профиль подсемейства и охарактеризовать качество его работы
Для хорошего подсемейства 3_F был построен HMM-профиль (ссылка).
На основе результатов поиска был получен файл Excel (на листе "Данные" содержатся данные об E-value для каждой находки и столбец с правильными находками (столбец "Результаты работы программы"). Затем с помощью функции ВПР были определены соответствия между полученными данными и предсказаниями. На основе этих данных была построена ROC-кривая (Рис. 3).
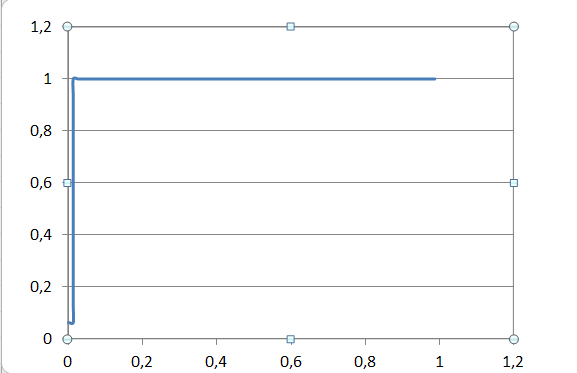
Рис. 3. ROC-кривая.
ROC-кривая получилась очень крутая. По ней можно выбрать порог E-value = 1,00E-64.
На самом деле
принадлежит подсемейству
не принадлежит
сумма
Выше порога по профилю
16
1
17
Ниже порога
0
75
75
сумма
16
76
92
По этим результатам можно судить о том, что HMM-профиль можно использовать для выделения подсемейства.