| Главная страница | Семестры | О себе | Ссылки |
Упражнения
1. seqret (1)
Все записи UniProt, AC которых начинается на R10, были собраны в файл (рис. 1).
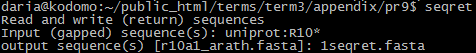
Рис. 1. Команда seqret
2. seqretsplit
Полученный в упражнении 1 файл я разделила на несколько (рис. 2).
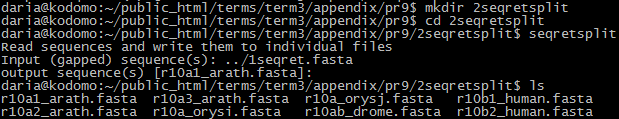
Рис. 2. Команда seqretsplit
3. seqret (6)
Полученный в упражнении 1 файл я перевела из формата .fasta в формат .msf (результат).

Рис. 3. Команда seqret
4. compseq
В последовательности одного из контигов сборки генома C. elegans нашла частоту тринуклеотидов и сравнила с ожидаемой частотой (опция -calcfreq). Результат, а команда на рисунке 4.

Рис. 4. Команда compseq
5. transeq (5)
Последовательность гена из практикума 6 транслирована в шести рамках - результат, команда на рисунке 5.

Рис. 5. Команда transeq
Сравните аннотации генов белков в геноме Streptococcus pneumoniae с трансляциями длинных открытых рамок считывания
У бактерии Streptococcus pneumoniae всего одна хромосома, соответствующая ей запись RefSeq - NC_003098.1. Также я сохранила запись с аннотациями в формате GeneBank, но для работы я использовала последовательность генома fasta, так как запись gb содержит только аннотации.
Для получения трансляций открытых рамок использовалась программа getorf пакета EMBOSS. Команда представлена на рисунке 6, а вот результат.

Рис. 6. Команда getorf
Затем нужно было получить список координат и ориентаций открытых рамок, для чего использовалась команда infoseq (рис. 7). Была получена таблица.

Рис. 7. Команда infoseq
Полученные данные также были сохранены в формате книги Excel.
Далее требовалось составить другую таблицу - со списком аннотированных генов белков. Информацию для ее составления я брала не из файлов .ptt и .faa, как указано в задании, а кликнув на ссылку Gene на странице генома в NCBI и выбрав фильтр Protein coding.
Затем две таблицы были склеены в одну, записи были сранжированы по координате начала. По таблице я вижу следующее:
-
Предсказанных генов примерно в 5 раз больше, чем аннотированных, то есть открытая рамка может не являться (и чаще не является) геном.
-
Есть аннотированные гены короче заявленной при отборе открытых рамок длины в 180 нуклеотидов, некоторые показаны на рисунке 8. Но в среднем аннотированные гены длиннее предсказанных.
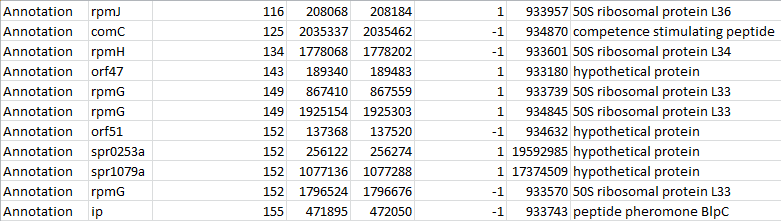
Рис. 8. Примеры коротких аннотированных генов
-
Открытые рамки начинаются раньше, чем соответствующие им существующие в реальности гены. Пример на рисунке 9.

Рис. 9. Реальный ген и предсказанная для него рамка считывания
-
Чаще всего рамка считывания длиннее соответствующего аннотированного гена, но не всегда.
-
Среди предсказанных рамок очень много перекрывающихся, но среди аннотированных генов это не распространено - по крайней мере, я не заметила перекрытия рамок при простотре частей таблицы глазами.
© Дарья Горбачева | изменено 3.03.2016 |

