| Главная страница | Семестры | О себе | Ссылки |
Выбор объектов и получение выравнивания
Для выполнения практикума взят домен zf-MIZ (ID в базе данных Pfam: PF02891). Это один из видов цинковых пальцев - ДНК-связывающего домена.
Для данного домена известно:
1688 последовательностей
551 вид
1 взаимодействие
6 пространственных структур
Непосредственно с Pfam было получено выравнивание семейства:
Проект .jvp (раскрашено CrustalX с порогом консервативности 17%)

Рис. 1. Выбранные доменные архитектуры
Для дальнейшей работы были выбраны архитектуры с рисунка 1. Одна содержит, помимо zf-MIZ, домен SAP, а другая - еще и домен PINIT.
Был запущен скрипт:
python swisspfam-to-xls.py -p PF02891 -i /srv/databases/pfam/swisspfam.gz -z -o architecture.txt |
architecture.txt - был перенесен в Excel.
Были скачаны полные записи всех последовательностей с Uniprot Retrieve. Файл с последовательностями был подан скрипту:
python uniprot-to-taxonomy.py -i seq.uniprot -o seq_taxes.txt |
Информация из полученного файла была добавлена к сводной таблице architecture.xlsx.
По данным из сводной таблицы отобраны представители архитектур так, чтобы в 2-х подтаксонах было не менее 5 представителей 2-х выбранных архитектур. Выбраны подтаксоны:
Eukaryota Metazoa (обозначение _Z)
Eukaryota Fungi (обозначение _F)
Выбранные последовательности были переданы Uniprot Retrieve. Полученные fasta были выровнены в Jalview - проект.
Построение филогенетического дерева домена
Дерево было построено алгоритмом Neighbour Joining cо 100 бутстреп-репликами по модифицированному файлу с выравниванием. Спереди к названиям последовательостей добавлены обозначения такономии (выше) и обозначения архитектуры - 1 или 2.

Рис. 2. Дерево
Скобочная формула делрева: файл
Видно, что хорошо разделились грибы и животные, а вот разные архитектуры - нет.
Профиль
Для выполнения этого задания была выбрана клада на дереве, соответствующая 2_М. Ее выравнивание сохранено отдельно в файл .fasta.
По нему был построен профиль в HMM:
hmm2build hmm_built profile.fasta |
Профиль калибруется:
hmm2calibrate --histfile hmm_histo hmm_built |
Профиль используется для поиска в файле со всеми последовательностями, содержащими домен (результат):
hmm2search hmm_built seq.uniprot > hmm_search |
Итог - таблица.
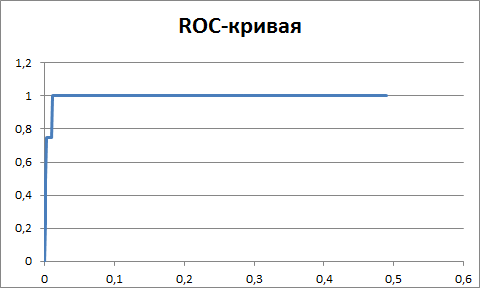
Рис. 3. Кривая
Порог E value - 0.
Таблица 1. Статистические параметры для данного E value
Принадлежит семейству |
Не принадлежит семейству |
Сумма |
|
Выше порога по профилю |
4 |
12 |
16 |
Ниже порога по профилю |
0 |
955 |
955 |
Сумма |
4 |
967 |
971 |
© Дарья Горбачева | изменено 4.10.2016 |

