BLAST
Таблица 1. Результаты поиска гипотетических гомологов белка XXXX_BACSU
| Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr" | |
1. Лучшая находка (с последовательностью исходного белка) |
|||
| Accession | P70994 | 2OP8_A | NP_391634.1 |
| E-value | 1e-37 | 1e-37 | 7e-36 |
| Вес (в битах) | 125 | 122 | 125 |
| Процент идентичности | 100% | 100% | 100% |
2. Число находок с E-value < 10–10 |
14 | 2 | 148 |
3. "Худшая из удовлетворительных" находка (последняя в выдаче с E-value < 1) |
|||
| Номер находки в списке описаний | 39 | 17 | 1268 |
| Accession | C6DH49 | 2VSF_A | ZP_00955968.1 |
| E-value | 0.86 | 0.62 | 0.99 |
| Вес (в битах) | 28.9 | 27.3 | 35.0 |
| % идентичности | 25 | 34 | 29 |
| % сходства | 57 | 65 | 49 |
| Длина выравнивания | 76 | 38 | 55 |
| Координаты выравнивания (от-до, в запросе и в находке) | 1-51 1-52 |
23-62 453-490 |
1-55 66-120 |
| Число гэпов | 1 | 3 | 0 |
Мне удалось найти исходный белок в Swiss-Prot и "nr", а также его структуру в PDB. Число явных гомологов (E-value < 1e-10) при поиске по разным базам данных различается. В базе PDB меньше всего, и это понятно – не так уж много белков имеют известную трехмерную структуру. Очень много гомологов из «nr», и это связано со структурой базы данных - много белков, которые только предполагаются. Номер последней находки Swiss-prot – 54 (E-value 10.0), PDB – 19 (E-value 9.8), «nr» - 1380 (E-value 9.8). Все запросы были лимитированы значением E-value 10.0.
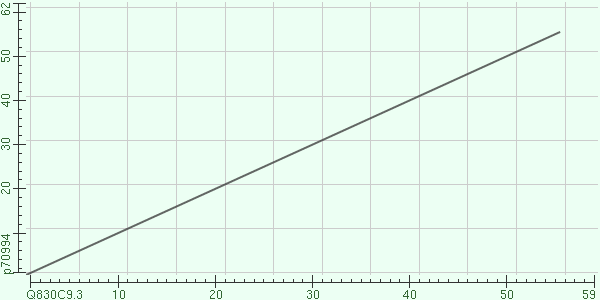
Составила карты локального сходства для моего белка и одного из его гомологов с помощью программы BLAST(рисунки 1 и 2).
Рис. 1. Карта локального сходства для E-value 10.
Рис. 2. Карта локального сходства для E-value 0.01.
Как видно, карты локального сходства данных двух белков для разных значений E-value не отличаются.
Мне удалось найти гомологов моего белка YWHB в базах Swiss-Prot, "nr" и PDB. В таблицах 2 и 3 представлены результаты поиска с использованием матриц BLOSUM62 и BLOSUM45 соответственно.
| База данных | Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr" |
| Таксон | Lactobacillales | Actinobacteria | Actinobacteria |
| Номер находки в списке описаний | 8 | 1 | 66 |
| Accession | Q9CHZ4.3 | 1223RY0_A | ZP_10803365.1 |
| E-value | 2e-06 | 2e-07 | 0.001 |
| Вес (в битах) | 40.4 | 40.4 | 37.7 |
| % идентичности | 49 | 32 | 31 |
| % сходства | 72 | 55 | 59 |
| Длина выравнивания | 55 | 59 | 61 |
| Координаты выравнивания (от-до, в запросе и в находке) | 1-55 1-55 |
2-60 1-59 |
1-58 1-61 |
| Число гэпов | 0 | 0 | 2 |
Таблица 2. Гомологи белка YWHB в различных таксонах. Использована матрица BLOSUM62.
| База данных | Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr" |
| Таксон | Clostridia | Actinobacteria | Actinobacteria |
| Номер находки в списке описаний | 1 | 1 | 75 |
| Accession | Q3A9N9.1 | 3RY0_A | YP_002780940.1 |
| E-value | 0.001 | 7e-08 | 0.001 |
| Вес (в битах) | 33.9 | 41.2 | 38.0 |
| % идентичности | 35 | 32 | 27 |
| % сходства | 67 | 55 | 48 |
| Длина выравнивания | 46 | 59 | 62 |
| Координаты выравнивания (от-до, в запросе и в находке) | 13-58 398-442 |
2-60 1-59 |
1-62 1-62 |
| Число гэпов | 1 | 0 | 0 |
Таблица 3. Гомологи белка YWHB в различных таксонах. Использована матрица BLOSUM45.
Как видно, при использовании матрицы BLOSUM45 появляются гомологи с меньшим процентом сходства, идентичности и весом. Первая находка появилась раньше таксономически. Все это происходит потому, что матрица BLOSUM45 устанавливает меньший порог для признания белков гомологичными.
Сравнение различных интерфейсов программы BLAST
BLAST на сервере NCBI представляет собой, на мой взгляд, самую "продвинутую" систему. Есть специализированный поиск по иммуноглобулинам, поиск консервативных доменов и др. Много настраиваемых параметров.
На сервере EMBL-EBI есть "заранее заготовленный" поиск по таксонам. Также удобно то, что все варианты таксонов, баз данных в одном окошке, и ничего искать не надо. Имеются несколько видов последовательностей. И еще очень милый дизайн - подходящие цвета, которые не режут глаз, мягкие формы.
Поиск на сервере UniProt мне не понравился. Недружелюбный интерфейс, поиск только по UniProt. Мало функций, очень мало.
Подведем итоги: поиск на NCBI серьезный, а на EMBL-EBI - красивый. Красивый - это немаловажно.