Паттерны и банк PROSITE
Вероятная встреча моего слова (CHICK) в базе Prosite (перемножение частот букв слова и количества букв) - 0.0137*0.0227*0.0596*0.0137*0.0584*191670831 = 2.842379905 - 2-3 раза.
В ответ на запрос c-h-i-c-k Prosite дал ответ «The search term has not been found in PROSITE entries.»
Создала "сильный" и "слабый" паттерны для поиска гомологов моего белка YWHB_BACSU в банке SwissProt, результаты поиска представила в таблице 1.
| Характеристика паттерна | Паттерн | В скольких последовательностях банка Swiss-Prot и Trembl найден мотив, удовлетворяющий паттерну | Сколько последовательностей из моего выравнивания найдено | Вероятность получения случайной последовательности в Swiss-Prot по такому паттерну |
| Сильный | P-x(3)-V-x(6)-[ST]-x(1)-[EK]-x(1)-K-x(2)-[LV]-[AV]-x(3)-T-x(15)-[VI]-x(3)-E-x(5)-[YW]-x(2)-[AG]-G-x(4)-[DE] | 208 | 14 | 3,30819E-07 |
| Слабый | [PV]-x(3)-[VIM]-x(6,9)-[STIND]-x(1)-[EDS]-x(1)-K-x(3)-[AIV]-x(1,3)-[TS]-x(15)-[VILM] | 2363 | 23 | 60 |
Таблица 1. Результаты поиска гомологов белков из моего множественного выравнивания по слабому и сильному паттерну.
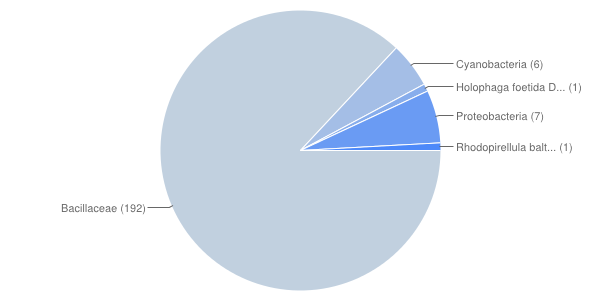
Выводы, которые можно сделать из результатов поиска: так как поиск последовательностей для множественного выравнивания производился по базе данных refseq, то, скорее всего, не все последовательности должны найтись при поиске в Swiss-Prot и Trembl, тем более только в Swiss-Prot. Но были найдены другие гомологи моего белка, поэтому результаты поиска с помощью паттернов PROSITE следует признать удовлетворительными. Правильность сделанных выводов доказывает рисунок 1.
Рис. 1. Распределение хитов по таксонам.
Нашла все мотивы PROSITE в последовательности моего белка. Находки представила в таблице 2.
| Идентификатор документа Prosite (AC) | Название мотива | Краткое описание мотива | Тип подписи (паттерн, профиль) | Паттерн (если это паттерн) | Специфична ли подпись? | Сколько мотивов нашлось в белке? |
| PS00005 | PKC_PHOSPHO_SITE | Protein kinase C phosphorylation site | Паттерн | [ST]-x-[RK] | Неспецифична | 1 |
| PS00009 | AMIDATION | Amidation site | Паттерн | x-G-[RK]-[RK] | Неспецифична | 1 |
| PS00004 | CAMP_PHOSPHO_SITE | cAMP- and cGMP-dependent protein kinase phosphorylation site | Паттерн | [RK](2)-x-[ST] | Неспецифична | 1 |
| PS00006 | CK2_PHOSPHO_SITE | Casein kinase II phosphorylation site | Паттерн | [ST]-x(2)-[DE] | Неспецифична | 1 |
Таблица 2. Мотивы PROSITE в последовательности моего белка.
Выбрала сильный паттерн, нашла еще раз и сохранила все находки в формате fasta, открыла в JalView и построила выравнивание последовательностей. На рисунке 2 представлено множественное выравнивание последовательностей, найденных по сильному паттерну.
Рис. 2. Множественное выравнивание последовательностей, найденных по сильному паттерну. Порог консервативности, используемый для окраски - 50.
Заметим особенности выравнивания: многие участки сильно консервативны, и это немного зависит от вторичной структуры, на которую мы опирались - YWHB_BACSU; отсутствуют гэпы.