A- и В- формы ДНК. Структура РНК
Для выполнения задания построила модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA: с помощью программы Рutty, используя протокол ssh, подсоединилась к серверу kodomo.cmm.msu.ru, перешла в директорию term3/block1/pr2. Ввела следующие команды, что бы указать путь к 3DNA:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
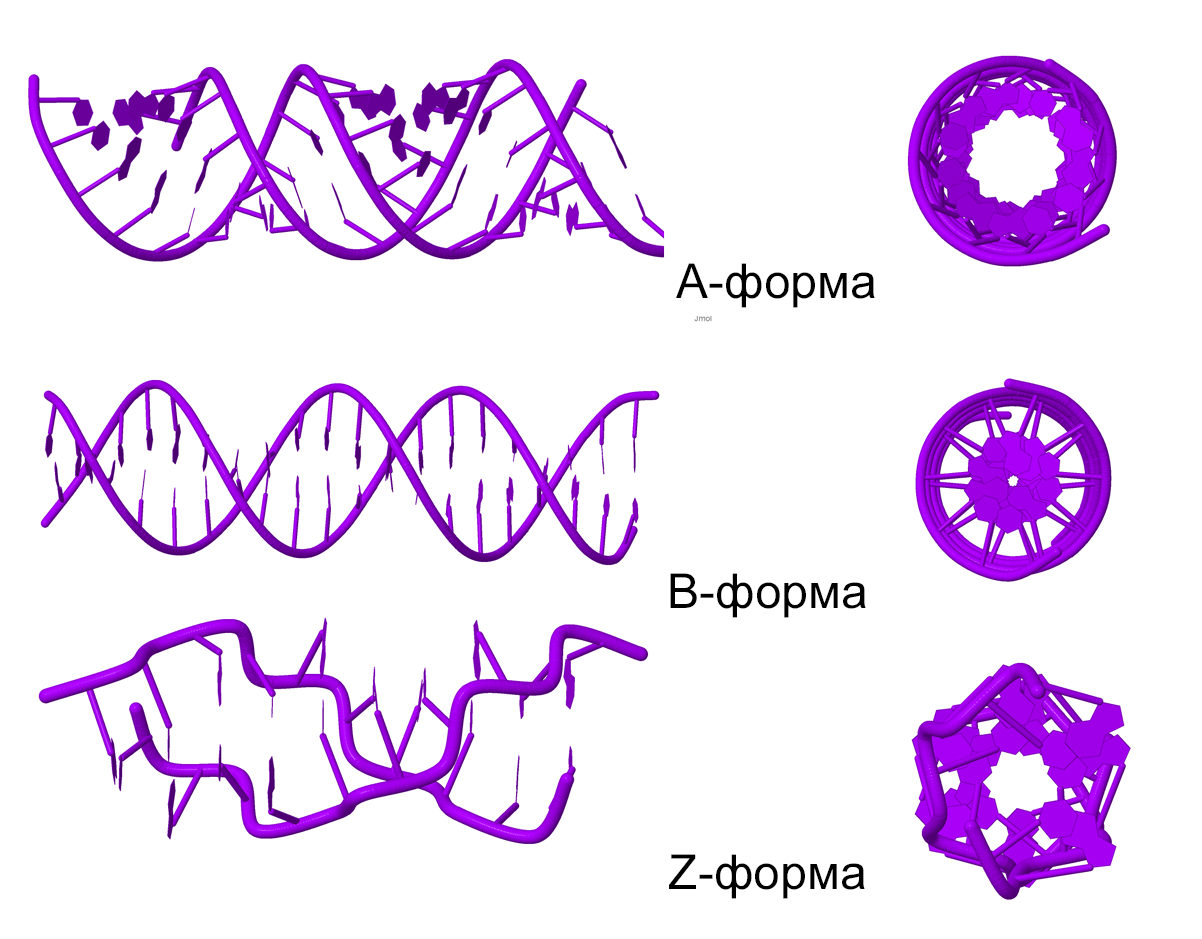
С помощью программы fiber пакета 3DNA построила A-, B- и Z-форму дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 раз повторенную последовательность "gatc". Структуру дуплекса в А-форме сохранила в файле gatc-a.pdb, структуру дуплекса в В-форме - в файле gatc-b.pdb, структуру дуплекса в Z-форме - в файле gatc-z.pdb. Построенные трехмерные модели представлены на рисунке 1.
Рисунок 1. Модели структур A-, B- и Z-формы ДНК, построенные с помощью инструментов пакета 3DNA.
Средства JMol для работы со структурами нуклеиновых кислот.
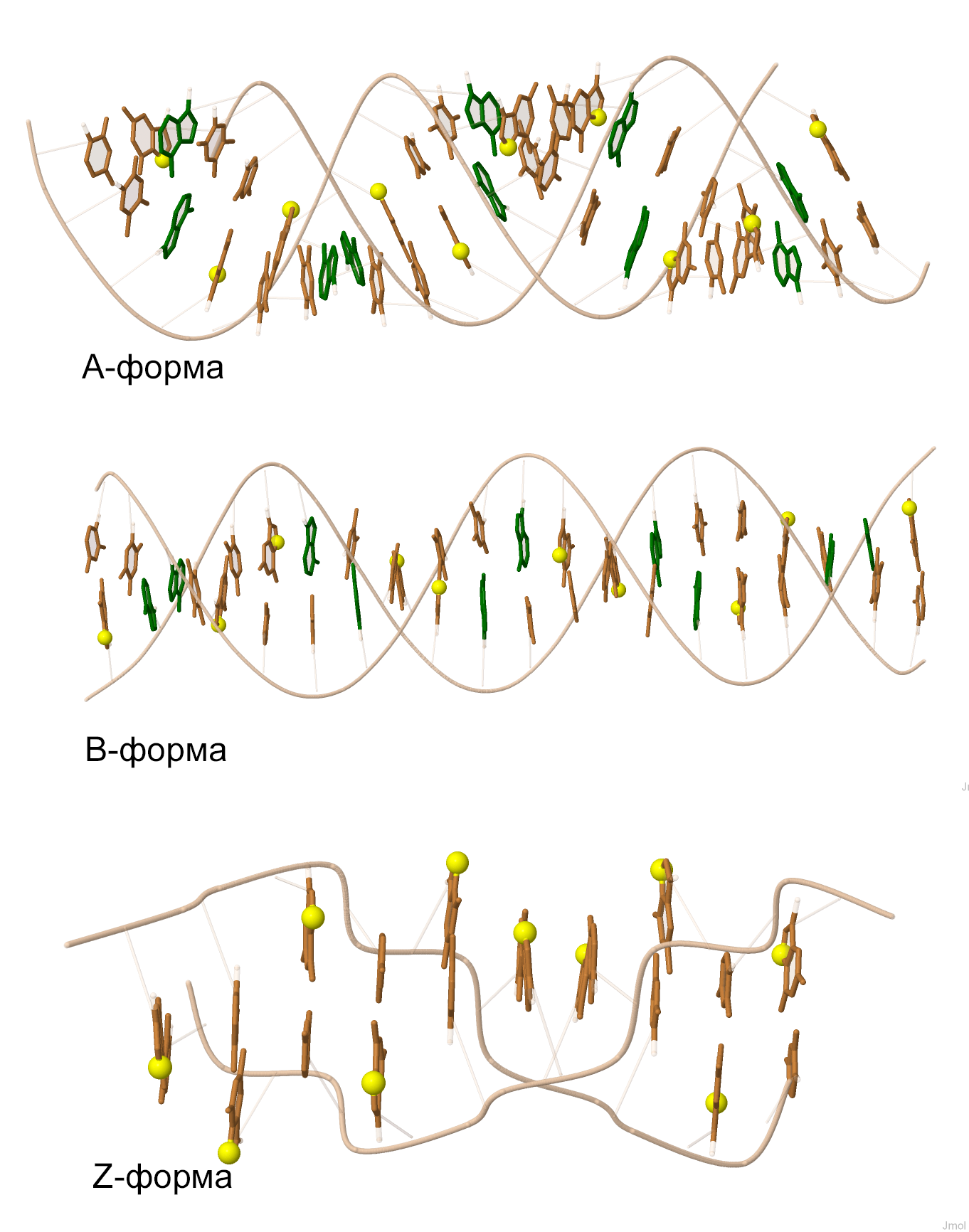
Научилась выделять разные атомы и химические группировки, используя предопределенные множества JMol. Выделила цветом или способом отображения сахарофосфатный остов ДНК, все нуклеотиды, все аденины, атом N7 во всех гуанинах (рисунок 2).
Рисунок 2. Модели структур A-, B- и Z-формы ДНК. Бежевым выделен сахарофосфатный остов, все нуклеотиды имеют палочковидную форму, зеленым цветом обозначены аденины, ярко-желтые шарики - все атомы N7 в гуанинах.
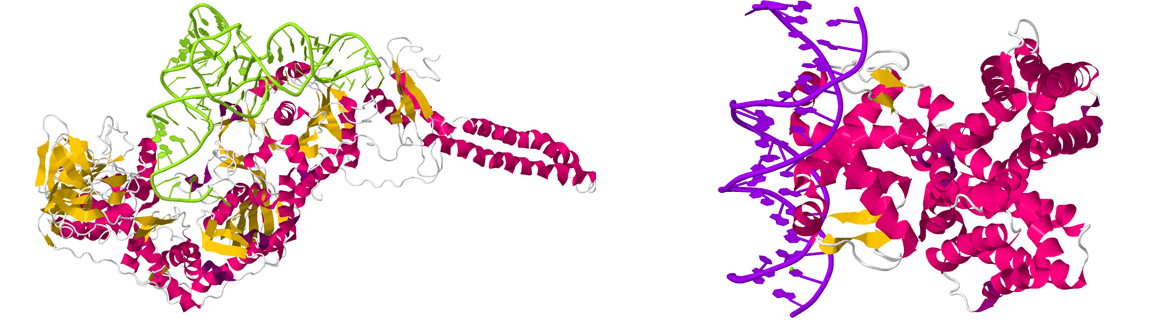
Получила файлы PDB по заданным в таблице идентификаторам (1eiy - фенилаланин-тРНК синтетаза; 1hw2 - FadR-ДНК комплекс), выбрала подходящую структуру (рисунок 3).
Рисунок 3. Фенилаланин-тРНК синтетаза (слева) и FadR-ДНК комплекс (справа). Окрашено в соответствии со вторичной структурой: светло-зеленый - РНК, фиолетовый - ДНК.
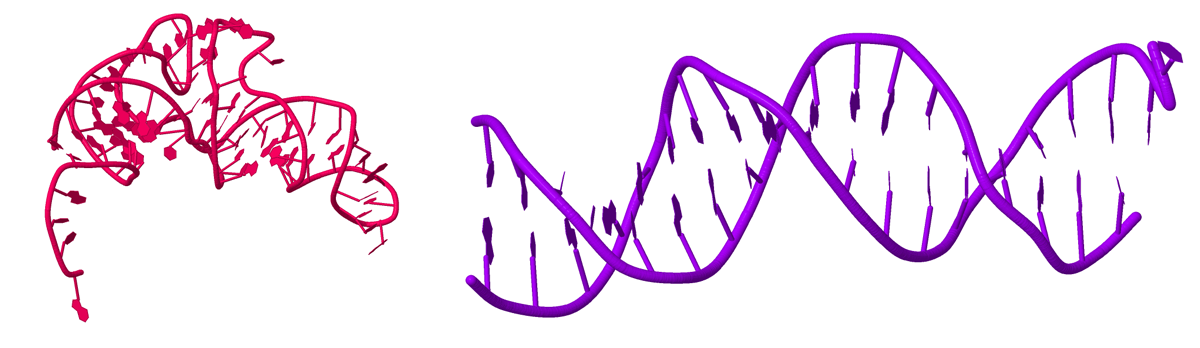
Открыла полученные файлы PDB в JMol. Получила изображение только нуклеиновой кислоты (рисунок 4). Внимательно рассмотрела и проверила заданные структуры ДНК и РНК на наличие разрывов (рисунки 5-7).
Рисунок 4. РНК из структуры фенилаланин-тРНК синтетазы (слева) и ДНК из структуры FadR-ДНК комплекса (справа).
Рисунок 5. Ленточная структура фенилаланин-тРНК синтетазы (вверху) и FadR-ДНК комплекса (внизу).Светло-зеленый шарик в структуре ДНК - атом магния.
Рисунок 6, 7. Нуклеотиды фенилаланин-тРНК синтетазы (слева) и FadR-ДНК комплекса (справа). Потерь атомов не наблюдается. Картинки кликабельны.
Рисунок 5 был создан для определения непрерывности цепи нуклеиновой кислоты; как видно, разрывов нет. Далее шла проверка на наличие всех атомов в нуклеотидах - удобнее просматривать разные азотистые основания по отдельности. Вывод: разрывов в изображении нуклеиновых кислот нет.
Сохранила координаты атомов только ДНК и РНК в отдельных файлах для дальнейшей работы.
Сравнение структур 3-х форм ДНК с помощью средств JMol.
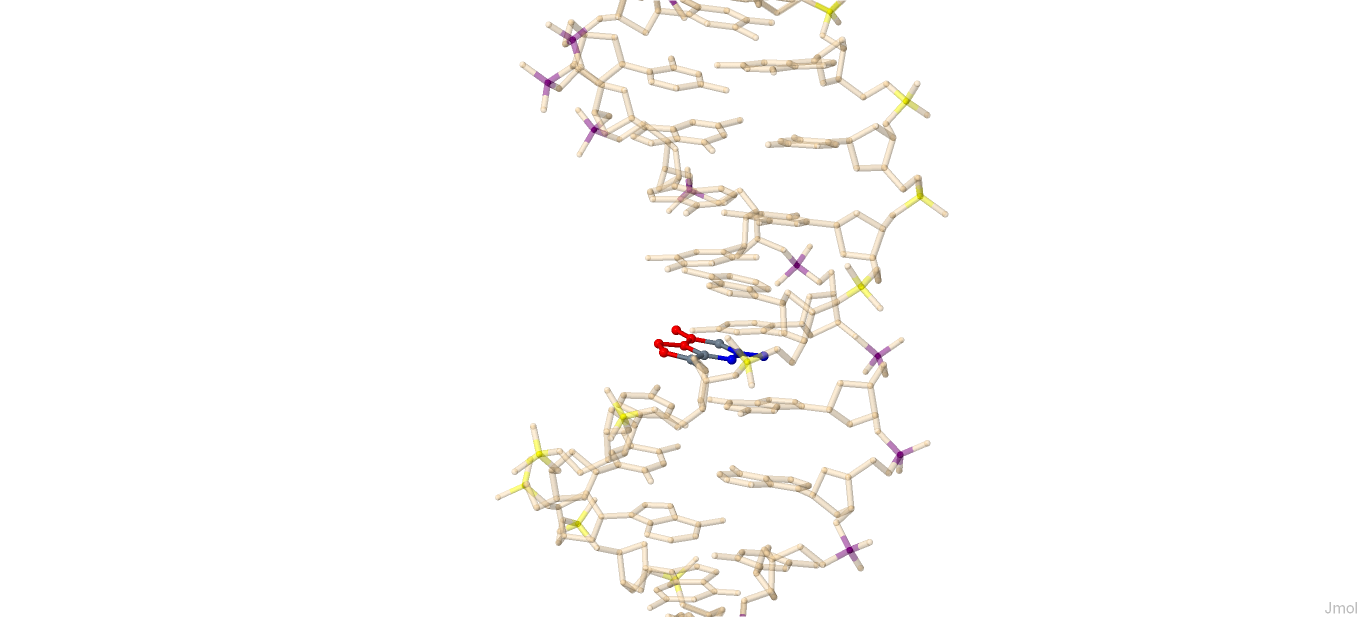
Открыла в JMol файл gatc-b.pdb, полученный при выполнении задания 1. Рассмотрела структуру и визуально определила большую и малую бороздку. Выбрала заданный мне гуанин в середине структуры (рисунок 8). Определила, какие атомы основания явно обращены в сторону большой бороздки (выделены красным), а какие - в сторону малой (выделены синим, рисунок 9).
Рисунок 8, 9. Гуанин в середине структуры В-спирали ДНК. Синим выделены атомы, обращенные в сторону малой бороздки, красным - в сторону большой бороздки.
Нужно отметить, что положение атомов не сильно зависит от структуры спирали, хотя в Z-форме в сторону малой бороздки обращается на один атом больше.
Сравнила основные спиральные параметры разных форм ДНК (таблица 1).
Таблица 1. Основные спиральные параметры разных форм ДНК.
| A-форма | B-форма | Z-форма | |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (Â) | 28.03 | 33.75 | 43.50 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (Â) | 16.81 | 17.91 | 18.30 |
| Ширина малой бороздки (Â) | 7.98 | 11.69 | 8.68 |
Все измерения проводились от фосфата гуанина.
Измерила торсионные углы гуанина. Сравнила значения углов в А- и В-форме со значениями, приведенными в презентации (таблица 2).
Таблица 2. Сравнение значений углов в А- и В-форме со значениями, приведенными в презентации.
| α | β | γ | δ | ε | ξ | χ | |
| А-форма (RasMol) | -64.11 | 174.80 | 41.70 | 79.09 | 157.76 | -65.12 | -157.20 |
| А-форма (презентация) | -62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| B-форма (RasMol) | -85.87 | 136.35 | 31.14 | 143.40 | 146.77 | -100.52 | -107.92 |
| B-форма (презентация) | -63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
По данным таблицы видно, что значения торсионных углов для В-формы, в частности, первые три угла значительно отличаются от значений в презентации.
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA.
Перевела файлы в старый формат, используя программу remediator.
remediator --old ''XXXX.pdb'' > ''XXXX_old.pdb
Научилась измерять торсионные углы. Данные представлены в таблице 3.
Таблица 3. Торсионные углы нуклеотидов. Запись вида dAB означает изменение угла в разных формах нуклеиновых кислот.
| form | base | α | β | γ | δ | ε | ξ | χ |
| A | G | -51.70 | 174.80 | 41.70 | 79.09 | -147.79 | -75.10 | -157.20 |
| B | G | -29.90 | 136.34 | 31.14 | 143.34 | -140.80 | -160.50 | -98.00 |
| Z | G | 51.93 | 179.00 | -173.80 | 94.90 | -103.60 | -64.80 | 58.70 |
| d AB | G | 21.80 | 38.46 | 10.56 | 64.25 | 6.99 | 85.40 | 59.20 |
| d AZ | G | 103.63 | 4.20 | 215.50 | 15.81 | 44.19 | 10.30 | 215.90 |
| d BZ | G | 81.83 | 42.66 | 204.94 | 48.44 | 37.20 | 95.70 | 156.70 |
| A | A | -51.70 | 174.80 | 41.70 | 79.08 | -147.80 | -75.10 | -157.20 |
| B | A | -29.90 | 136.34 | 31.14 | 143.34 | -140.80 | -160.50 | -98.00 |
| Z | A | none | none | none | none | none | none | none |
| d AB | A | 21.80 | 38.46 | 10.56 | 64.26 | 7.00 | 85.40 | 59.20 |
| d AZ | A | none | none | none | none | none | none | none |
| d BZ | A | none | none | none | none | none | none | none |
| A | T | -51.70 | 174.80 | 41.70 | 79.10 | -147.80 | -75.10 | -157.20 |
| B | T | -29.90 | 136.34 | 31.14 | 143.34 | -140.80 | -160.50 | -97.98 |
| Z | T | none | none | none | none | none | none | none |
| d AB | T | 21.80 | 38.46 | 10.56 | 64.24 | 7.00 | 85.40 | 59.22 |
| d AZ | T | none | none | none | none | none | none | none |
| d BZ | T | none | none | none | none | none | none | none |
| A | C | -51.70 | 174.80 | 41.70 | 79.09 | -147.79 | -75.09 | -157.20 |
| B | C | -29.90 | 136.34 | 31.14 | 143.34 | -140.80 | -160.50 | -97.98 |
| Z | C | -139.50 | -136.77 | 50.87 | 137.60 | -96.50 | 81.97 | -154.30 |
| d AB | C | 21.80 | 38.46 | 10.56 | 64.25 | 6.99 | 85.41 | 59.23 |
| d AZ | C | 87.80 | 311.57 | 9.17 | 58.51 | 51.29 | 157.05 | 2.90 |
| d BZ | C | 109.60 | 273.10 | -19.73 | 5.74 | -44.30 | -242.47 | 56.33 |
Мы обнаруживаем, что в формах A и B сильно различаются торсионные углы δ, ξ и χ. А Z-форма ДНК отличается от форм A и B почти по всем торсионным углам нуклеотидов с гуанином и цитозином.
Для анализа структур нуклеиновых кислот использовала программы find_pair и analyze. По полученным данным составила таблицу 4.
Таблица 4. Анализ структуры нуклеиновых кислот. + - изолированная пара, х - изменение направления цепи
| Нуклеотиды первой цепи | Нуклеотиды второй цепи | Изменения цепи | Вторичная структура РНК | ||
| 1 | G | C | 72 | Акцепторный стебель | |
| 2 | C | G | 71 | ||
| 3 | C | G | 70 | ||
| 4 | G | C | 69 | ||
| 5 | A | U | 68 | ||
| 6 | G | C | 67 | ||
| 7 | G | C | 66 | ||
| 49 | C | G | 65 | T-стебель | |
| 50 | G | C | 64 | ||
| 51 | C | G | 63 | ||
| 52 | G | C | 62 | ||
| 53 | G | C | 61 | x | |
| 39 | U | A | 31 | Антикодоновый стебель | |
| 40 | C | G | 30 | ||
| 41 | G | C | 29 | ||
| 42 | C | G | 28 | ||
| 43 | A | U | 27 | ||
| 44 | G | A | 26 | ||
| 10 | G | C | 25 | D-стебель | |
| 11 | C | G | 24 | ||
| 12 | U | A | 23 | ||
| 13 | C | G | 22 | ||
| 14 | A | U | 8 | ||
| 15 | G | C | 48 | ||
| 16 | U | U | 59 | x | |
| 18 | G | U | 55 | ||
| 19 | G | C | 56 | x | |
| 73 | A | C | 74 | + | |
| 45 | U | A | 9 | + | |
Научилась определять структуру водородных связей. Определила номера нуклеотидов, образующих стебли во вторичной структуре заданной тРНК, неканонические пары оснований в структуре тРНК; определила, есть дополнительные водородные связи в тРНК, стабилизирующие ее третичную структуру (комплементарные пары, не имеющие отношения к стеблям).