Онлайн BLAST
Поиск организма по фрагменту нуклеотидной последовательности
По заданному 300-нуклеотидному фрагменту, используя программу megablast, выбрав нуклеотидный blast (blastn) и указав алгоритм "megablast", определила, какому организму принадлежит данный фрагмент. Он принадлежит Methanococcus voltae, AC записи RefSeq - NC_014222.1, координаты данного фрагмента в записи - от 1145 до 1444, ничего не кодирует.
Поиск гомолога белка человека в слоне
Выбрала любой белок человека, идентификатор которого в Swiss-Prot начинается с той же буквы, что и моя фамилия - DAD1_HUMAN. Получила последовательность данного белка. На сайте ENA провела поиск гомолога этого белка в геноме африканского слона. При поиске на сайте ENA выбрала чекбокс "spliced translated nucleotide search" – это позволило искать белок полностью.
Находка появилась всего одна: ее e-value 9E-57, длина выравнивания 113, identity полученного выравнивания 100%, координаты найденного гена в геноме слона - 1199950-1189654 (обратная цепь), количество интронов в данном гене слона - 1. Ниже привожу выравнивание своей находки.
1 : MetSerAlaSerValValSerValIleSerArgPheLeuGluGluTyrLeuSerSe : 19
||||||||||||||||||||||||||||||||||||||||||||||||||||||||
MetSerAlaSerValValSerValIleSerArgPheLeuGluGluTyrLeuSerSe
1199950 : ATGTCGGCGTCGGTAGTGTCGGTGATCTCGCGGTTCTTAGAAGAGTACTTGAGCTC : 1199896
20 : rThrProGlnArgLeuLysLeuLeuAspAlaTyrLeuLeuTyrIleLeuLeuThrG : 38
||||||||||||||||||||||||||||||||||||||||||||||||||||||||
rThrProGlnArgLeuLysLeuLeuAspAlaTyrLeuLeuTyrIleLeuLeuThrG
1199895 : TACTCCGCAGCGTCTGAAGTTGCTGGACGCGTACCTCCTGTATATACTGCTGACCG : 1199839
39 : lyAlaLeuGlnPheGlyTyrCysLeuLeuValGlyThrPheProPheAsnSerPhe : 56
||||||||||||||||||||||||||||||||||||||||||||||||||||||||
lyAlaLeuGlnPheGlyTyrCysLeuLeuValGlyThrPheProPheAsnSerPhe
1199838 : GGGCGCTGCAGTTCGGTTATTGTCTTCTCGTGGGGACTTTTCCTTTCAACTCTTTC : 1199785
57 : LeuSerGlyPheIleSerCysValGlySerPheIleLeuAla{V} >>>> Targ : 71
||||||||||||||||||||||||||||||||||||||||||{|}
LeuSerGlyPheIleSerCysValGlySerPheIleLeuAla{V}++
1199784 : CTCTCGGGCTTCATCTCTTGTGTGGGAAGCTTCATCCTGGCG{G}gt......... : 1199737
72 : et Intron 1 >>>> {al}CysLeuArgIleGlnIleAsnProGlnAsnLysA : 83
9958 bp {||}||||||||||||||||||||||||||||||||||
++{al}CysLeuArgIleGlnIleAsnProGlnAsnLysA
1199736 : ................ag{TT}TGTCTGAGAATACAGATCAACCCACAGAACAAAG : 1189746
84 : laAspPheGlnGlyIleSerProGluArgAlaPheAlaAspPheLeuPheAlaSer : 101
||||||||||||||||||||||||||||||||||||||||||||||||||||||||
laAspPheGlnGlyIleSerProGluArgAlaPheAlaAspPheLeuPheAlaSer
1189745 : CGGATTTCCAAGGCATCTCCCCGGAGCGAGCCTTTGCTGATTTTCTCTTTGCCAGC : 1189692
102 : ThrIleLeuHisLeuValValMetAsnPheValGly : 113
||||||||||||||||||||||||||||||||||||
ThrIleLeuHisLeuValValMetAsnPheValGly
1189691 : ACCATCTTGCACCTTGTTGTGATGAACTTCGTTGGC : 1189654
Поиск некодирующих последовательностей программой BLAST, сравнение программ BLASTN и MegaBLAST.
Нашла и вырезала в отдельный файл последовательность tRNA-Thr из генома заданной бактерии - Sulfurihydrogenibium azorense.
tRNA complement(683..758) FT /locus_tag="SULAZ_0002" FT /product="tRNA-Thr" >CP001229 CP001229.1 Sulfurihydrogenibium azorense Az-Fu1, complete genome. gcccaggtagctcagtcggcagagcacacccttggtaagggtgaggtcgcgggttcaagt cccgtcttgggcttag
Провела поиск гомологов данной последовательности по всем бактериям, относящимся к тому же порядку, что и моя - Aquificales. Указала в отчете число находок с e-value < 0,001. Поиск провела тремя разными вариантами: a) алгоритмом megablast; b) алгоритмом blastn с параметрами по умолчанию; c) алгоритмом blastn с длиной слова = 7, match/mismatch = 1/-1 (максимально чувствительные параметры, доступные на сайте). В отчете привожу генеалогические деревья данных последовательностей.
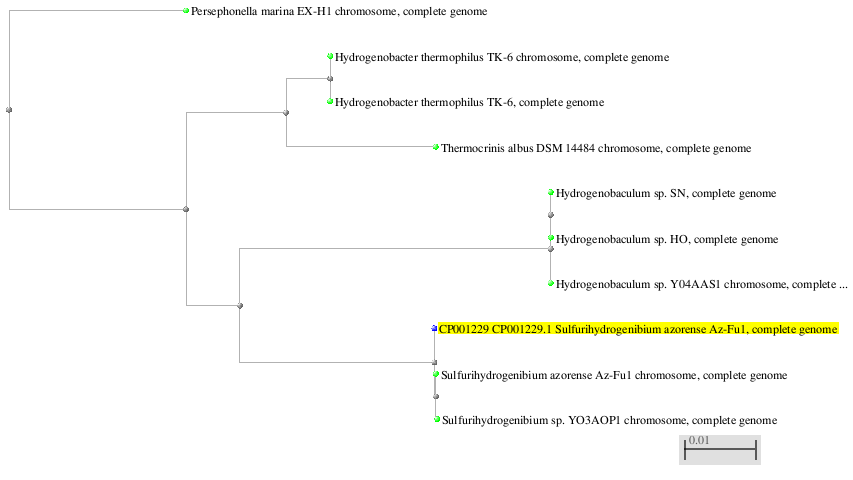
С помощью алгоритма megablast получила 9 результатов. Их генеалогическое дерево представлено на рисунке 1. Мы видим, что полученные находки относятся к двум семействам и пяти родам.
Рисунок 1. Результаты поиска по нуклеотидному банку с помощью алгоритма.
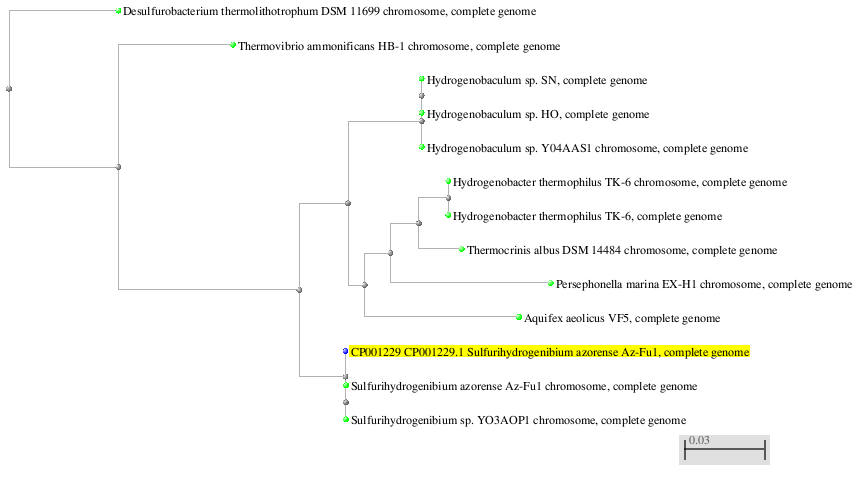
С помощью алгоритма blastn с параметрами по умолчанию получила 12 результатов. Их генеалогическое дерево представлено на рисунке 2. Полученные находки относятся к двум семействам и восьми родам.
Рисунок 2. Результаты поиска по нуклеотидному банку с помощью алгоритма blastn с параметрами по умолчанию.
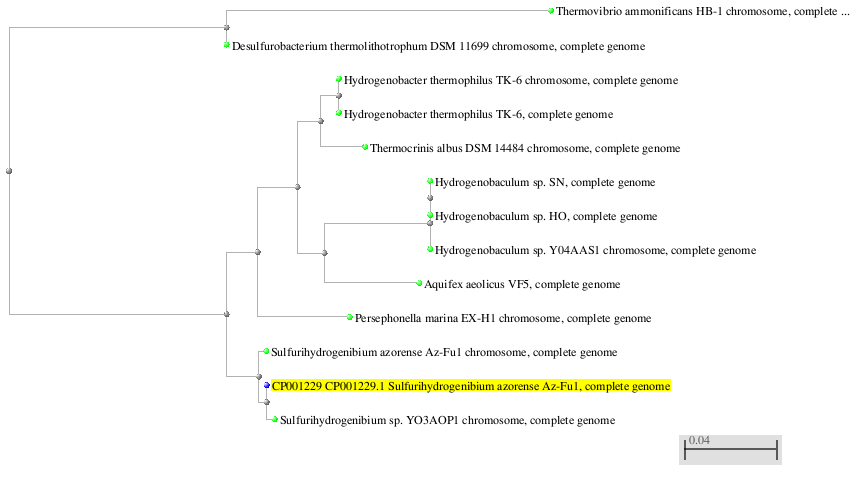
С помощью алгоритма blastn с длиной слова = 7, match/mismatch = 1/-1 получила 12 результатов. Снова полученные находки относятся к двум семействам и восьми родам, но генеалогическое дерево выглядит немного по-другому (рисунок 3).
Рисунок 3. Результаты поиска по нуклеотидному банку с помощью алгоритма blastn с длиной слова = 7, match/mismatch = 1/-1.
Для сравнения результатов поиска выбрала результат, относящийся к Hydrogenobacter thermophilus. В поиске megablast найдена одна последовательность длиной 75, в поиске blastn с параметрами по умолчанию - 6 находок длиной 75, 57, 61 и 56, в поиске blastn с длиной слова = 7, match/mismatch = 1/-1 - 8 последовательностей такой же длины. Результаты различаются выравниваниями, количеством гэпов. По данным результатам можно сказать, что поиск с помощью megablast находит только самые близкие гомологи, а результаты программы blastn подходят для изучения значительно различающихся гомологов.