Эволюционные домены
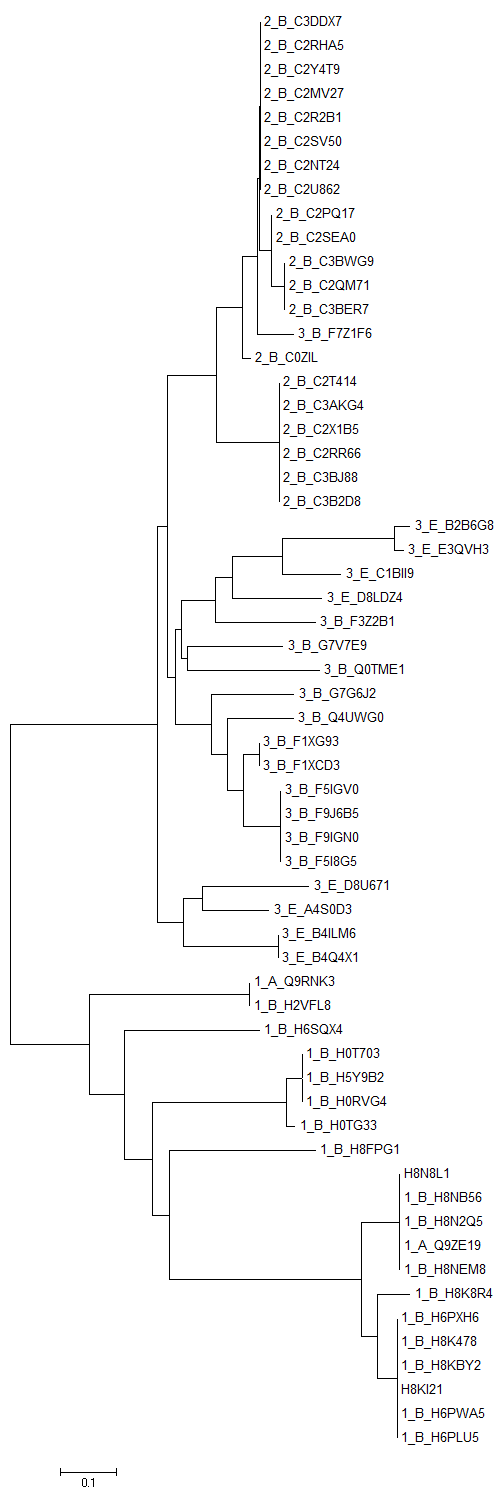
Закодировала доменные архитектуры (1 для MipZ (PF09140); 3 для DUF59 (PF01883) + MipZ (PF09140) + ParA (PF10609); 2 для MipZ (PF09140) + ParA (PF10609)), закодировала сравниваемые таксоны (B - Bacteria, A - Archea, E - Eukaryota). К именам всех последовательностей спереди добавила коды архитектуры и таксона. Построила филогенетическое дерево с помощью программы MEGA. Использовала методы Neighbor-Joining (Рис. 1 и 2) (не использует гипотезу о молекулярных часах и строит неукоренённое дерево) и UPGMA (Рис. 3 и 4) (предполагает молекулярные часы, строит укоренённое дерево) с параметрами по умолчанию.
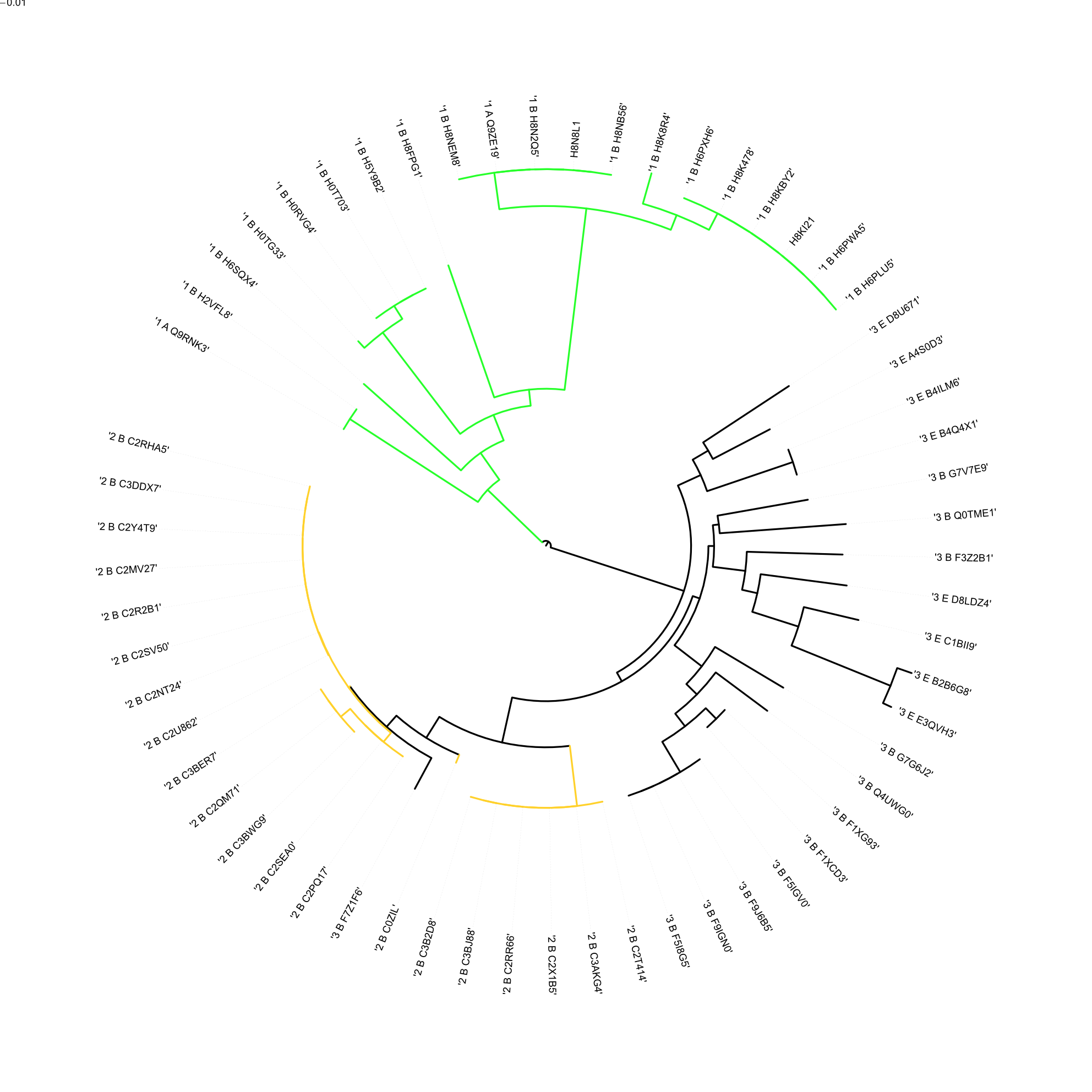
Создала рисунки деревьев с использованием методов выделения ветвей для наглядной демонстрации результата с помощью программы ITOL онлайн (рис. 5 и 6).
Скобочные формулы деревьев, построенных методом Neighbor-Joining (1.nwk) и UPGMA (2.nwk) в формате Newick с длиной ветвей.
На получившихся деревьях видно, что домены из разных доменных архитектур хорошо различаются программой. На получившихся деревьях видно, что однодоменные белки отделились раньше остальных доменов; двухдоменные белки выделились из трехдоменных, которые в свою очередь, имеют более разветвленные ветви, что означает высокую полиморфность этих белков.