Классификация ферментов (EC)
Задание №0
Для трех трансмембранных бета-баррелей и трех трансмембранных альфа-спиральных белков определила параметры, перечисленные в Таблице 1. Использовала базу данных OPM. При определении медианы числа остатков в одном трансмембранном участке производила округление до целого по правилам округления.
Таблица 1. Параметры для трансмембранных белков
| PDB код | Тип (спираль, баррель) | Какая мембрана (внутренняя или внешняя, организм, органелла) | Толщина гидрофобной части мембраны в ангстремах | Медиана числа остатков в одном трансмембранном участке |
| 3m73 | спираль | Бактериальная внутренняя мембрана | 29.1 | 22 |
| 4iar | спираль | Эукариотическая плазматическая мембрана | 33.6 | 24 |
| 1l9b | спираль | Бактериальная внутренняя мембрана | 31.6 | 24 |
| 1a0s | баррель | Бактериальная внешняя мембрана | 23.8 | 9 |
| 1uun | баррель | Бактериальная внешняя мембрана | 40.7 | 10 |
| 2erv | баррель | Бактериальная внешняя мембрана | 24.7 | 11 |
Из таблицы видно, что для альфа-спиралей среднее количество трансмембранных остатков равно 23, а для бета-тяжей - около 10 остатков. Скорее всего, цифры будут сильнее различаться при разной толщине мембраны и разном угле наклона трансмембранных участков.
Задание №1: отбор гомологов
Создала репрезентативную выборку гомологов выданного белка 4JR9. Нашла его номер на сайте Uniprot (P10903). Бактерия (Escherichia coli), из которой взят данный белок, относится к филуму Proteobacteria. На сайте NCBI исключила поиск по данному филуму и по эукариотам, выставила порог e-value равный 1 и количество последовательностей, выдаваемых при поиске 1000. В качестве базы для поиска выбрала RefSeq (если в качестве базы данных выбрать Swissprot, то будет получено всего 11 последовательностей, что представляет собой недостаточно репрезентативную выборку). Далее с помощью таксономического дерева выбрала последовательности для выравнивания. Делала я это вручную, так как раз пять бласт выдавал ошибку при открытии страницы с деревом.Новая репрезентативная выборка
| Репрезентативная выборка гомологов | ||||
| Филум | Порядок | Вид | ||
| Chrysiogenetes | Chrysiogenales | Desulfurispirillum indicum S5 | ||
| Firmicutes | Bacillales | Brevibacillus laterosporus | ||
| Actinobacteria | Actinomycetales | Citricoccus sp. CH26A | ||
| Firmicutes | Selenomonadales | Veillonella parvula | ||
| Actinobacteria | Actinomycetales | Actinoplanes globisporus | ||
| Bacteroidetes | Cytophagales | Belliella baltica DSM 15883 | ||
| Bacteroidetes | Cytophagales | Cyclobacteriaceae bacterium AK24 | ||
| Actinobacteria | Actinomycetales | Tsukamurella paurometabola | ||
| Bacteroidetes | Flavobacteriales | Flavobacterium succinicans | ||
| Deinococcus-Thermus | Thermales | Thermus oshimai | ||
| Proteobacteria | Burkholderiales | Delftia acidovorans | ||
| Firmicutes | Clostridiales | Clostridium carboxidivorans P7 | ||
| Chloroflexi | Sphaerobacterales | Nitrolancea hollandica | ||
| Planctomycetes | Planctomycetales | Zavarzinella formosa DSM 19928 | ||
| Acidobacteria | Acidobacteriales | Terriglobus roseus DSM 18391 | ||
Задание №2: анализ структуры выданного белка
Использовала базу данных OPM для того, чтобы найти в ней выданный белок. На основании полученных данных заполнила таблицу 2.
| PDB ID | Организм | Тип мембраны | TC-код | Угол наклона спиралей (бета-тяжей) к нормали | Количество трансмембранных спиралей (бета-тяжей в бочонке) |
| 4JR9 | Escherichia coli | Внутренняя | 2.A.1 | 8 ± 0° | 12 |
Таблица 2. Описание структуры трансмембранного нитрат/нитрит обменного белка NarK (идентификатор PDB 4JR9, цепь A).
Задание №3: анализ множественного выравнивания трансмембранных белков
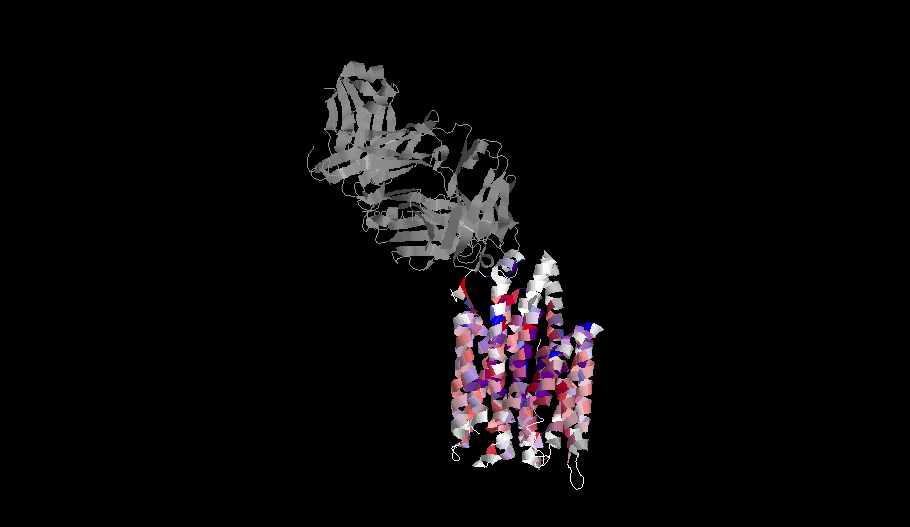
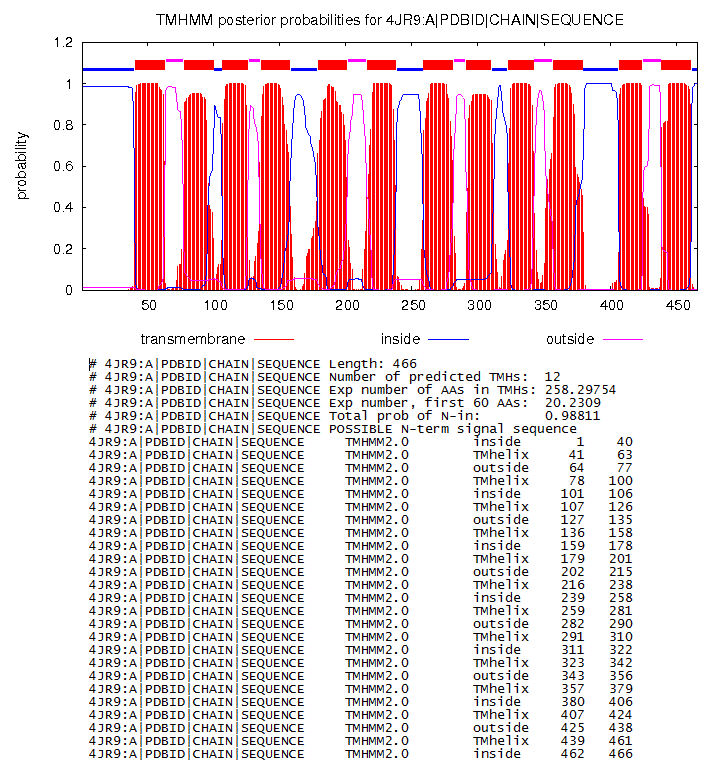
Построила множественное выравнивание отобранных гомологов с помощью программы Muscle. Загрузила множественное выравнивание в программу JalView. Добавила к выравниванию дополнительную аннотацию положения трансмембранных спиралей. Для этого добавила новую пустую строку аннотации и назвала ее "TM_REAL". Переместила исходный белок, для которого есть структура, в верхнюю строку выравнивания. Прикрепила к нему структуру 4JR9, цепь А. Используя появившуюся связь между последовательностью и структурой, пометила участки выравнивания, отвечающие трансмембранным спиралям в белке со структурой в строке-аннотации буквой "М". Добавила к выравниванию предсказание трансмембранных спиралей, выдаваемых программой TMHMM, для гомолога YP_004113709.1 [Desulfurispirillum indicum S5]. Взяла последовательность гомолога и получила для него результат предсказания TMHMM. Создала новую строку аннотации и назвала ее "TM_PREDICTED", после чего нанесла вручную участки, предсказанные TMHMM, в эту строку. Выбрала цветовую схему, позволяющую визуально различать гидрофобные и гидрофильные остатки. Затем установила галочку на "By Conservation" (теперь интенсивность цвета зависит от того, насколько позиция консервативна). Покрутила белок так, чтобы его часть, ориентированная в n-сторону мембраны оказалась сверху, а ориентированная в p-сторону - снизу. Сохранила полученное изображение структуры белка (рисунок 1) и изображение выравнивания (рисунок 2). Предсказание программы TMHMM представлены на рисунке 3.
Рисунок 1.Нитрат/нитрит обменный белок NarK. Цепь А окрашена в цвета выравнивания - более гидрофобные участки - синим, более гидрофильные - красным.
Рисунок 2.Выравнивание репрезентативной выборки. Цветовая схема по гидрофобности, порог консервативности 24%.
Рисунок 3.Предсказание программы TMHMM для белка NarK, цепь А.
По результатам предсказания можно сделать вывод, что программа работает хорошо, так так, во-первых, количество трансмембранных спиралей совпадает, во-вторых, предсказанные и реальные перекрываются довольно сильно, примерно на 70-95%.
Для новой выборки сделала выравнивание (рисунки 4 и 5). На рисунке 5 заряженными аминокислотами считались аспарагиновая и глутаминовая кислота (отрицательно) и гистидин, лизин и аргинин (положительно).
Рисунок 4.Выравнивание репрезентативной выборки. Цветовая схема по гидрофобности, порог консервативности 30%.
Рисунок 5.Выравнивание репрезентативной выборки. Цветовая схема по заряду аминокислот при нейтральном pH, отрицательно заряженные - синие, положительно - красные.
Исходя из полученных данных видно, что заряженные аминокислоты находятся вне мембраны. Правило "positive inside" с помощью данного белка подтвердить нельзя - заряженных аминокислот очень мало.