Подготовка файлов с координатами и топологией
Индекс-файл 1.ndx с группой из одной молекулы этана. et.gro файл с одной молекулой и заданной ячейкой.
Исправленный файл топологии et.top из прошлого задания (изменено количество молекул).
Визуальный анализ
Для каждой из пяти систем проводим конвертацию в pdb-файл. Получаем
et_an.pdb - молекула неподвижна, вращение по СС отсутствует, немного колеблются СН связи.
et_be.pdb - сначала вращение по связям СН, потом и по СС, молекула вращается.
et_nh.pdb - вращение только по связям С-Н.
et_sd.pdb - молекула движется, вращение по С-С связям, углы с водородами не изменяются.
et_vr.pdb - сначала изменяются углы между углеродом и водородом, потом немного вращение по С-С связи.
Сравнение потенциальной и кинетической энергии и распределения длины С-С связи во время моделирования
| Метод | Зависимость энергии | Зависимость длины С-С-связи |
| Андерсена | 
|
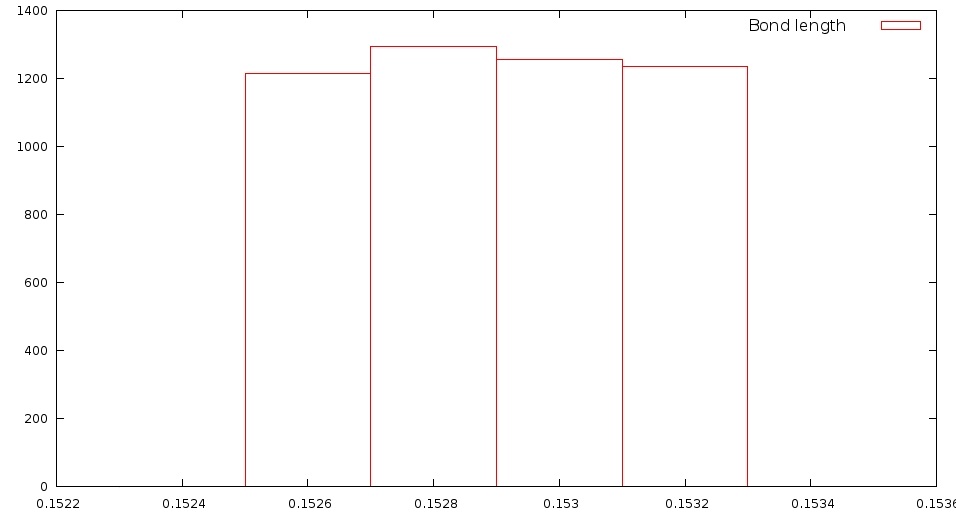
|
| Берендсена | 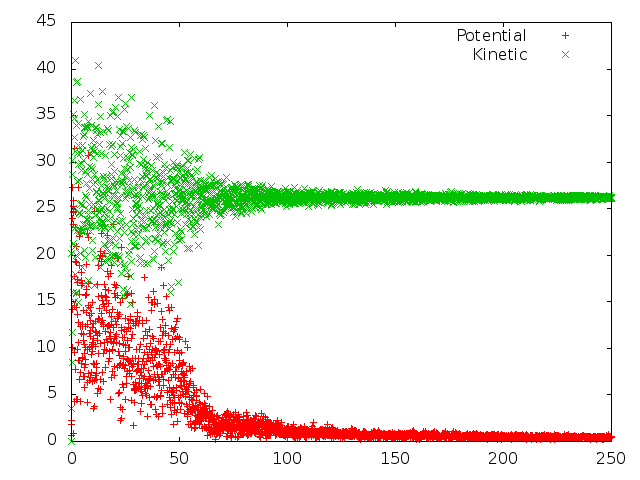
|
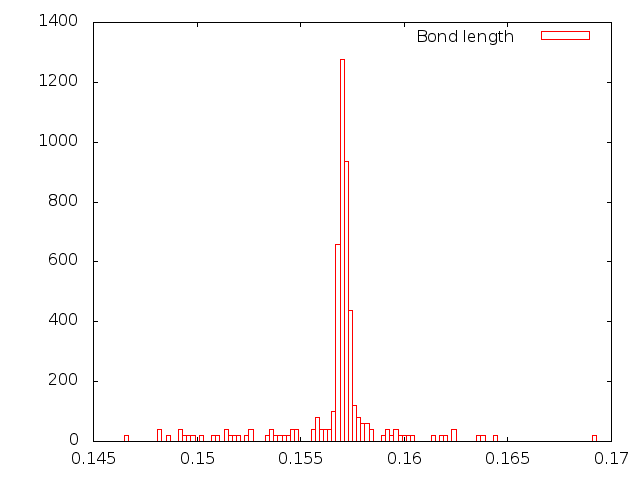
|
| Нуза-Хувера | 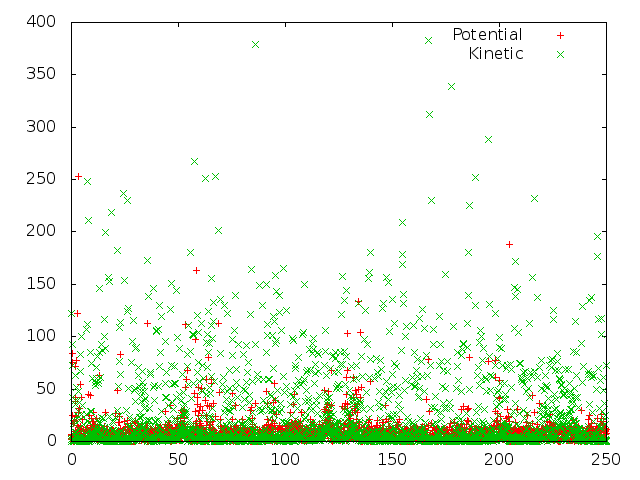
|
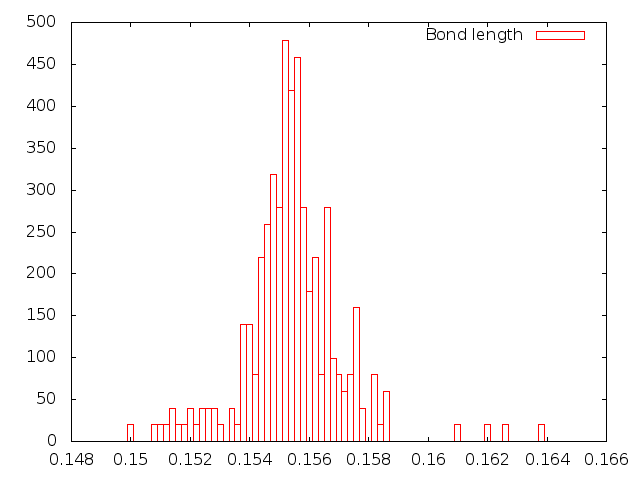
|
| Стохастической мол.динамики | 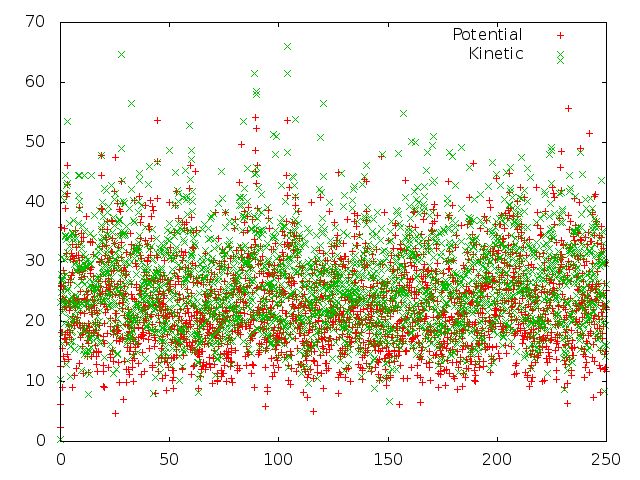
|
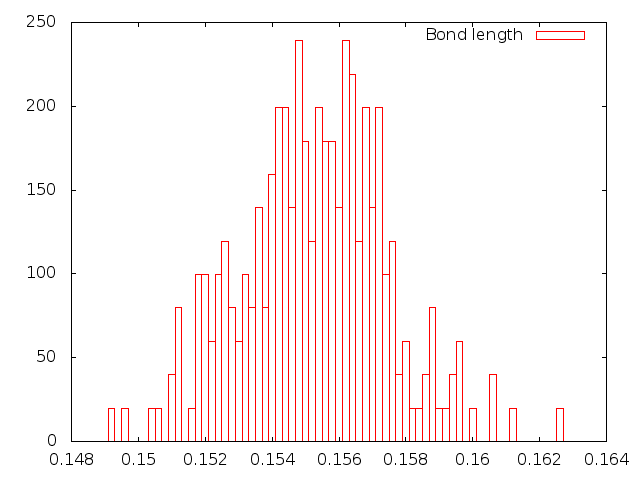
|
| Velocity rescale | 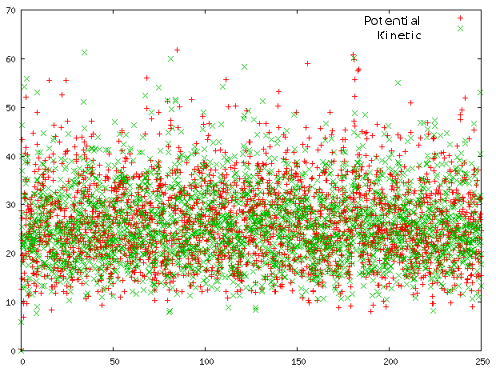
|
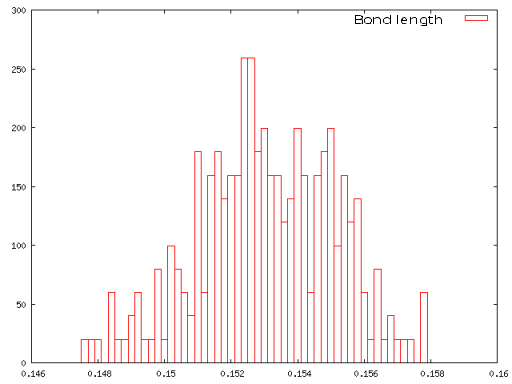
|
Из полученных зависимостей более всего похожи на распределение Больцмана те, что получены методами Velocity rescale и стохастической молекулярной динамики. Однако в Pymol Velocity rescale выглядит адекватнее, без постоянного хаотичного движения молекулы, поэтому лучшим для контроля температуры методом я бы выбрала именно его.