1. Подготовка файлов.
В банке pdb находим SMILES нотацию для NAG: nag.smi
C помощью obgen строим 3D структуру этого сахара в pdb формате:
obgen nag.smi > nag.mol
babel -imol nag.mol -opdb nag.pdb
nag.mol
nag.pdb
Создаем pdbqt файл лиганда:
/home/preps/golovin/progs/bin/prepare_ligand4.py -l nag.pdb -o nag.pdbqt
nag.pdbqt
Создаем pdbqt файл белка:
/home/preps/golovin/progs/bin/prepare_ligand4.py -r seq05.pdb -o seq05.pdbqt
seq05.pdbqt
Файл с параметрами докинга:
vina.cfg
2. Докинг.
vina --config vina.cfg --receptor seq05.pdbqt --ligand nag.pdbqt --out nag_seq05.pdbqt --log nag_seq05.log
babel -ipdbqt nag_seq05.pdbqt -opdb nag_seq05.pdb
Энергии трёх лучших расположений и геометрическая разница между ними
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -4.2 0.000 0.000
2 -4.1 15.660 16.831
3 -4.0 15.427 16.826
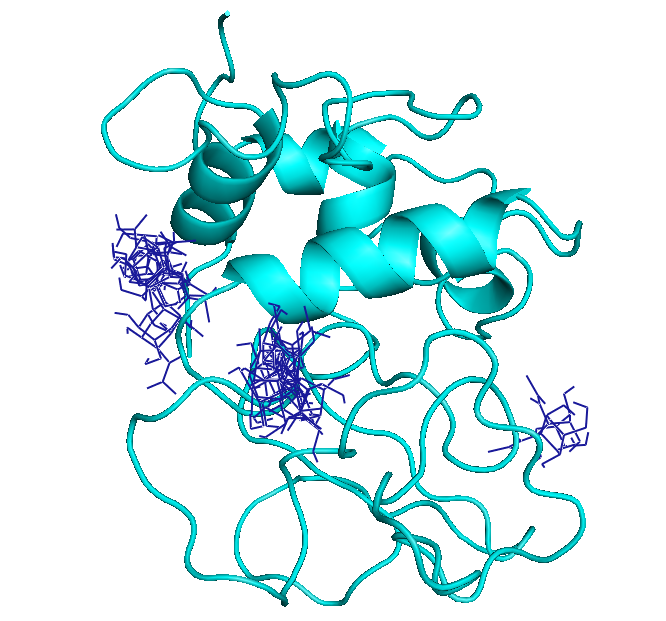
Белок - голубой
Все состояния лиганда - синие
3. Докинг с учетом подвижности боковых радикалов белка
Сначала разобьем белок на две части, подвижную и неподвижную. Для подвижной
части выберем 3 аминокислоты для позиционирования лиганда: Lys142, Gly144, Leu84.
python /usr/share/pyshared/AutoDockTools/Utilities24/prepare_flexreceptor4.py -r seq05.pdbqt -s LYS142_GLY144_LEU84
vina --config vina.cfg --receptor seq05_rigid.pdbqt --flex seq05_flex.pdbqt --ligand nag.pdbqt --log nag_seq05_flex.log --out nag_seq05_flex.pdbqt
Энергии трёх лучших расположений и геометрическая разница между ними
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -4.2 0.000 0.000
2 -4.1 12.820 13.889
3 -4.0 12.674 13.832
По времени докинг шел немного дольше.
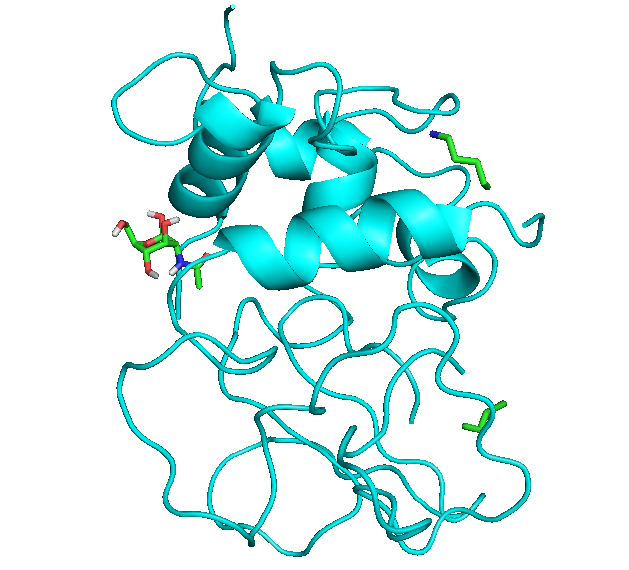
Сравнивая с обычным докингом (пункт 2), видно, что лиганд встроился примерно туда же.
Сравнивая же оба докинга с известной структорой 1lmp с рецептором:
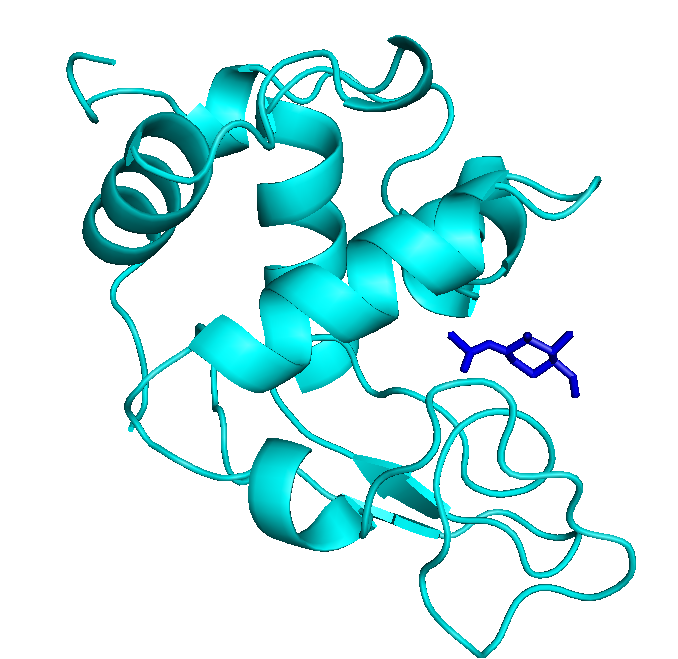
то можно сказать, что обычный докинг тоже предполагал встраивание в такую же область.
Неточности, возможно, связаны с тем, что все таки
это не один и тот же белок, а похожий на него. Так же из-за неточности области в белке, в которой
проводился докинг.
Таким образом, докинг может расположить лиганд наиболее близким образом. Энергия при этом: состояние 14 (-3.4), состояние 18 (-3.4).
4. Изменение радикала NAG
Метильный радикал группы СH3C(=O)NH заменен на:
-H nag_h.pdb
-OH nag_oh.pdb
-NH2 nag_nh2.pdb
-фенил nag_ph.pdb
Обыкновенный докинг
Три лучшие конформации для каждого лиганда с энергиями:
| Конформация |
Радикал |
|||
| H |
OH |
Nh2 |
Ph |
|
|
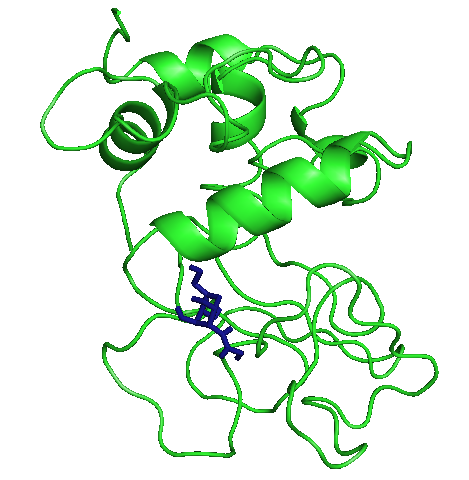
|
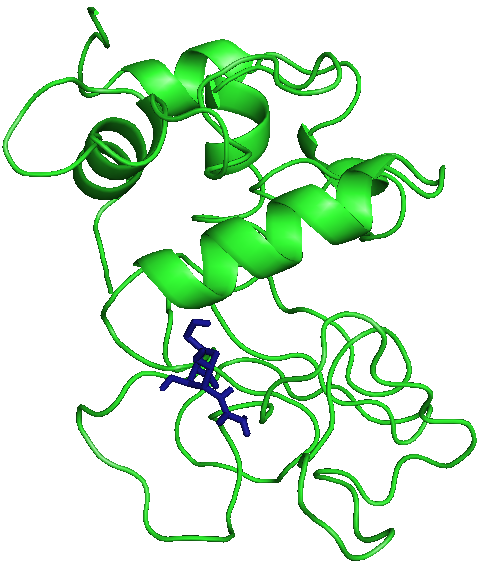
|

|
|
| 1 |
-4.2 |
-4.6 |
-4.7 |
-5.6 |
| 2 |
-3.9 |
-4.5 |
-4.5 |
-5.6 |
| 3 |
-3.8 |
-4.4 |
-4.1 |
-5.3 |
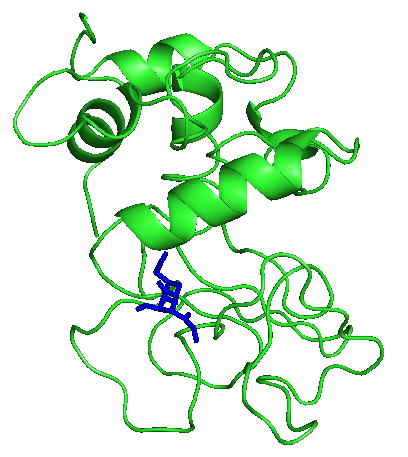
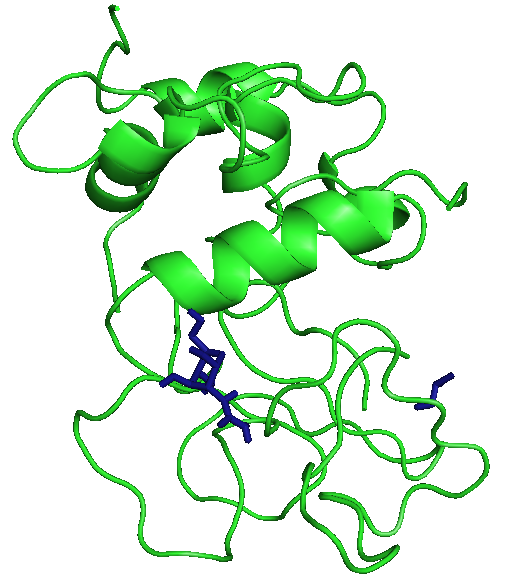
На рисунках отображены расположения лигандов при первой конформации. Видно, что тут только радикал - фенил правильно расположился. Этот же радикал имеет и наибольшие энергии.
Докинг с учетом подвижности боковых радикалов белка
Три лучшие конформации для каждого лиганда с энергиями:
| Конформация |
Радикал |
|||
| H |
OH |
Nh2 |
Ph |
|
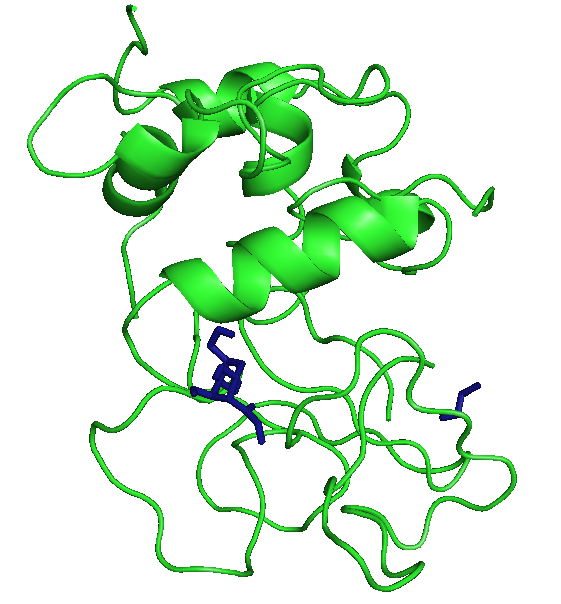
|
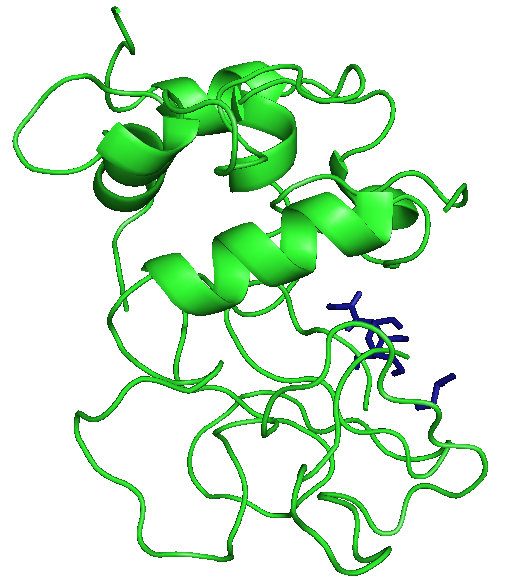
|
|

|
|
| 1 |
-4.1 |
-4.3 |
-4.6 |
-4.9 |
| 2 |
-3.9 |
-4.3 |
-4.6 |
-4.8 |
| 3 |
-3.7 |
-4.0 |
-4.0 |
-4.7 |
В этом случае лучше всего встал ОН, а фенил все равно имеет большие энергии.