Поиск по сходству (BLAST)
Определение таксономии и функции последовательности
В начале для определения таксономии был проведен алгоритм blastn с фильтрами по identity percent (>=60%) и E-value [0;0.0001] для последовательности из практикума 6. В основном были получены последовательности 1 субъединицы цитохром-C оксидазы (CO1), а первая 21 последовательность относилась к роду Polycirrus кольчатых червей. Из них 4 первые c самыми большими баллами относились к виду Polycirrus medusa.
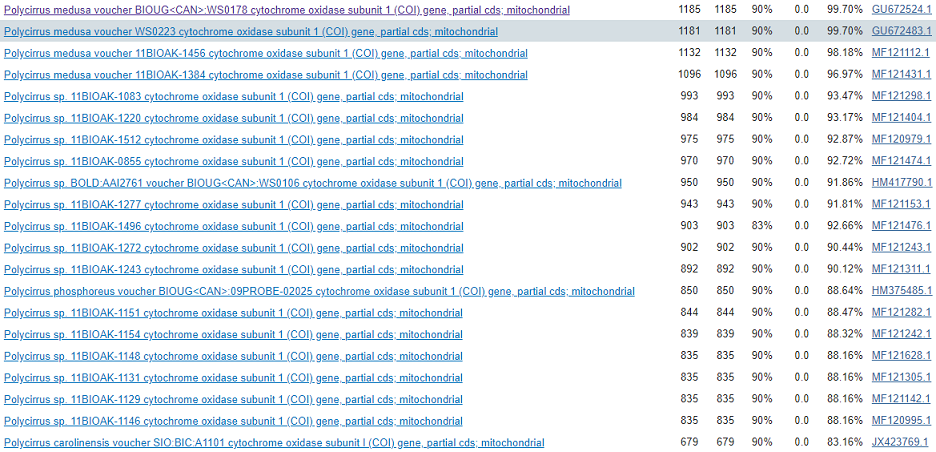
Так как последовательность кодирует белок, был проведен blastx для подтверждения функции этого белка. Как и ожидалось, больше всего последовательностей относились к CO1.

При проведении megablast, он показал похожие результаты с blastn. Первыми 4-мя последовательностями с самыми большими баллами (>1100), процентом идентичности (>96%) и E-value равной 0 принадлежали виду Polycirrus medusa.
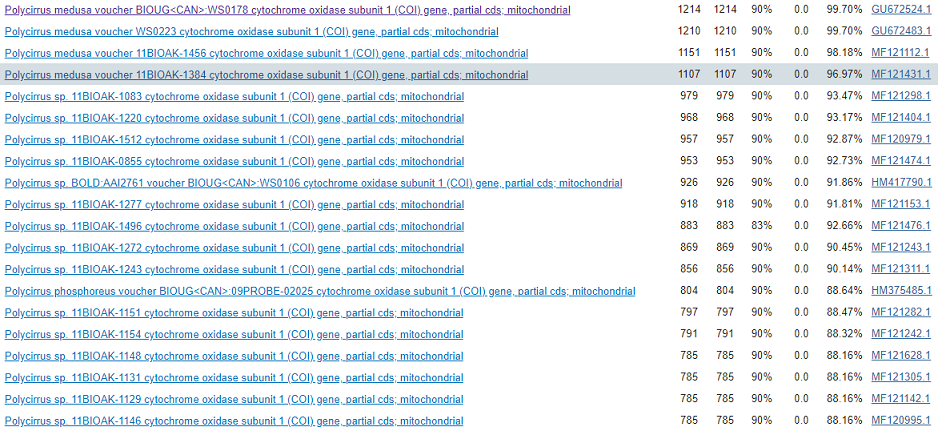
Таким образом, можно утверждать, что данная последовательность являлась фрагментом гена цитохром-С оксидазы 1 из Polycirrus medusa.
Сравнение списков находок нуклеотидных последовательностей
В этом задании было проведено сравнение выдачи алгоритмов blastn (по-умолчанию и чувствительного) и megablast. Было использовано 2 чувствительных blastn, различающиеся параметрами match/mismatch и gap penalty. Во поисках применялись фильтры percent identity [60;99], E-value [0;0.001]. Для Polycirrus medusa также применялось ограничение по семейству Terebellidae, не включая род Polycirrus.
Таблица 1.
Параметры 5 запусков blast
| Алгоритм |
Max target sequences |
Word size |
Match/Mismatch Scores |
Gap Costs |
| blastn (default) |
1000 |
11 |
2,-3 |
Existence: 5
Extension: 2 |
| blastn 1 |
1000 |
7 |
2,-3 |
Existence: 5
Extension: 2 |
| blastn 2 |
1000 |
7 |
1,-1 |
Existence: 5
Extension: 2 |
| blastn 3 |
1000 |
7 |
1,-1 |
Existence: 0
Extension: 2 |
| megablast |
1000 |
28 |
1,-2 |
Linear |
Для вируса из предыдущего практикума был использован фильтр по таксономии viruses. Так как ни одной находки, подходящей под фильтры не было найдено, то был сделан также запрос без фильтров.
Таблица 2.
Количество находок
| Алгоритм |
Polycirrus medusa |
Acholeplasma virus MV-L1 (с фильтрами) |
Acholeplasma virus MV-L1 (без фильтров) |
| blastn (default) |
68 |
0 |
103 |
| blastn 1 |
68 |
0 |
114 |
| blastn 2 |
66 |
0 |
191 |
| blastn 3 |
62 |
0 |
639 |
| megablast |
14 |
0 |
1 |
Изменение параметров при работе с Polycirrus medusa не сильно повлияло на количество находок. Уменьшение количества находок (от blastn 1 к blastn 3) может быть в следствие того, что только топ 20 организмов отображается при использовании таксономического фильтра (организмы ниже в листе могли иметь больше хитов). Не один из алгоритмов не нашел последовательности, подходящие к фильтрам при поиске по вирусу Acholeplasma virus MV-L1, вероятно, из-за высокой специфичности данной кодирующей последовательности у этого вируса (Также следует отметить очень низкое покрытие - при самом чувствительном blastn 3 оно составило всего лишь 53%). При использовании фильтра Viruses были обнаружены 2 вируса с очень маленькими баллами (< 55). Даже использовав самую короткую CDS последовательность результат оказался примерно тот же, что подтверждает версию о специфичности этих CDS для этого вируса. Взяв другой вирус этого же семейства - Inoviridae sp. isolate ctbh45 - и проведя для одного из CDS поиск по стандартному blastn с фильтрами был найден 1 вирус из того же семейства (Inoviridae sp. isolate ctcf41). Megablast без фильтров опять указал на единственную заданную последовательность. Blastn 3 c фильтрами обнаружил сходство в одной из бактерий (на которой вирус вероятно паразитирует) и в других, совсем не связанных с вирусом организмах.

Таким образом, при поиске без фильтра по вирусу лучше всего видна разница при использовании разных параметров. Так изменение длины слова не всегда значительно увеличивает количество находок, в то время как дополнительное изменение gap costs и match/mismatch score этому способствуют. Однако эти дополнительные находки не являются значимыми, так как не имеют необходимой E-value, Percent identity, Score и/или Query coverage.
Проверка наличия гомологов трех белков в неаннотированном геноме
Были взяты 3 консервативных для эукариот белка из UniProt. Был выбран гистон H3.2, тубулин (бета-цепь) и субъединица 1 цитохрома C.
Гистоны играют важнейшую роль в процессе репарации, транскрипции, репликации и свертывании ДНК у эукариот; Тубулин является основным компонентом микротрубочек, которые являются основой цитоскелета эукариот; Цитохром C оксидаза является ключевым белком в аэробном дыхании как у эукариот, так и у прокариот. С помощью команд из EMBOSS были обнаружены гомологичные находки в сборке генома Amoeboaphelidium protococcarum. Скрипт:
seqret sw:H32_HUMAN -stdout H32_HUMAN.fasta
seqret sw:TBB5_HUMAN -stdout TBB5_HUMAN.fasta
seqret sw:COX1_DROME -stdout COX1_DROME.fasta
makeblastdb -in X5.fasta -dbtype nucl -out pr8
tblastn -query H32_HUMAN.fasta -db pr8 -out H32_HUMAN.txt
tblastn -query TBB5_HUMAN.fasta -db pr8 -out TBB5_HUMAN.txt
tblastn -query COX1_DROME.fasta -db pr8 -out COX1_DROME.txt
В результате были получены файлы с информацией о находках: H32_HUMAN, TBB5_HUMAN, COX1_DROME и была составлена таблица 3.
Таблица 3.
Находки гомологов в геноме Amoeboaphelidium protococcarum
| |
H32_HUMAN |
TBB5_HUMAN |
COX1_DROME |
| Количество находок |
10 |
6 |
5 |
| Количество находок c E-value < 0.001 |
6 |
5 |
1 |
| Лучшая находка |
scaffold-126, scaffold-104 |
unplaced-665 |
unplaced-887 |
| Score лучшей находки, bits |
245 |
763 |
226 |
| Е-value лучшей находки |
1e-74 |
0.0 |
5e-63 |
| Identity лучшей находки, % |
93 |
85 |
53 |
| Покрытие лучшей находки, % |
100 |
96,17 |
44,92 |
В геноме Amoeboaphelidium protococcarum с большой вероятностью существуют гомологичные домены гистону и тубулину человека, исходя из E-value и Identity находок. Белок COX1 дрозофиллы имеет меньшее количество значимых находок и identity у лучшей из них не велико. К тому же покрытие лучшей находки составило всего 44,92%. Поэтому, хотя в выравнивании и видны гомологичные блоки и E-value совсем не большая, о гомологичности нужно говорить с осторожностью (рисунок ниже).

Поиск гена белка в контиге
Для этого задания был взят скэффолд из сборки из предыдущего практикума с AC:RRCB01000092.1 из GenBank (Taeniopygia guttata isolate Black17 scaffold_2_arrow_ctg1). Его длина составила 39859 пар оснований. С помощью алгоритма blastx по базе данных refseq_protein и таксону Animalia (taxid:33208) были получены находки на рисунке ниже.

Как видно, большую часть находок занимают последовательности обонятельных рецепторов 14J1-like и 14A16-like. При просмотре выравнивания с самым большим счетом видна абсолютная идентичность аминокислот в обоих последовательностях. Длина гена совпадает с таковой в выравнивании, то есть он целиком есть в скэффолде.
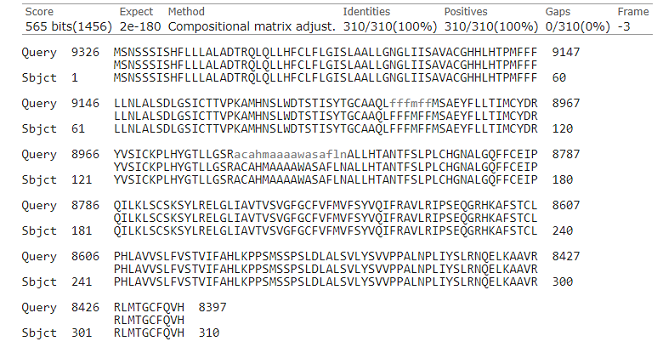
Таким образом, можно утверждать, что в скэффолде с большой вероятностью содержится ген, кодирующий белок olfactory receptor 14J1-like (Sequence ID: XP_030117861.1).
Назад
На главную






