Работа в базе данных UniProt. Сравнение белка предполагаемая Zn-зависимая пептидаза бактерии G.sulfurreducens PCA с белками-ортологами бактерий рода Geobacteraceae
На занятиях по информатике мы уже успели поработать с такими базами данных как GBK, EMBL (базы данных нуклеиновых последовательностей) и PubMed (база данных публикаций). В этот раз наше задание связано с использованием базы данных последовательностей белков Uniprot.
Для начала нужно было определить идентификатор белка в этой базе данных с помощью функции ID mapping: в поле для поиска я ввела идентификатор в Refseq, и на выходе получила ссылку на страницу моей пептидазы в базе данных.
Далее я воспользовалась обычным поиском (Search), чтобы найти его ортологов. Начала я просто с поиска по названию белка: Peptidase lipoprotein. Результатом такого запроса оказалось 21,306 организмов, из которых в базе данных Swiss-Prot лежит 994 записи и в TrEMBL - 20,312. Тогда я рещила сократить выборку до пептидаз из семейства M48: Peptidase lipoprotein, M48 family.
По этому запросу нашлось 396 организмов, из которых 1 в Swiss-Prot и 395 в TrEMBL.
Тогда я вспомнила определение ортологов. Ортологи - это гомологичные последовательности, разделившиеся в результате видообразования и выполняющие сходные функции. Раз схожие функции, значит класс белков должен совпадать, а разделение при видообразовании означает, что эти гомологи могут найтись в пределах одного рода. В итоге для расширенного поиска (Advanced search) подходят следующие поля: protein name [DE] - Peptidase lipoprotein, и Taxonomy [OC] - Geobacteraceae. В итоге Uniprot сфорфировал следующий запрос:
name:"Peptidase lipoprotein" AND taxonomy:Geobacteraceae
По данному запросу найдено 6 результатов, среди которых пептидаза бактерии G.sulfurreducens PCA. Для дальнейшей работы я выбрала следующие два белка: пептидазу B5EB93 бактерии G.bemidjiensis Bem и пептидазу Q39WA0 бактерии G.metallireducens GS-15.
Следующим этапом было получение файла с информацией о белке из Uniprot. Для этого я воспользовалась пакетом EMBOSS, установленном в PUTTY. Чтобы получить файлы, были сделаны следующие запросы:
entret uniprot:Q74D82
entret uniprot:Q39WA0
entret uniprot:B5EB93
После получения информационных файлов о белках, было проведено их "исследование", результаты которого можно увидеть в таблице 1.
Таблица 1. Разбор Uniprot-файлов белков бактерий G.sulfurrreducens PCA, G.bemidjiensis Bem и G.metallireducens GS-15 (получено с помощью базы данных Uniprot).
| Метка поля | Содержание | |||
| Пептидаза G.sulfurrreducens PCA | Пептидаза G.bemidjiensis Bem | Пептидаза G.metallireducens GS-15 | ||
| Идентификатор записи | ID | Q74D82_GEOSL | Q39WA0_GEOMG | B5EB93_GEOBB |
| Код доступа первый ("Accession number") | AC | Q74D82 | Q39WA0 | B5EB93 |
| Код(ы) доступа остальные) | AC | - | - | - |
| Дата создания документа | DT | 05-JUL-2004 | 22-NOV-2005 | 14-OCT-2008 |
| Дата последнего исправления аннотации | DT | 13-NOV-2013, entry version 69 | 13-NOV-2013, entry version 61 | 13-NOV-2013, entry version 34 |
| Название (краткое описание) белка | DE | Peptidase lipoprotein, M48 family | Peptidase lipoprotein, M48 family | Peptidase lipoprotein, M48 family |
| Название организма | OS | Geobacter sulfurreducens (strain ATCC 51573 / DSM 12127 / PCA) | Geobacter metallireducens (strain GS-15 / ATCC 53774 / DSM 7210) | Geobacter bemidjiensis (strain Bem / ATCC BAA-1014 / DSM 16622) |
| Таксономия | OC | Bacteria; Proteobacteria; Deltaproteobacteria; Desulfuromonadales; Geobacteraceae; Geobacter | Bacteria; Proteobacteria; Deltaproteobacteria; Desulfuromonadales; Geobacteraceae; Geobacter | Bacteria; Proteobacteria; Deltaproteobacteria; Desulfuromonadales; Geobacteraceae; Geobacter |
| Название локуса гена | GN | GSU1437 | Gmet_1238 | Gbem_3390 |
| Номер публикции (брать только 1ю) | RX | PubMed=14671304; DOI=10.1126/science.1088727 | - | - |
| Автор(-ы) публикации | RA | Methe B.A., Nelson K.E., Eisen J.A., Paulsen I.T., Nelson W., Heidelberg J.F., Wu D., Wu M., Ward N., Beanan M.J., Dodson R.J., Madupu R., Brinkac L.M., Daugherty S.C., DeBoy R.T., Durkin A.S., Gwinn M., Kolonay J.F., Sullivan S.A., Haft D.H., Selengut J., Davidsen T.M., Zafar N., White O., Tran B., Romero C., Forberger H.A., Weidman J., Khouri H., Feldblyum T.V., Utterback T.R., Van Aken S.E., Lovley D.R., Fraser C.M. | Copeland A., Lucas S., Lapidus A., Barry K., Detter J.C., Glavina T., Hammon N., Israni S., Pitluck S., Di Bartolo G., Chain P., Schmutz J., Larimer F., Land M., Kyrpides N., Ivanova N., Richardson P. | Lucas S., Copeland A., Lapidus A., Glavina del Rio T., Dalin E., Tice H., Bruce D., Goodwin L., Pitluck S., Kiss H., Brettin T., Detter J.C., Han C., Kuske C.R., Schmutz J., Larimer F., Hauser L., Kyrpides N., Lykidis A., Lovley D., Richardson P.; Land M. |
| Название публикации | RT | "Genome of Geobacter sulfurreducens: metal reduction in subsurface environments." | "Complete sequence of Geobacter metallireducens GS-15." | "Complete sequence of Geobacter bemidjiensis BEM." |
| Журнал | RL | Science 302:1967-1969(2003) | Submitted (OCT-2005) to the EMBL/GenBank/DDBJ databases | Submitted (JUL-2008) to the EMBL/GenBank/DDBJ databases |
| Чем обосновано существование белка | PE | 1: Evidence at protein level | 3: Inferred from homology | 3: Inferred from homology |
| Ссылка (-и) на базу 3D структур PDB | DR | PDB; 3C37; X-ray; 1.70 A; A/B=21-264 | - | - |
Как видно из таблицы 1, существование пептидаз B5EB93 бактерии G.bemidjiensis Bem и Q39WA0 бактерии G.metallireducens GS-15 было выявлено только при предположении, что у них должен быть такой же фермент, как и у их сородича, так что никаких глубоких исследований делать без крайней нужды не стали. Поэтому мною было проведено небольшое исследование на различие их строения. Обобщение результатов можно увидеть в таблице 2.
Таблица 2. Сравнение последовательностей белков бактерий G.sulfurrreducens PCA, G.bemidjiensis Bem и G.metallireducens GS-15 (получено с помощью базы данных Uniprot).
| Метка поля | Содержание | |||
| Пептидаза G.sulfurrreducens PCA | Пептидаза G.bemidjiensis Bem | Пептидаза G.metallireducens GS-15 | ||
| Комментарии о функции | DR(GO) | metalloendopeptidase activity; proteolysis / металлопептидазная активность; протеолиз |
metalloendopeptidase activity; proteolysis / металлопептидазная активность; протеолиз |
metalloendopeptidase activity; proteolysis / металлопептидазная активность; протеолиз |
| Местонахождение в клетке | DR(GO) | membrane / мембрана |
membrane / мембрана |
membrane / мембрана |
| Особенность: связывание металла | CC | Binds 1 zinc ion per subunit (By similarity); связывается с валином 125, глицином 129, аспарагином 181 и пролином 227 | Binds 1 zinc ion per subunit (By similarity); связывается с гистидином 125, гистидином 129, глутаминовой к-той 181 и гистидином 227 | Binds 1 zinc ion per subunit (By similarity); связывается с гистидином 126, аланином 130, аспарагином 182 и пролином 228 |
| Длина аминокислотной последовательности | 264 | 265 | 268 | |
| Последовательность 2-ой α-цепи | KELGNKFAVEIEKQQQPV (12 с ort1), KELGNKFAVEIEKQQQPV (13 с ort2) | KELGTKFAAEIEKQYPVY | KQLGDKFAVEIEKQHKVV | |
Скачать данные таблицы можно здесь.
Из таблицы 2 видно, что все три белка между собой очень похожи: выполняют одинаковые функции, имеют одинаковое местонахождение, один центральный атом и др. ДЛя этого практикума нам предлагалось раскрыть несколько вопросов о белке, мой выбор пал на следующие:
- В какой части бактериальной клетки локализован Ваш белок? На какой участок вашего белка Вы бы стали воздействовать,чтобы помешать его правильной локализации в клетке?
Все пептидазы встроены в мембрану, что можно было предположить, исходя из расположения его гидрофобных кластеров. Поэтому чтобы нарушить его локализацию, я бы заменила большую часть этого гидрофобного "ядра" на гидрофильные цепочки, тогда белку будет невыгодно прятаться в липидном слое, и он уйдёт в цитоплазму.
- Какие аминокислотные остатки участвуют в образовании активного центра? (перечислите № позиций и полные названия)
В активный центр пептидаз входит ион металла Zn2+ и связывающие его аминокислоты. У Q74D82 это валин 125, глицин 129, аспарагин 181 и пролин 227 (согласно Uniprot). Так как для двух других белков PDB-файлов нет, то я предположила, что у них связывающие аминокислоты будут иметь примерно такие же координаты: у Q39WA0 - гистидин 125, гистидин 129, глутаминовая к-та 181 и гистидин 227 (координаты я выбрала такие же, так как длина цепи отличается всего на одну аминокислоту), а у B5EB93 - гистидин 126, аланин 130, аспарагин 182 и пролин 228 (у этого белка я сместила номера на единицу вправо. Почему? Объяснено в разделе с α-спиралями.)
- Какие ионы связываются с белком?
Ионы цинка - Zn2+.
- Получите последовательность 2-й альфа-спирали, используя команду seqret пакета EMBOSS.
Извлечь аминокислотную последовательность можно с помощью команды seqret. Сначала для удобства я извлекла всю последовательность белка:
seqret mybelok
Далее я извлекла конкретно последовательность аминокислот, соответствующую 2-ой α-спирали (её координаты я посмотрела в файле PDB - они оказались 39..56). Для этого есть подфункция -sask1:
seqret -sask1 seq
Begin at position [start]: 39
End at position [end]: 56
output sequence(s) [q74d82_geosl.fasta]: seqhelixАналогичным способом я получила последовательности α-спиралей двух других белков, предположив, что координаты их начал и концов совпадают. В случае с Q39WA0 это так и оказалось - сразу обнаружилось 12 совпадений по аминокислотам (из 18 возможных) - совпадающие аминокислоты выделена в таблице красным. А вот в случае с B5EB93 я заметила, что если сместить координаты на единицу вправо, то совпадений станет больше - 13 из возможных 18 (выделены синим). Возможно, это смещение обусловлено встраиванием лишней аминокислоты до последовательности альфа-спирали, ведь у этого онтолога длина цепи увеличилась уже на 4 аминокислоты.
Работа в базе оперонов DOOR2. Предсказание оперонов, включающих белки пептидаза и АТФ-синтаза
Следующим заданием было знакомство с базой оперонов DOOR2. Оперон - это последовательность генов, лежащих друг за другом на цепи ДНК и считывающихся совместно в одну молекулу мРНК, образуя "заготовку" для клеточного компонента. В первом семестре я уже делала предположения о составе оперона АТФ-синтазы. Мои размышления основывались на со- и разнонаправленности генов. Сейчас мне нужно будет сравнить свои предположения с предположениями учёных, создавших DOOR2.
Сначала я нашла оперон АТФ-синтазы с помощью следующего запроса: geobacter sulfurreducens | atpA. По этому запросу было найдено 2,886 записей, интересующая меня оказалась на 13-ом месте. оказалось, что в базе данных DOOR2 оперон АТФ-синтазы содержит всего 5 генов из предполагаемых мною 11 (на рисунке 1 изображены, какие опероны определены для этого белка).

Рисунок 1. Опероны, соответствующие 11 белкам, определённым мною как оперон АТФ-синтазы. Рисунок получен с помощью базы данных DOOR2. Расширенную версию можно посмотреть тут
Согласно DOOR2, опероном АТФ-синтазы является оперон под номером 28646 (входящие в него гены перечислены в таблице 3), в то время как границы "моего оперона" распространяются ещё и на опероны 28645, 1428446, 1428447 и 28647. Определить однозначно, кто прав, невозможно, но у DOOR2 довольно высокий уровень предсказания: ~90%. И тем не менее, я удивлена, что atpX и atpC получили отдельные опероны, ведь они явно являются частями этой синтазы - B'-субъединица АТФ-синтазы Fo и ε-субъединица АТФ-синтазы типа FoF1 соответственно. Так что мне кажется, что здесь допущена погрешность, и в окончательный оперон входят 7 генов: atpX, atpF, atpH, atpA, atpG, atpD, atpC.
Таблица 3. Гены в опероне 28646 - предполагаемом опероне АТФ-синтазы бактерии G.sulfurrreducens PCA.
| Ген | Координаты гена в геноме (begin..end) | Длина в аминокислотных остатках | Продукт гена | Перевод названия продукта на русский язык |
| atpF | 123982..124602 | 621 | ATP synthase F0 subunit B | B-субъединица АТФ-синтазы Fo |
| atpH | 124599..125141 | 543 | F0F1 ATP synthase subunit delta | δ-субъединица АТФ-синтазы FoF1 |
| atpA | 125150..126661 | 1,512 | F0F1 ATP synthase subunit alpha | α-субъединица АТФ-синтазы FoF1 |
| atpG | 126683..127546 | 864 | F0F1 ATP synthase subunit gamma | γ-субъединица АТФ-синтазы FoF1 |
| atpD | 127604..129016 | 1,413 | F0F1 ATP synthase subunit beta | β-субъединица АТФ-синтазы FoF1 |
Также в задании предлагалось найти оперон своего белка. По своему запросу: Geobacter sulfurreducens PCA | GSU1437 | M48 family peptidase, я получила 18 результатов, интересующая меня была 4-ой в очереди - ID 28921. В этот оперон, помимо гена пептидазы, входят ещё 6 генов. Информация о них представлена в таблице 4, а взаимное расположение изображено на рисунке 2.
Таблица 4. Гены в опероне 28921, содержащем Zn-зависимую пептидазу бактерии G.sulfurrreducens PCA.
| Ген | Координаты гена в геноме (begin..end) | Длина в аминокислотных остатках | Продукт гена | Перевод названия продукта на русский язык |
| GSU1432 | 1569651..1571372 | 1,722 | hypothetical protein | гипотетический белок |
| GSU1433 | 1571369..1572985 | 1,617 | peptide ABC transporter periplasmic peptide-binding protein | периплазматический белок из надсемейства ABC, участвующий в транспорте белков |
| GSU1434 | 1572987..1573967 | 981 | peptide ABC transporter permease | транспортная пермеаза из надсемейства ABC |
| GSU1435 | 1573971..1574819 | 849 | peptide ABC transporter permease | транспортная пермеаза из надсемейства ABC |
| GSU1436 | 1574843..1575367 | 525 | hypothetical protein | гипотетический белок |
| GSU1437 | 1575388..1576182 | 795 | peptidase lipoprotein, M48 family | липопротеиновая пептидаза из семейства M48 |
| GSU1438 | 1576185..1577063 | 879 | hypothetical protein | гипотетический белок |
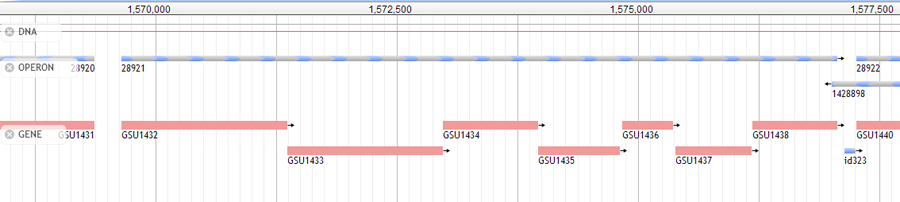
Рисунок 2. Оперон 28921, соответствующий белку Zn-зависимой пептидазы и ещё 6 белкам. Рисунок получен с помощью базы данных DOOR2. Расширенную версию можно посмотреть тут
Дата последнего изменения: 16.03.2014
