Определение таксономии и функции нуклеотидной последовательности
Для выполнения данного задания была выбрана консенсусная последовательность из практикума 6. Поиск проводился программой blastn (для поиска сколько–нибудь похожих нуклеотидных последовательностей). Был изменен только один параметр — Word Size на 7 (остальные параметры оставил неизменными).
Что получили?
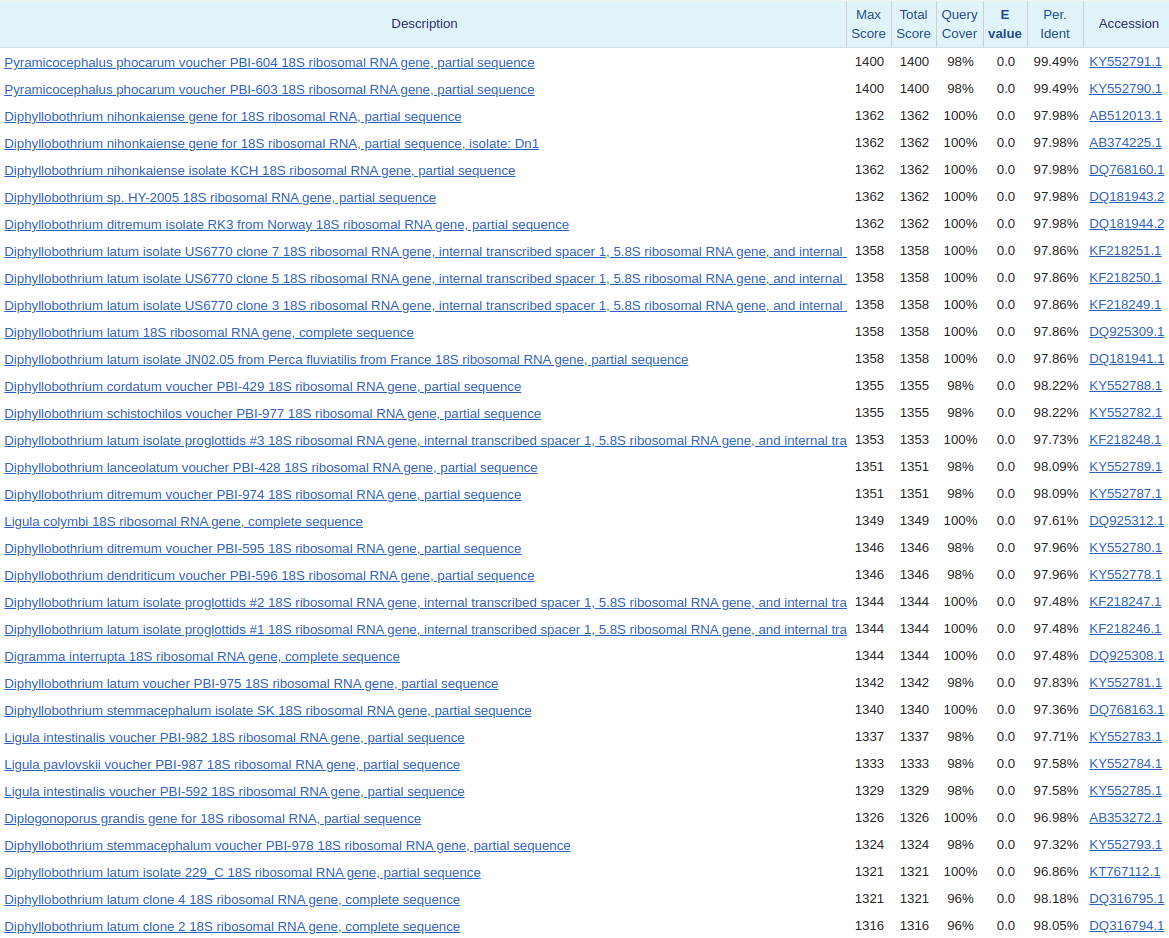
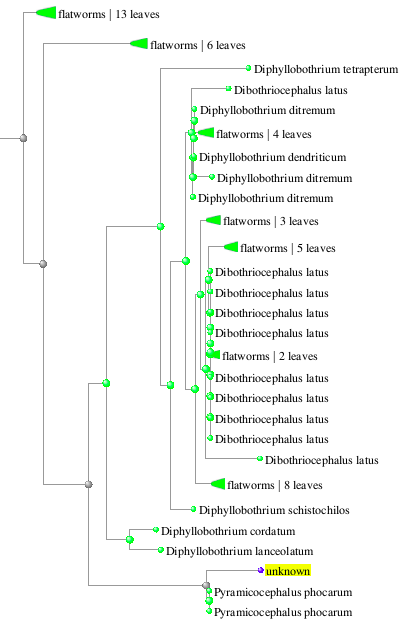
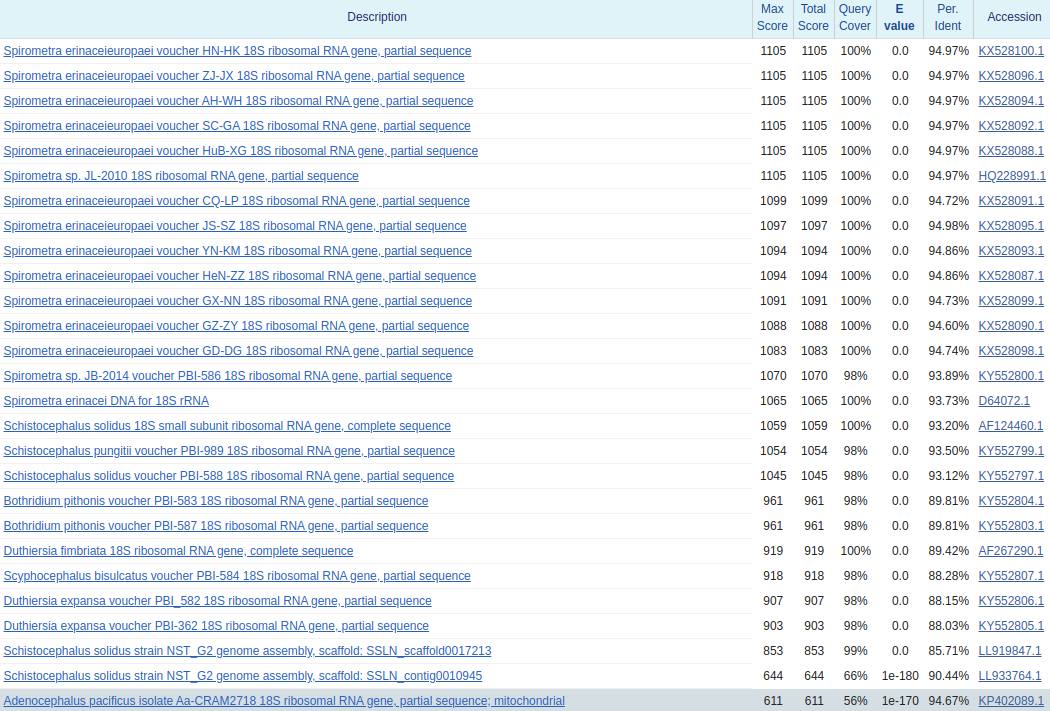
Некоторая часть списка находок изображена на рисунке 2. Результаты поиска с помощью blastn говорят о том, что данная нуклеотидная последовательность, скорее всего, кодирует рибосомальную РНК. Если быть точным, то исходя из того, что все первые 100 результатов с довольно большими E–value кодируют 18S rRNA, которая является структурным компонентом малой субъединицы рибосомы эукариот, то и исследуемая последовательность также, скорее всего, кодирует 18S rRNA. Она довольно консервативна, ее активно используют в филогенетике. Что касается таксономии организма, из которого был получен образец ДНК, то можно сказать, что его происхождение следующее:
Metazoa Eumetazoa Bilateria Platyhelminthes Cestoda Eucestoda Diphyllobothriidea DiphyllobothriidaeЭто предположение выдвинуто из–за того, что у четырех первых организмов с лучшими выравниваниями эти таксоны оказались максимально совпавшими. Также это вытекает из филогенетического древа, изображенного на рисунке 3.

Выравнивания
1. Выравнивание исследуемой последовательности (Query) и KY552791.1 (Sbjct) из Pyramicocephalus phocarum
Query 1 TGACGGAAGGGCACCACCAGGAGTGGAGCCTGCGGCTTAATTTGACTCAACACGGGAAAA 60
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1240 TGACGGAAGGGCACCACCAGGAGTGGAGCCTGCGGCTTAATTTGACTCAACACGGGAAAA 1299
Query 61 CTCACCCGGCCCGGACACTGTGAGGATTGACAGATTGATAGCTCTTTCTTGATTTGGTGG 120
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1300 CTCACCCGGCCCGGACACTGTGAGGATTGACAGATTGATAGCTCTTTCTTGATTTGGTGG 1359
Query 121 TTGGTGGTGCATGGCCGTTCTTAGTTGGTGGAGCGATTTGTCTGGTTAATTCCGATAACG 180
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1360 TTGGTGGTGCATGGCCGTTCTTAGTTGGTGGAGCGATTTGTCTGGTTAATTCCGATAACG 1419
Query 181 AACGAGACTCTAGCCTGCTAATTAGTACGCCTGTCCTCTGTTCCTGTGCTGGCGGTGCTT 240
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1420 AACGAGACTCTAGCCTGCTAATTAGTACGCCTGTCCTCTGTTCCTGTGCTGGCGGTGCTT 1479
Query 241 TGGTGAAACTGCTCACATATGCCGACTGCTATCGGGACGCGTTCTTGCGTGCGTTGCGAG 300
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1480 TGGTGAAACTGCTCACATATGCCGACTGCTATCGGGACGCGTTCTTGCGTGCGTTGCGAG 1539
Query 301 CTCCTCGTGGGCGAGTGGCGTGTGTAGGGGCGTGAACTGTGGTGGAAGGTGTGTGCGGTG 360
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1540 CTCCTCGTGGGCGAGTGGCGTGTGTAGGGGCGTGAACTGTGGTGGAAGGTGTGTGCGGTG 1599
Query 361 GGTCCGGTGCTTGCCTCCGGGTACGGCGCAGGTGTGTCTACTTCTTAGAGGGACAAGCAT 420
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1600 GGTCCGGTGCTTGCCTCCGGGTACGGCGCAGGTGTGTCTACTTCTTAGAGGGACAAGCAT 1659
Query 421 TACGAATGTGTACGAAATTGAGCAATAACAGGTCTGTGATGCCCTTAGATGTCCGGGGCC 480
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1660 TACGAATGTGTACGAAATTGAGCAATAACAGGTCTGTGATGCCCTTAGATGTCCGGGGCC 1719
Query 481 GCACGCGCGCTACAATGACGGTGCCAACGAGTCTGACCTTCTGGCCCGAAAGGGTTGGGG 540
|||||||||||||||||||||||||||||||||||||||||||||||||||||||| |||
Sbjct 1720 GCACGCGCGCTACAATGACGGTGCCAACGAGTCTGACCTTCTGGCCCGAAAGGGTT-GGG 1778
Query 541 CAAACTGGTCAATCACCGTCATGACAGGGATCGGGGCTTGGAATTGTTCCCCGTGAACGA 600
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1779 CAAACTGGTCAATCACCGTCATGACAGGGATCGGGGCTTGGAATTGTTCCCCGTGAACGA 1838
Query 601 GGAATTCCTAGTAAGTGCAAGTCATAAGCTTGCGCTGATTACGTCCCTGCCCTTTGTACA 660
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1839 GGAATTCCTAGTAAGTGCAAGTCATAAGCTTGCGCTGATTACGTCCCTGCCCTTTGTACA 1898
Query 661 CACCGCCCGTCGCTACTACCGATTGAATGGTTTASTAAGGTCCTCGGATTGGCGCCATTG 720
|||||||||||||||||||||||||||||||||| |||||||||||||||||||||||||
Sbjct 1899 CACCGCCCGTCGCTACTACCGATTGAATGGTTTAGTAAGGTCCTCGGATTGGCGCCATTG 1958
Query 721 CAGTATCACCCGCAAGGTTGGTGCTCGACAGGTGCCGAGAAGACGACCAAACYTGATCAY 780
|||||||||||||||||||||||||||||||||||||||||||||||||||| ||||||
Sbjct 1959 CAGTATCACCCGCAAGGTTGGTGCTCGACAGGTGCCGAGAAGACGACCAAACTTGATCAT 2018
Query 781 TTAGA 785
|||||
Sbjct 2019 TTAGA 2023
2. Выравнивание исследуемой последовательности (Query) и AB512013.1 (Sbjct) из Diphyllobothrium nihonkaiense
Query 1 TGACGGAAGGGCACCACCAGGAGTGGAGCCTGCGGCTTAATTTGACTCAACACGGGAAAA 60
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1329 TGACGGAAGGGCACCACCAGGAGTGGAGCCTGCGGCTTAATTTGACTCAACACGGGAAAA 1388
Query 61 CTCACCCGGCCCGGACACTGTGAGGATTGACAGATTGATAGCTCTTTCTTGATTTGGTGG 120
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1389 CTCACCCGGCCCGGACACTGTGAGGATTGACAGATTGATAGCTCTTTCTTGATTTGGTGG 1448
Query 121 TTGGTGGTGCATGGCCGTTCTTAGTTGGTGGAGCGATTTGTCTGGTTAATTCCGATAACG 180
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1449 TTGGTGGTGCATGGCCGTTCTTAGTTGGTGGAGCGATTTGTCTGGTTAATTCCGATAACG 1508
Query 181 AACGAGACTCTAGCCTGCTAATTAGTACGCCTGTCCTCTGTTCCTGTGCTGGCGGTGCTT 240
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1509 AACGAGACTCTAGCCTGCTAATTAGTACGCCTGTCCTCTGTTCCTGTGCTGGCGGTGCTT 1568
Query 241 TGGTGAAACTGCTCACATATGCCGACTGCTATCGGGACGCGTTCTTGCGTGCGTTGCGAG 300
||| ||||||||||||||||| |||||||||| |||||||||||||||||| | |||||
Sbjct 1569 TGGCGAAACTGCTCACATATGTCGACTGCTATGGGGACGCGTTCTTGCGTGTGCCGCGAG 1628
Query 301 CTCCTCGTGGGCGAGTGGCGTGTGTAGGGGCGTGAACTGTGGTGGAAGGTGTGTGCGGTG 360
|||||||||||| ||||||||| |||||||||||||||||||| | ||| ||||||||||
Sbjct 1629 CTCCTCGTGGGCAAGTGGCGTGCGTAGGGGCGTGAACTGTGGTAGGAGGCGTGTGCGGTG 1688
Query 361 GGTCCGGTGCTTGCCTCCGGGTACGGCGCAGGTGTGTCTACTTCTTAGAGGGACAAGCAT 420
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1689 GGTCCGGTGCTTGCCTCCGGGTACGGCGCAGGTGTGTCTACTTCTTAGAGGGACAAGCAT 1748
Query 421 TACGAATGTGTACGAAATTGAGCAATAACAGGTCTGTGATGCCCTTAGATGTCCGGGGCC 480
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1749 TACGAATGTGTACGAAATTGAGCAATAACAGGTCTGTGATGCCCTTAGATGTCCGGGGCC 1808
Query 481 GCACGCGCGCTACAATGACGGTGCCAACGAGTCTGACCTTCTGGCCCGAAAGGGTTGGGG 540
|||||||||||||||||||||||||||||||||||||||||||||||||||||||| |||
Sbjct 1809 GCACGCGCGCTACAATGACGGTGCCAACGAGTCTGACCTTCTGGCCCGAAAGGGTT-GGG 1867
Query 541 CAAACTGGTCAATCACCGTCATGACAGGGATCGGGGCTTGGAATTGTTCCCCGTGAACGA 600
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1868 CAAACTGGTCAATCACCGTCATGACAGGGATCGGGGCTTGGAATTGTTCCCCGTGAACGA 1927
Query 601 GGAATTCCTAGTAAGTGCAAGTCATAAGCTTGCGCTGATTACGTCCCTGCCCTTTGTACA 660
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1928 GGAATTCCTAGTAAGTGCAAGTCATAAGCTTGCGCTGATTACGTCCCTGCCCTTTGTACA 1987
Query 661 CACCGCCCGTCGCTACTACCGATTGAATGGTTTASTAAGGTCCTCGGATTGGCGCCATTG 720
|||||||||||||||||||||||||||||||||| |||||||||||||||||||||||||
Sbjct 1988 CACCGCCCGTCGCTACTACCGATTGAATGGTTTAGTAAGGTCCTCGGATTGGCGCCATTG 2047
Query 721 CAGTATCACCCGCAAGGTTGGTGCTCGACAGGTGCCGAGAAGACGACCAAACYTGATCAY 780
|||||||| ||||||||||||||||||||||||||||||||||||||||||| ||||||
Sbjct 2048 CAGTATCAGCCGCAAGGTTGGTGCTCGACAGGTGCCGAGAAGACGACCAAACTTGATCAT 2107
Query 781 TTAGAGGAAGTAAA 794
||||||||||||||
Sbjct 2108 TTAGAGGAAGTAAA 2121
3. Выравнивание исследуемой последовательности (Query) и DQ181944.2 (Sbjct) из Diphyllobothrium ditremum
Query 1 TGACGGAAGGGCACCACCAGGAGTGGAGCCTGCGGCTTAATTTGACTCAACACGGGAAAA 60
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 127 TGACGGAAGGGCACCACCAGGAGTGGAGCCTGCGGCTTAATTTGACTCAACACGGGAAAA 186
Query 61 CTCACCCGGCCCGGACACTGTGAGGATTGACAGATTGATAGCTCTTTCTTGATTTGGTGG 120
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 187 CTCACCCGGCCCGGACACTGTGAGGATTGACAGATTGATAGCTCTTTCTTGATTTGGTGG 246
Query 121 TTGGTGGTGCATGGCCGTTCTTAGTTGGTGGAGCGATTTGTCTGGTTAATTCCGATAACG 180
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 247 TTGGTGGTGCATGGCCGTTCTTAGTTGGTGGAGCGATTTGTCTGGTTAATTCCGATAACG 306
Query 181 AACGAGACTCTAGCCTGCTAATTAGTACGCCTGTCCTCTGTTCCTGTGCTGGCGGTGCTT 240
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 307 AACGAGACTCTAGCCTGCTAATTAGTACGCCTGTCCTCTGTTCCTGTGCTGGCGGTGCTT 366
Query 241 TGGTGAAACTGCTCACATATGCCGACTGCTATCGGGACGCGTTCTTGCGTGCGTTGCGAG 300
||| ||||||||||||||||| |||||||||||||||||||||||||||| | |||||
Sbjct 367 TGGCGAAACTGCTCACATATGTCGACTGCTATCGGGACGCGTTCTTGCGTATGCCGCGAG 426
Query 301 CTCCTCGTGGGCGAGTGGCGTGTGTAGGGGCGTGAACTGTGGTGGAAGGTGTGTGCGGTG 360
|||||||||||||||||| ||| |||||||||||||||||||| | ||| ||||||||||
Sbjct 427 CTCCTCGTGGGCGAGTGGTGTGCGTAGGGGCGTGAACTGTGGTAGGAGGCGTGTGCGGTG 486
Query 361 GGTCCGGTGCTTGCCTCCGGGTACGGCGCAGGTGTGTCTACTTCTTAGAGGGACAAGCAT 420
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 487 GGTCCGGTGCTTGCCTCCGGGTACGGCGCAGGTGTGTCTACTTCTTAGAGGGACAAGCAT 546
Query 421 TACGAATGTGTACGAAATTGAGCAATAACAGGTCTGTGATGCCCTTAGATGTCCGGGGCC 480
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 547 TACGAATGTGTACGAAATTGAGCAATAACAGGTCTGTGATGCCCTTAGATGTCCGGGGCC 606
Query 481 GCACGCGCGCTACAATGACGGTGCCAACGAGTCTGACCTTCTGGCCCGAAAGGGTTGGGG 540
|||||||||||||||||||||||||||||||||||||||||||||||||||||||| |||
Sbjct 607 GCACGCGCGCTACAATGACGGTGCCAACGAGTCTGACCTTCTGGCCCGAAAGGGTT-GGG 665
Query 541 CAAACTGGTCAATCACCGTCATGACAGGGATCGGGGCTTGGAATTGTTCCCCGTGAACGA 600
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 666 CAAACTGGTCAATCACCGTCATGACAGGGATCGGGGCTTGGAATTGTTCCCCGTGAACGA 725
Query 601 GGAATTCCTAGTAAGTGCAAGTCATAAGCTTGCGCTGATTACGTCCCTGCCCTTTGTACA 660
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 726 GGAATTCCTAGTAAGTGCAAGTCATAAGCTTGCGCTGATTACGTCCCTGCCCTTTGTACA 785
Query 661 CACCGCCCGTCGCTACTACCGATTGAATGGTTTASTAAGGTCCTCGGATTGGCGCCATTG 720
|||||||||||||||||||||||||||||||||| |||||||||||||||||||||||||
Sbjct 786 CACCGCCCGTCGCTACTACCGATTGAATGGTTTAGTAAGGTCCTCGGATTGGCGCCATTG 845
Query 721 CAGTATCACCCGCAAGGTTGGTGCTCGACAGGTGCCGAGAAGACGACCAAACYTGATCAY 780
|||||||| ||||||||||||||||||||||||||||||||||||||||||| ||||||
Sbjct 846 CAGTATCAGCCGCAAGGTTGGTGCTCGACAGGTGCCGAGAAGACGACCAAACTTGATCAT 905
Query 781 TTAGAGGAAGTAAA 794
||||||||||||||
Sbjct 906 TTAGAGGAAGTAAA 919
4. Выравнивание исследуемой последовательности (Query) и KF218251.1 (Sbjct) из Diphyllobothrium latum
Query 1 TGACGGAAGGGCACCACCAGGAGTGGAGCCTGCGGCTTAATTTGACTCAACACGGGAAAA 60
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1347 TGACGGAAGGGCACCACCAGGAGTGGAGCCTGCGGCTTAATTTGACTCAACACGGGAAAA 1406
Query 61 CTCACCCGGCCCGGACACTGTGAGGATTGACAGATTGATAGCTCTTTCTTGATTTGGTGG 120
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1407 CTCACCCGGCCCGGACACTGTGAGGATTGACAGATTGATAGCTCTTTCTTGATTTGGTGG 1466
Query 121 TTGGTGGTGCATGGCCGTTCTTAGTTGGTGGAGCGATTTGTCTGGTTAATTCCGATAACG 180
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1467 TTGGTGGTGCATGGCCGTTCTTAGTTGGTGGAGCGATTTGTCTGGTTAATTCCGATAACG 1526
Query 181 AACGAGACTCTAGCCTGCTAATTAGTACGCCTGTCCTCTGTTCCTGTGCTGGCGGTGCTT 240
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1527 AACGAGACTCTAGCCTGCTAATTAGTACGCCTGTCCTCTGTTCCTGTGCTGGCGGTGCTT 1586
Query 241 TGGTGAAACTGCTCACATATGCCGACTGCTATCGGGACGCGTTCTTGCGTGCGTTGCGAG 300
||| ||||||||||||||||| |||||||||| ||||||||||||||||| | |||||
Sbjct 1587 TGGCGAAACTGCTCACATATGTCGACTGCTATTGGGACGCGTTCTTGCGTATGCCGCGAG 1646
Query 301 CTCCTCGTGGGCGAGTGGCGTGTGTAGGGGCGTGAACTGTGGTGGAAGGTGTGTGCGGTG 360
|||||||||||||||||| ||| |||||||||||||||||||| | ||| ||||||||||
Sbjct 1647 CTCCTCGTGGGCGAGTGGTGTGCGTAGGGGCGTGAACTGTGGTAGGAGGCGTGTGCGGTG 1706
Query 361 GGTCCGGTGCTTGCCTCCGGGTACGGCGCAGGTGTGTCTACTTCTTAGAGGGACAAGCAT 420
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1707 GGTCCGGTGCTTGCCTCCGGGTACGGCGCAGGTGTGTCTACTTCTTAGAGGGACAAGCAT 1766
Query 421 TACGAATGTGTACGAAATTGAGCAATAACAGGTCTGTGATGCCCTTAGATGTCCGGGGCC 480
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1767 TACGAATGTGTACGAAATTGAGCAATAACAGGTCTGTGATGCCCTTAGATGTCCGGGGCC 1826
Query 481 GCACGCGCGCTACAATGACGGTGCCAACGAGTCTGACCTTCTGGCCCGAAAGGGTTGGGG 540
|||||||||||||||||||||||||||||||||||||||||||||||||||||||| |||
Sbjct 1827 GCACGCGCGCTACAATGACGGTGCCAACGAGTCTGACCTTCTGGCCCGAAAGGGTT-GGG 1885
Query 541 CAAACTGGTCAATCACCGTCATGACAGGGATCGGGGCTTGGAATTGTTCCCCGTGAACGA 600
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1886 CAAACTGGTCAATCACCGTCATGACAGGGATCGGGGCTTGGAATTGTTCCCCGTGAACGA 1945
Query 601 GGAATTCCTAGTAAGTGCAAGTCATAAGCTTGCGCTGATTACGTCCCTGCCCTTTGTACA 660
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1946 GGAATTCCTAGTAAGTGCAAGTCATAAGCTTGCGCTGATTACGTCCCTGCCCTTTGTACA 2005
Query 661 CACCGCCCGTCGCTACTACCGATTGAATGGTTTASTAAGGTCCTCGGATTGGCGCCATTG 720
|||||||||||||||||||||||||||||||||| |||||||||||||||||||||||||
Sbjct 2006 CACCGCCCGTCGCTACTACCGATTGAATGGTTTAGTAAGGTCCTCGGATTGGCGCCATTG 2065
Query 721 CAGTATCACCCGCAAGGTTGGTGCTCGACAGGTGCCGAGAAGACGACCAAACYTGATCAY 780
|||||||| ||||||||||||||||||||||||||||||||||||||||||| ||||||
Sbjct 2066 CAGTATCAGCCGCAAGGTTGGTGCTCGACAGGTGCCGAGAAGACGACCAAACTTGATCAT 2125
Query 781 TTAGAGGAAGTAAA 794
||||||||||||||
Sbjct 2126 TTAGAGGAAGTAAA 2139
- | обозначает полное соответствие нуклеотидов
- обозначает схожесть нуклеотидов (например, G входит в класс S)
- обозначает полное несоответствие нуклеотидов
По–видимому, в интерпретации хроматограммы из практикума 6 была допущена ошибка: был добавлен лишний гуанин на позицию 547 (по последовательности Query). Возможно, конечно, это не ошибка, так как инсерция одного нуклеотида для рРНК не так страшна, как для белка.
Изучаемая последовательность с достаточно большой вероятностью может относиться к виду Pyramicocephalus phocarum, так как практически никаких разногласий между двумя последовательностями (изучаемой и из этого вида) найдено не было. А вот от последовательностей из организма рода Diphyllobothrium искомая последовательность отличается довольно сильно: примерно в области с 241 по 360 позицию находится по 12 несовпадений.
Сравнение результатов работы алгоритмов BLAST
Последовательность из практикума 6
Для данной последовательности были запущены 3 программы: megablast (для очень схожих последовательностей), blastn с обычными параметрами (для довольно разных, но все–же схожих последовательностей) и blastn с чувствительными параметрами (для хоть сколько–нибудь схожих последовательностей). Некоторые параметры запусков описаны в таблице 1. megablast оказался самой быстрой программой, а blastn с обычными параметрами оказался быстрее, чем blastn с модифицированными параметрами.
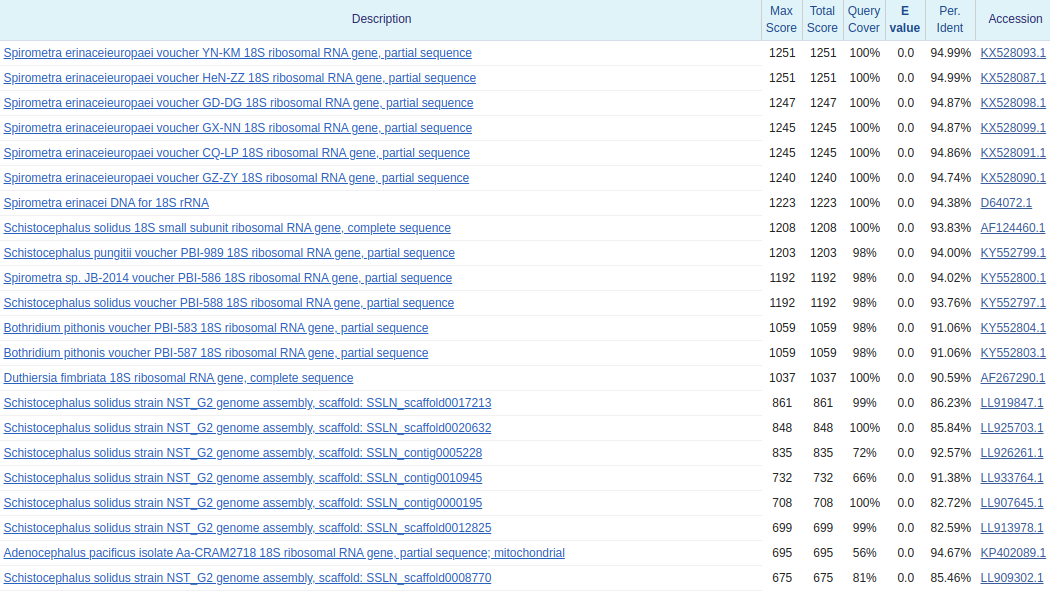
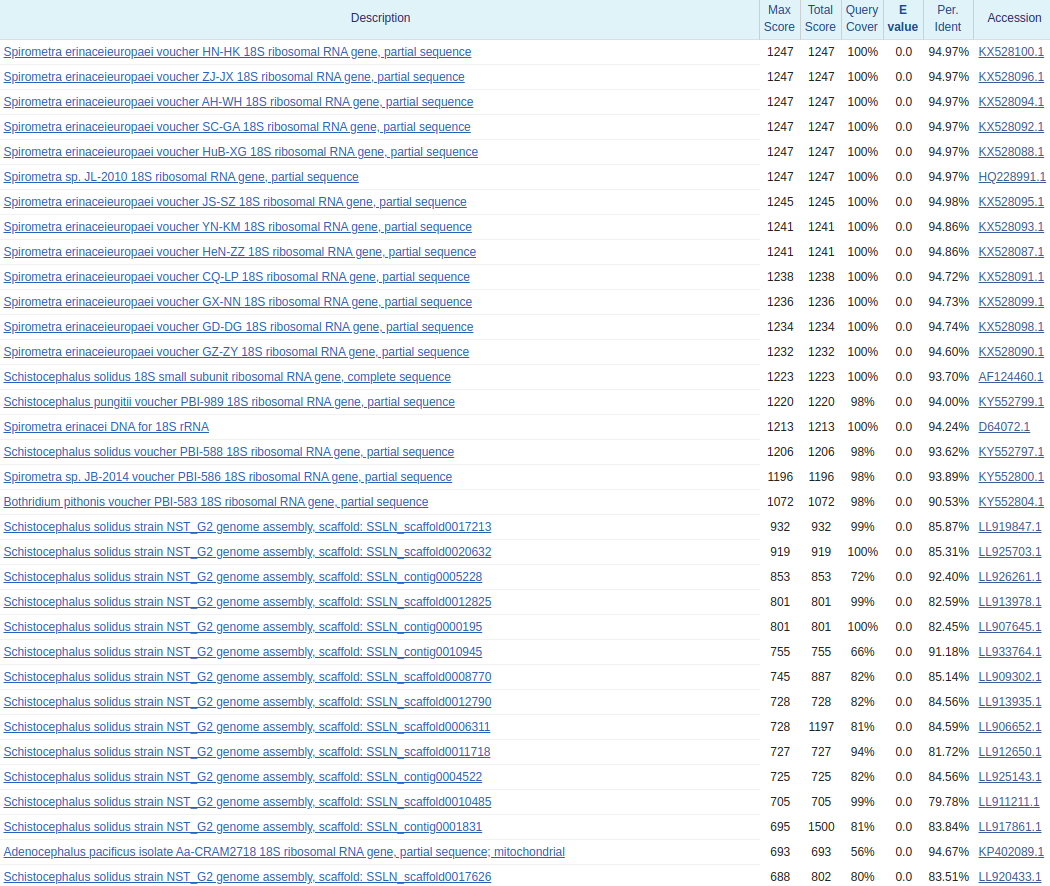
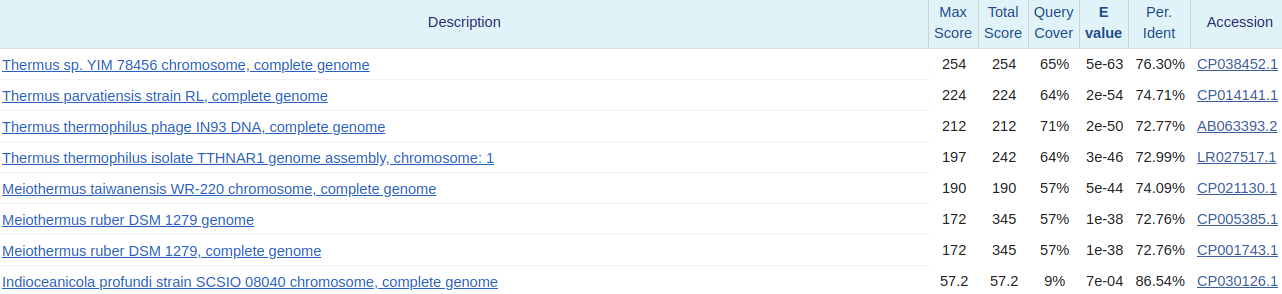
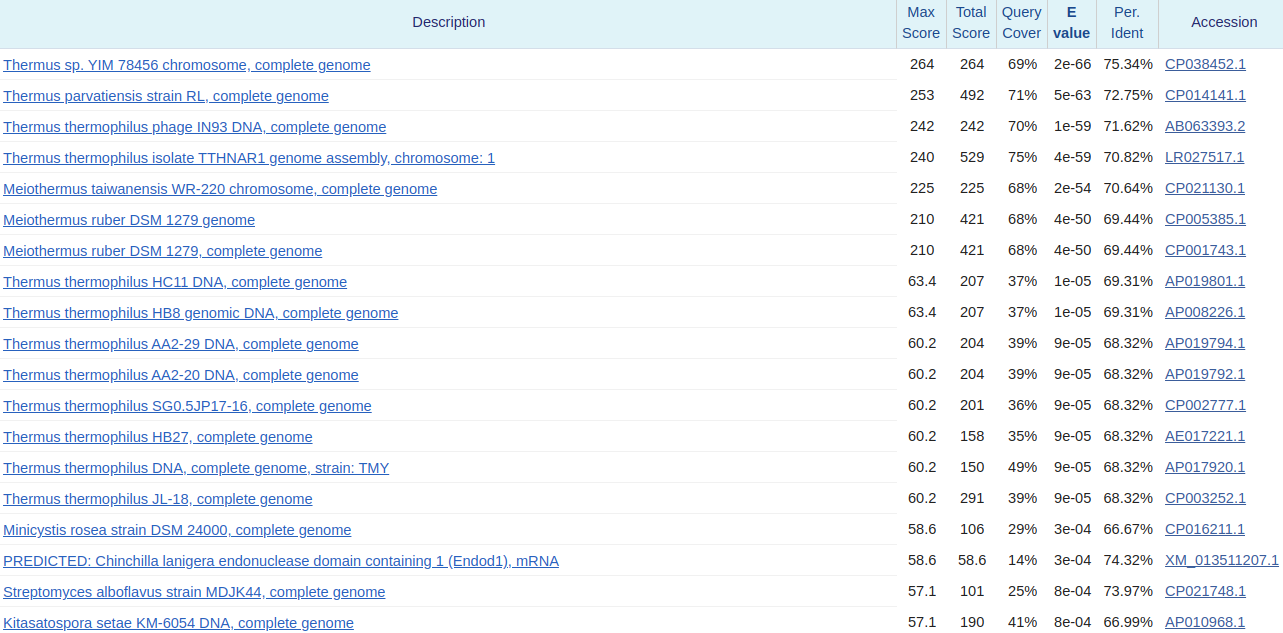
Результат работы программ немного удивил. Почему–то, при запуске blastn с чувствительными параметрами находок получилось меньше, чем в обычном blastn и даже megablast. Когда же провели фильтрацию результатов (учитывали только те последовательности, у которых процент идентичности находился между 60% и 95%, а также E–value было меньше, чем 0,001), то получили следующие количества находок: megablast — 22, blastn обычный — 34, blastn чувтсвительный — 27. На рисунках 4, 5, 6 изображены результаты работы программ megablast'а, обычного blastn'а и чувтсвительного blastn'а.
Хочется заметить, что megablast упустил целый ряд важных находок. Например, он не учел запись Spirometra erinaceieuropaei voucher ZJ-JX 18S ribosomal RNA gene, partial sequence и записи других штаммов этого вида. А результатов чувствительного blastn оказалось меньше, чем у blastn с обычными настройками из-за того, что blastn с обычными настройками наткнулся на сборку генома Schistocephalus solidus strain NST_G2, а с измененными параметрами — нет.
| Алгоритм | Word Size | Match/Mismatch scores | Число находок |
|---|---|---|---|
| MegaBlast | 28 | 1, -2 | 922 |
| Blastn стандартный | 11 | 2, -3 | 962 |
| Blastn чувствительный | 7 | 1, -1 | 866 |
Последовательность CDS Thermus phage P23–77
В данной части задания нужно было провести такое же сравнение разных алгоритмов BLAST, как и в предыдущей части задания. Только вот для сравнения был взят кодирущий регион 4378–4899 NC_013197.1 из генома Thermus phage P23–77. Результат можно найти в таблице 2.
Все результаты работы можно найти на рисунках После того, как мы отфильтровали результаты (Identity 60%–95%, E–value < 0,001), оказалось, что megablast не нашел ничего, blastn с обычными параметрами выдал 8 находок, а чувствительный blastn — 19.
| Алгоритм | Word Size | Match/Mismatch scores | Число находок |
|---|---|---|---|
| MegaBlast | 28 | 1, -2 | 1 |
| Blastn стандартный | 11 | 2, -3 | 576 |
| Blastn чувствительный | 7 | 1, -1 | 540 |
Интересно, что чувствительный blastn нашел запись Kitasatospora setae KM-6054 DNA, complete genome, то есть запись того организма, с которым я работал в практикумах первого семестра.
Выводы
Для быстрого поиска практически идентичных последовательностей подходит megablast, blastn с обычными параметрами хорошо подходит для поиска схожих последовательностей, а вот blastn с чувствительными параметрами подходит для поиска сколько–нибудь схожих последовательностей, но не всегда.
Гомологи белков в неаннотированном геноме
В данном задании рассматривается неаннотированный геном Amoeboaphelidium protococcarum. Так как было необходимо найти гомологи белков в геноме, то я пользовался tblastn. Последовательности белков я получил с сайта UniProt (View Format FASTA (canonical)).
ACT_YEAST — актин из Saccharomyces cerevisiae. Это глобулярный белок, который при сборке друг с другом дает микрофиламенты — основу цитоскелета.
SUCB_YEAST — β субъединица сукцинил-КоА-синтетазы (SCS) из S. cerevisiae. SCS — фермент, катализирующий обратимую реакцию превращения сукцинил КоА в сукцинат (одновременно образуя ГТФ или АТФ из ГДФ или АДФ и фосфата). Этот фермент принимает участие в цикле Кребса.
DPOD_YEAST — каталитическая субъединица ДНК полимеразы δ (Polδ) из S. cerevisiae. Polδ — фермент, осуществляющий синтез отстающей цепочки ДНК и ее пруфридинг.
Результаты
ACT_YEAST: лучший результат среди всех трех белков по качеству и количеству совпадений. Итог: положительный – есть гомолог, вероятно, выполняющий ту же функцию. Самое маленькое E-value — 0 (скэффолды 444, 514 и 17). Всего 18 находок. Покрытие для лучшего результата стопроцентное, гэпов нет.
SUCB_YEAST: худший результат среди трех рассматриваемых белков. Итог положительный. Количество совпадений — 4, лучшее E-value — 3 * 1084 (скэффолд 361). Покрытие для лучшей находки — 92%, гэпов мало.
DPOD_YEAST: средний результат. Итог положительный. Нашлось 8 похожих последовательностей. Две последовательности (скэффолды 81 и 529) дали выравнивания с нулевым E-value. Процент покрытия для первого результата — 97%, гэпов очень мало.
Поиск гена белка в контиге
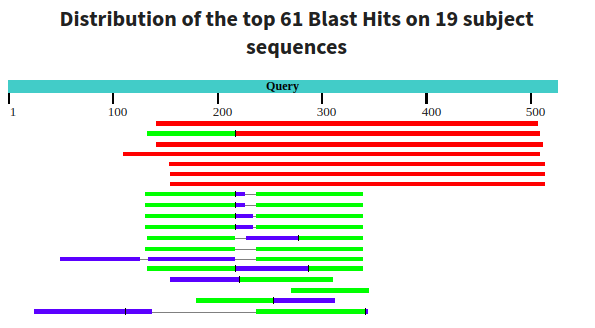
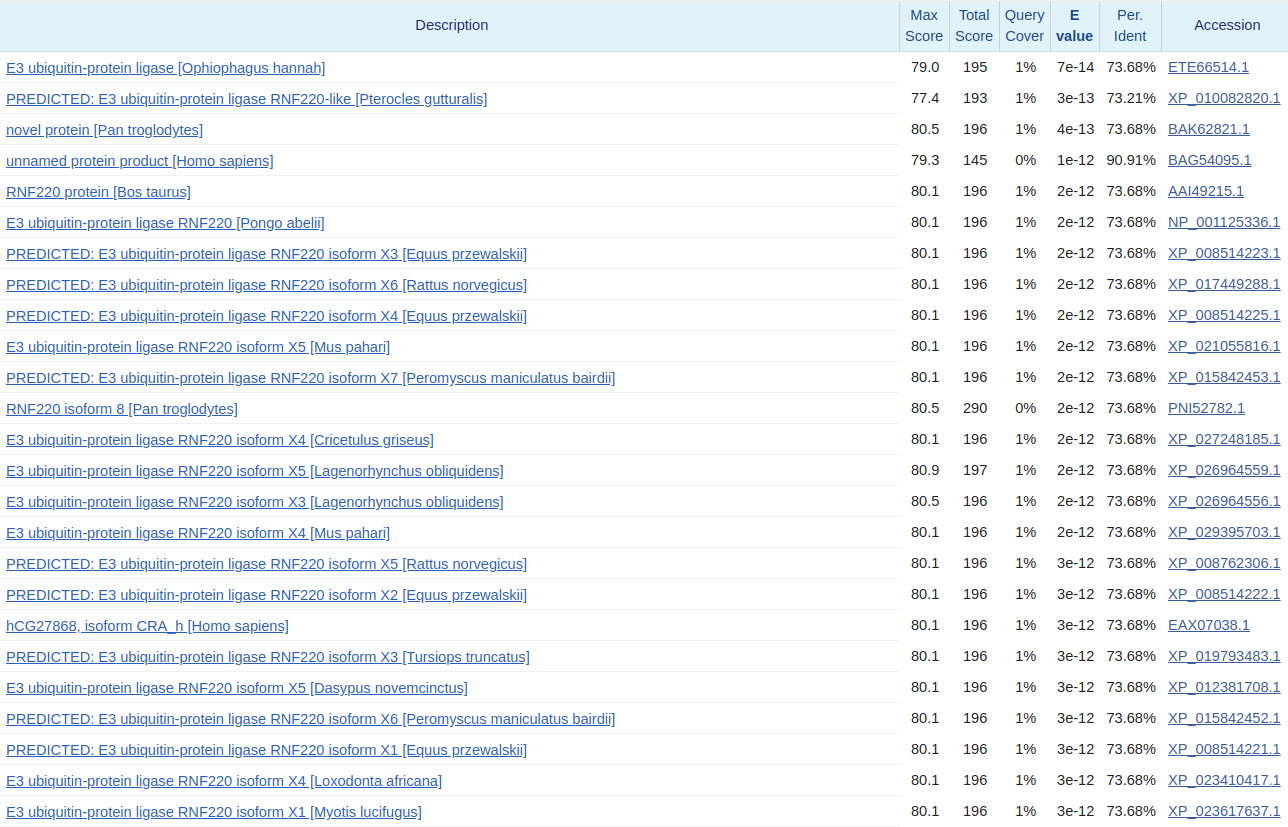
Для выполнения данного задания был выбран контиг JDSB01000005.1 из неаннотированного генома Aquila chrysaetos canadensis.
Результат изображен на рисунке 11. Можно сказать, что в контиге находится ген, кодирующий убиквитинлигазу (E3 ubiquitin ligase), так как результаты хорошие (лучшее E-value — 7 * 10-14.
Дополнительные материалы