Я выбрала один из контигов организма X5 (Amoboaphelidium) - сборка генома X5 - с помощью программы infoseq (пакета EMBOSS). Контиг называется unplaced-1014 и имеет длину от 20 кб до 100 kb, а именно 32260 bp.
Я предсказывала гены с помощью AUGUSTUS. В AUGUSTUS необходимо произвести обучение сервиса, для этого нужно было найти самого близкого родственника из списка организмов, для которых уже произведено обучение. Чтобы найти самого близкого родственника я запустила программу BLAST для своего контига. Так как я хочу предстаказать гены, которые кодируют белки в организме X5, я использовала алгоритм blastx при поиске родственников, потому что консервативность аминокислотной последовательности белка выше, чем консервативность нуклеотидной последовательности гена.
Для программы blastx я взяла фрагмент последовательности из контига unplaced-1014. Результаты работы программы blastx представлены на рис.1. На рис.2 можно увидеть таксономию находок. Почти все находки blastx принадлежат к царству Fungi. Наиболее часто из имеющихся в списке организмов, для которых произведено обучение, встречается род Rhizopus. Поэтому в качестве близкого родственника я использовала Rhizopus oryzae.
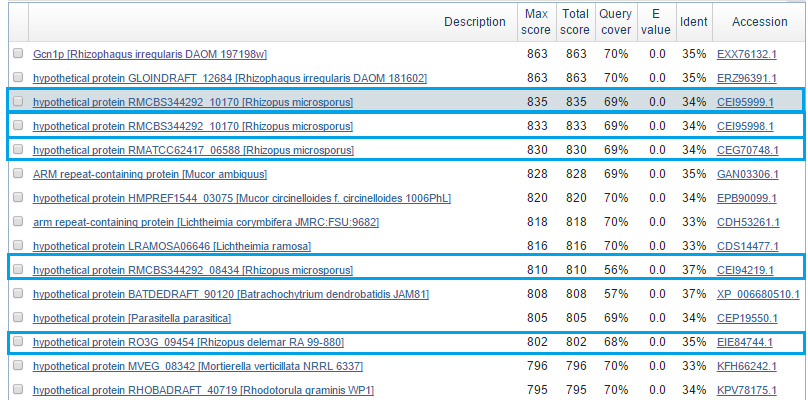
Рис.1 Фрагмент находок программы blastx.
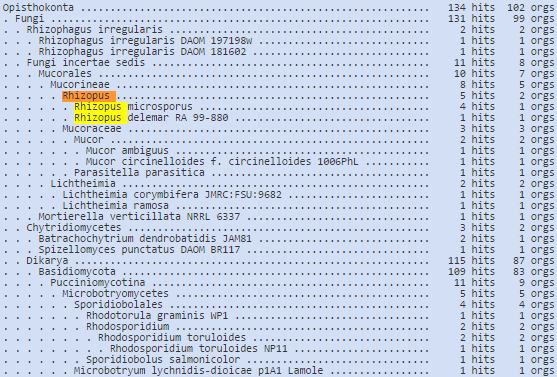
Рис.2 Фрагмент таксономии находок.
На сайте AUGUSTUS я выбрала ссылку "AUGUSTUS prediction submission". Там я прикрепила файл с последовательностью контига unplaced-1014 и выбрала specify a project identifier - rhizopus_oryzae. Остальные параметры оставила по умолчанию. Через несколько минут я получила результат.
В архиве содержатся файлы:
- augustus.aa - предсказание аминокислотной последовательности генов в fasta-формате
- augustus.cdsexons - предсказанные экзоны, последовательности нуклеиновых кислот в fasta-формате
- augustus.codingseq - предсказание последовательности гена в виде нуклеиновых кислот в fasta-формате
- augustus.gbrowse - файл с таблицей предсказанных генов без описания и без последовательностей
- augustus.gff - файл, который содержит полное описание предсказанных генов с их нуклеотидными и аминокислотными последовательностями
- augustus.gtf - файл с таблицей предсказанных генов без описания и без последовательностей
Cписок предсказанных генов, их экзон-интронная струтктура представлена в таблице.
Проверку предсказания я осуществила с помощью BLAST. Из файла augustus.aa я взяла аминокислотные последовательности и запустила blastp.
Фрагмент находок blastp для 8 гена представлены на рис.3. Находки довольно хорошие. Суда по ним, можно сделать вывод, что границы генов и их экзон-интронная структура 8 гена предсказана правильно. Возможная функция гена - glutamine-fructose-6-phosphate transaminase.
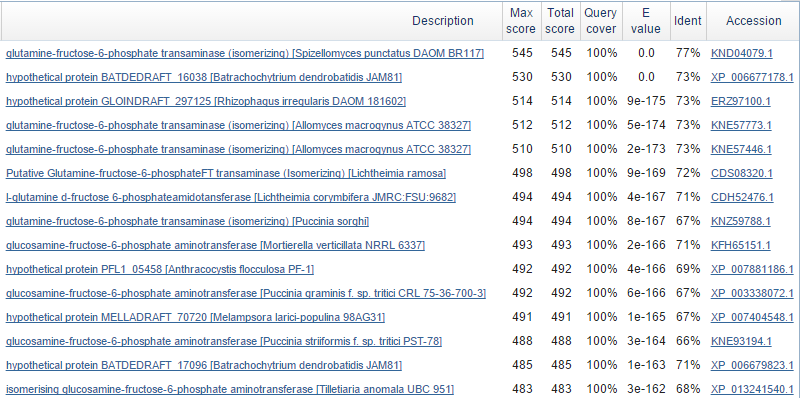
Рис.3 Фрагмент находок blastp 8 гена.
Фрагмент находок blastp для 13 гена представлены на рис.4. Процент идентичности находок низкий, много находок с гипотетическими белками, что говорит о том, что достоверность данной предсказанной находки довольно низкая. Сделать более подробные выводы об правильности экзон-интронной структуры и функции белка я не могу на основании полученных данных.
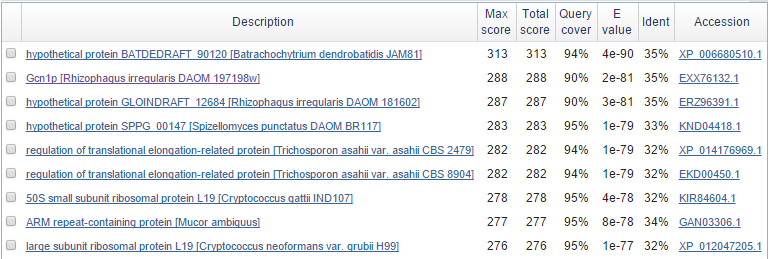
Рис.4 Фрагмент находок blastp 13 гена.
Фрагмент находок blastp для 8 гена представлены на рис.5. Находки довольно хорошие. Суда по ним, можно сделать вывод, что границы генов и их экзон-интронная структура 8 гена предсказана правильно. Возможная функция гена - glutamine-fructose-6-phosphate transaminase.


Рис.5 Фрагмент находок blastp 8 гена.
Сравние аннотации Refseq и AUGUSTUS одного гена человекаДля выполнения данного задания я использовала UCSC Genome Browser, в которой выбрала последнюю сборку генома hg38. Я оставила только три трэка: base position, Refseq и AUGUSTUS.
Я выбрала ген VDR - рецептор витамина D (1,25- dihydroxyvitamin D3) из организма Homo sapiens. На рис.6 представлен скриншот окна браузера с двумя аннотациями гена. Координаты гена: 12 хромосома, ориентация цепи -1, от 47841537 до 47905031 (NCBI)/ от 47841536 до 47905031 (Refseq)/ от 47844269 до 47963447 (AUGUSTUS).

Рис.6 Интрон-экзонная структура гена VDR.
В таблице agustus.xlsx представлена экзон-интронная структура гена VDR согласно предсказанию AGUSTUS. В таблице Refseq.xlsx представлена экзон-интронная структура гена VDR согласно предсказанию Refseq. В таблице a&r.xlsx представлено сравнение аннотиций экзон-интронной структуры гена VDR согласно предсказаниям Refseq и AGUSTUS. Аннотации Refseq и AGUSTUS идентично определили все экзоны кроме первого и последних экзонов. В первом гене Refseq взял начала экзона намного раньше, чем AGUSTUS. Refseq аннотировал два последних экзона, а AGUSTUS только один.
.jpg)