Далее мною были выбраны два вертикальных блока, расположенных рядом.(рис. 5). Вместе они содержат 40 позиций. Позиций с гэпами - 10. Процент позиций с гэпами - 25%. Стоит заметить, что гэпы "обусловлены" лишь одной из последовательностей. Поэтому в данном случае я считаю, что эти два блока можно объединить в единый кластер. Скорее всего, при таком оценивании стоит учитывать "плотность" гэпов, при этом качество выравнивания будет обратно пропорционально плотности, т.е чем больше доля гэпов в позициях, содержащих хотя бы один гэп, тем меньше их значение.(в данном случае плотность гэпов будет равна 16/17).
Скачать файл с кластером можно по этой ссылке
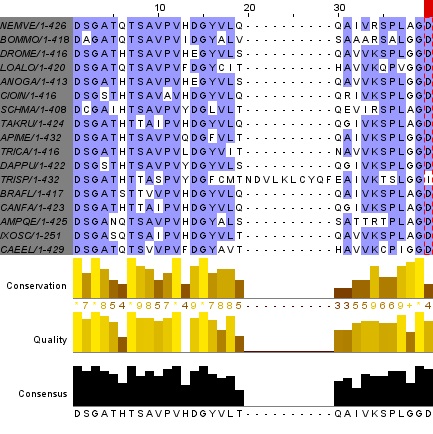
Рис. 5. Участок, содержащий два рядом расположенных вертикальных блока.
Затем я добавил еще одну последовательность к множественному выравниванию и попытался её выравнять(скачать последовательность можно по этой ссылке). Результат можно увидеть на рис. 6. Скачать файл с выравниванием можно по этой ссылке. Скачать блок, показанный на рисунке можно по этой ссылке.

Рис. 6. Выравнивание с добавленной последовательностью
Так как выравнивание вручную в данном случае крайне неэффективно(к примеру, в конце выравнивания есть также совпадающий блок, но вручную выровнять так, чтобы оба блока совпадали не удается), то была применена сервиса muscle. Полученное
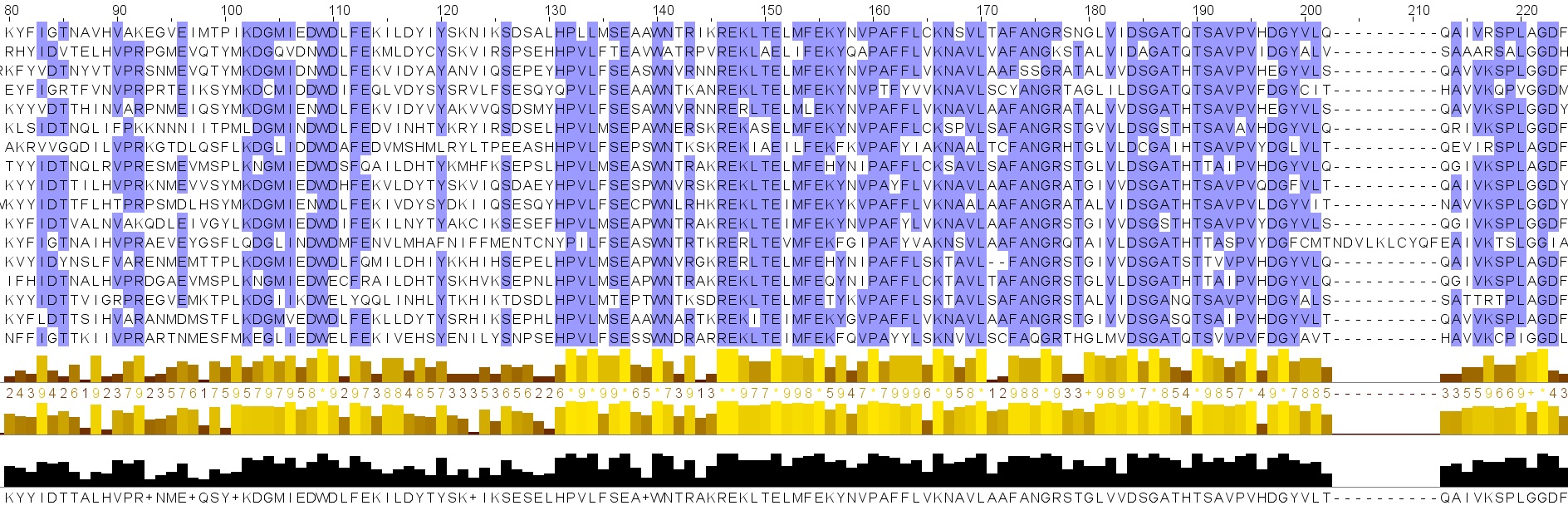
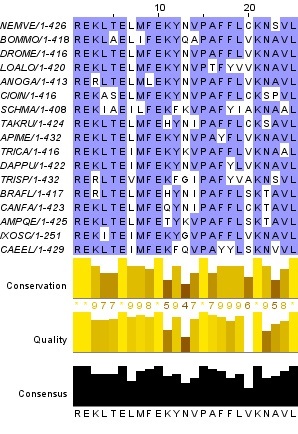
множественное выравнивание можно скачать в формате проекта JalView(.jar) по этой ссылке. Часть этого выравнивания можно увидеть на рис. 7. На рис. 8 представлен один блок из выравнивания.
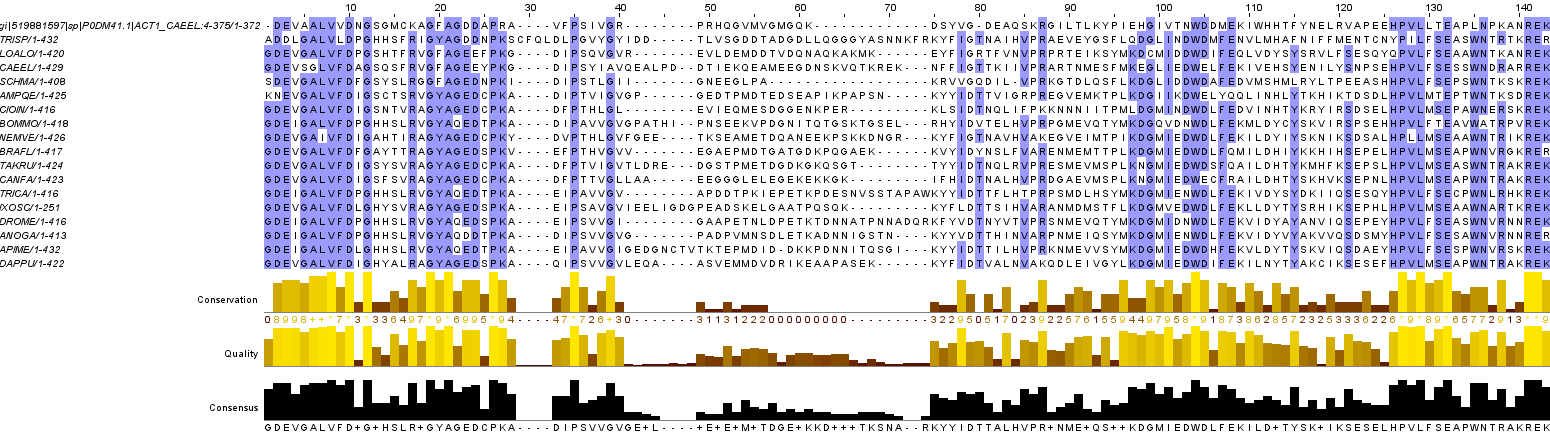
Рис. 7. Выравнивание, полученное с помощью сервиса muscle.
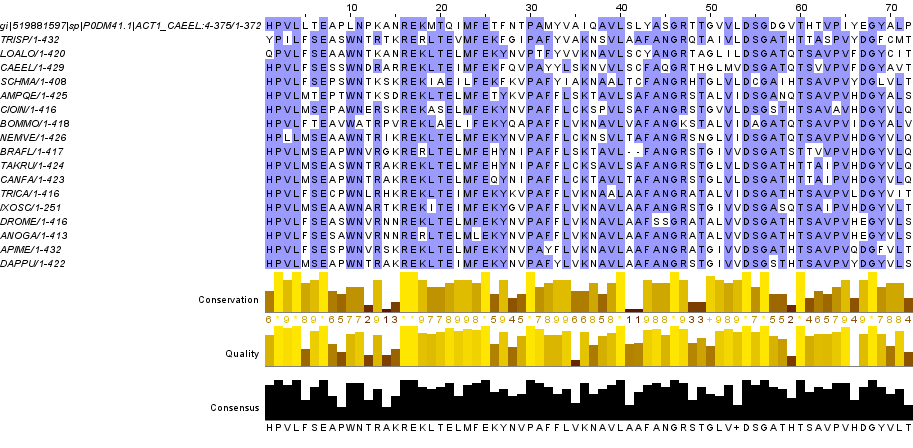
Рис. 8. Блок из выравнивания, полученный с помощью сервиса muscle.
Консенсусная последовательность, полученная с помощью EMBOSS:
GDEVGALVFDxGxHSLRVGYAGEDxPKA
Изображение, созданное на сайте
WebLogo можно увидеть на рис. 9
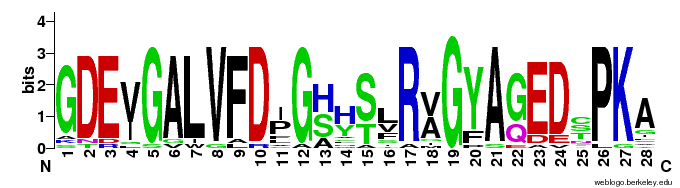
Рис. 9. Logo, полученное при помощи WebLogo.
Также мною было получено дерево сходства между последовательностями(использовалась опция "Calculate Tree->Average Distance Using Identity%"). Его можно увидеть на рис. 10. Исходя из него можно предположить, что добавленная последовательность давно отошла от общего дерева.
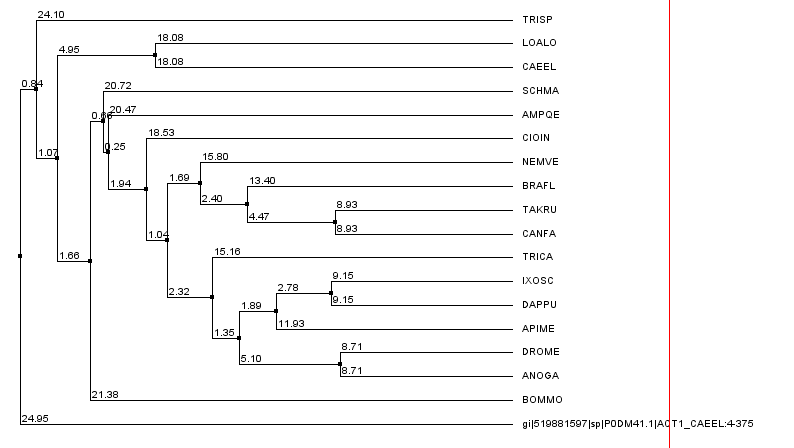
Рис. 10. Дерево сходства последовательностей
После этого я создал файл, содержащий семь белков из списка белков студентов моего курса. Скачать файл можно по ссылке. После этого
с помощью Web Service в Jalview 2.8. Файл выравнивания в формате .jar можно можно по ссылке. Из выравнивания ясно, что белки гомологами не являются(рис. 11, рис.12, рис. 13). Файл с "блоком 1" можно скачать по этой ссылке.
Файл с "блоком 2" можно скачать по этой ссылке .

Рис. 11. Выравнивание аминокислотных последовательностей белков из списка белков студентов моего курса.
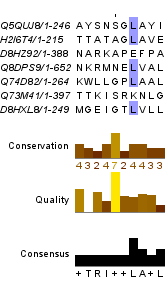
Рис. 12. "Блок 1" выравнивания

Рис. 13. "Блок 2" выравнивания
Дата последнего изменения: 15.04.2014
Все материалы разрешается использовать только при извещении правообладателя.
© Penzar Dmitry. All rights reserved.