Работа в BLAST
Поиск гомологов фминокислотной последовательности белка β-порфираназы А из бактерии Zobellia galactinovorans в банке Uniprot/SwissProt.
Мною был осуществлен поиск гомологов моего белка в банке Uniprot/SwissProt с помощью программы Protein BLAST. Первые результаты выдачи можно увидеть на рис.1
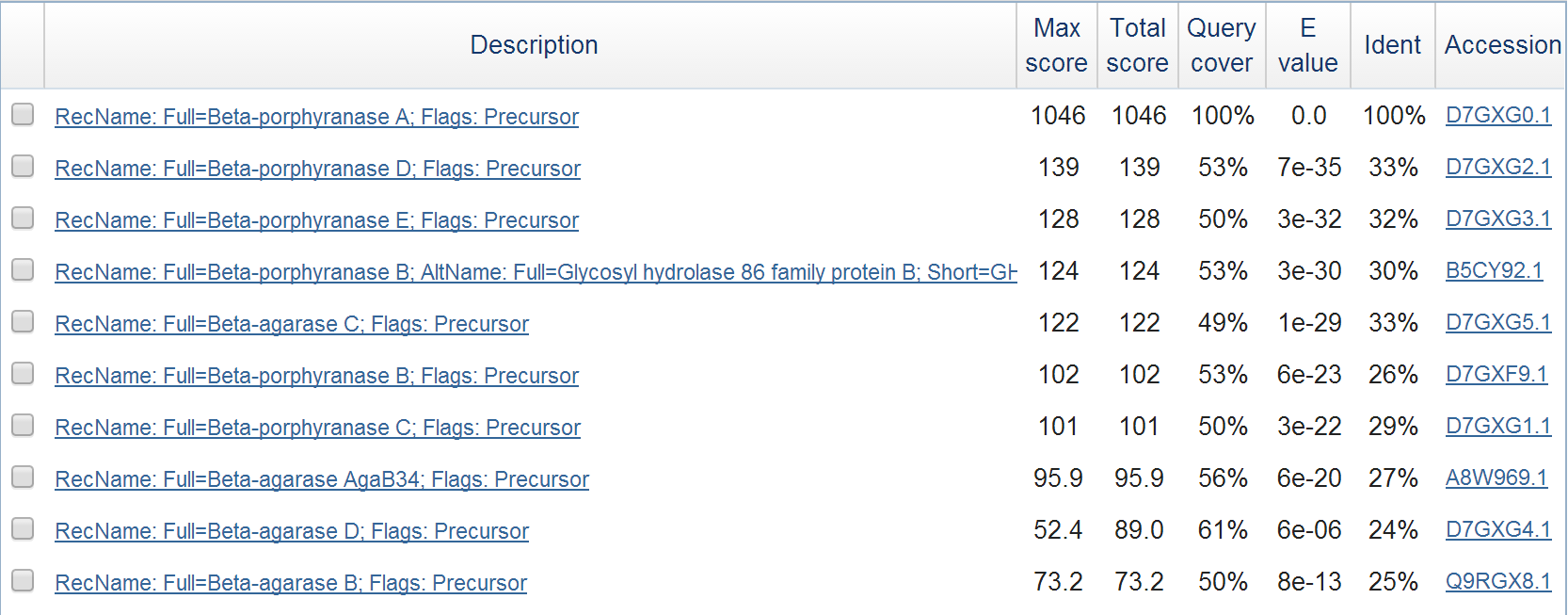
Рис. 1. Результаты работы Protein BLAST
Видно, что выдача BLAST представляет собой таблицу с несколькими колонками
- Поле Description - краткое описание белка.
- Max score - максимальный вес выравнивания.
- Total score - общий вес, он может отличаться от максимального, если в последовательностях выравнялось несколько участков.
- Query cover - покрытие последовательности, т.е какая часть последовательности покрыто выравниванием.
- E-value - величина, характеризующая математическое ожидание того, что в случайно м банке аминокислотных последовательностей такой же длины и такого же аминокислотно состава найдется последовательноость, дающая такой или более вес. Соответсвенно, чем она меньше, тем больше мы можем доверять выравниванию.
- Ident - процент совпавших позиций в двух последовательностях.
- Accession - код белка(AC) в базе данных.
Из находок мною была выбрана β-агараза C. Этот белок такде принадлежит Zobellia galactinovorans и участвует в расщеплении порфиранов.
Её характеристики, а также характеристики лучшей находки приведены ниже, в таблице 1.Таблица 1. Характеристика выбранной мною находки и лучшей находки.
| Description | Max score | Total score | Query cover | E value | Ident | Accession |
| RecName: Full=Beta-agarase C; Flags: Precursor | 122 | 122 | 49% | 1,00E-29 | 33% | D7GXG5.1 |
| RecName: Full=Beta-porphyranase D; Flags: Precursor | 139 | 139 | 53% | 7,00E-35 | 33% | D7GXG2.1 |
Мною была построена карта локального сходства моей последовательности и находки. Для этого использовалась функция BLAST - "align two or more sequences". Карта локального сходства представлена на рис.2
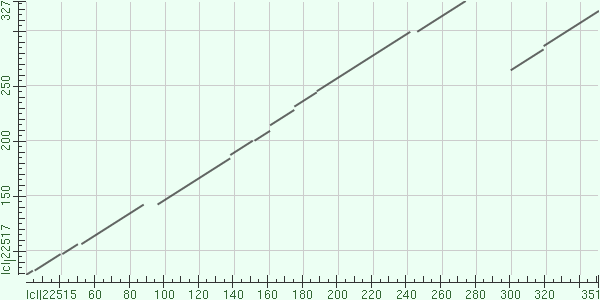
Рис. 2. Карта локального сходства β-агаразы C и β-порфираназы A из бактерии Zobellia galactinovorans
Из карты лкоального сходства видно, что похожи участки 79-327 β-агаразы C и 21-274 β-порфираназы A, при этом, судя по всему расхождение между белками произошло достаточно давно давно, т.к наличествует много вставок. Кроме этих участков совпадают участки 265-318 β-агаразы C и 301-351. Таким образом можно предположить, что в β-порфираназе A произошла дупликация конечного участка( так как получается, что участок 220-275 и 300-351 β-порфираназы A гомологичны друг другу).
Далее мною было проверено, какая часть находок принадлежит эукариотам. Выдача BLAST представлена на рис.3. Видно, что даже самое маленькое E-value все равно не полволяет говорить о гомологичности β-порфираназы А и этих находок.
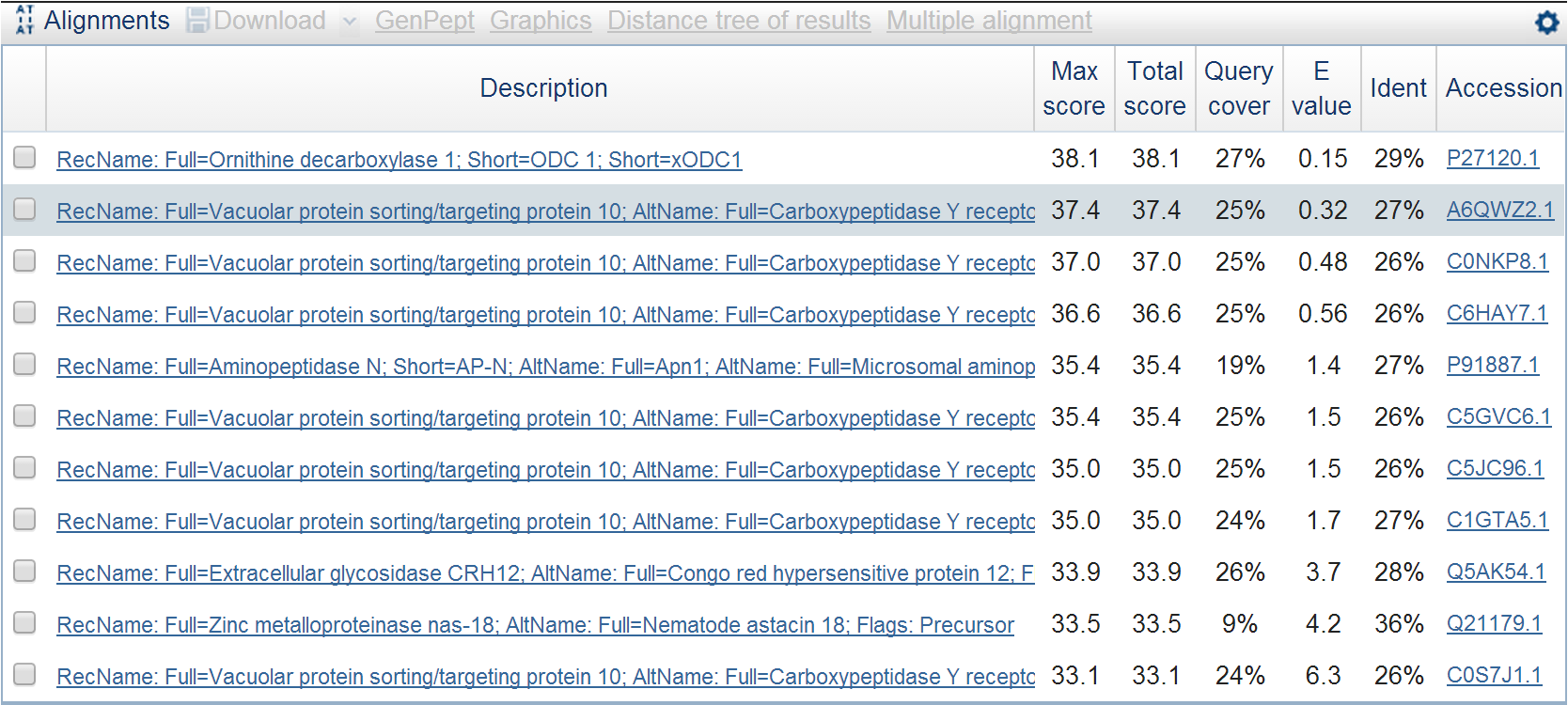
Рис. 3. Выдача BLAST с фильтром по эукариотам.
Далее был проиведен поиск среди находок тех, которые входят в отдел бактерий Firmicutes. Выдачу можно увидеть на рис.4. Среди этих находок наилучшей является β-глюканаза бактерии Bacillus circulans. Интересно, что альтернативное название этого белка - ламинариназа.
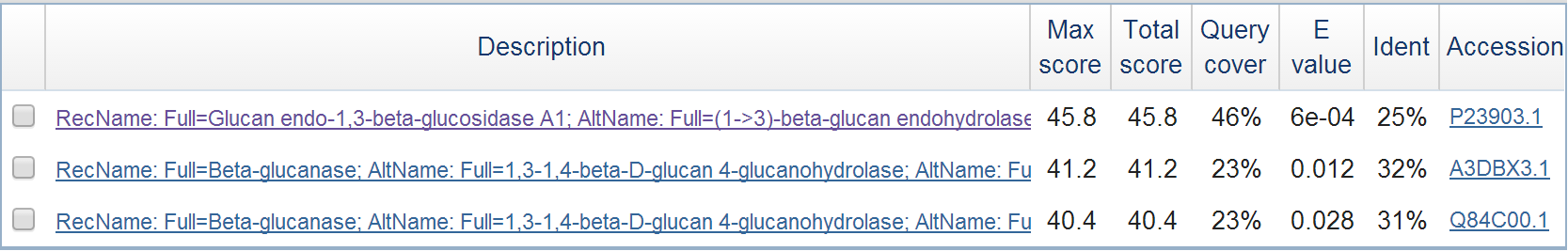
Рис. 4. Выдача BLAST с фильтром по отделу бактерий Firmicutes.
После этого мною были скачены последовательности находок с E-value < 0.0001 и длиной, близкой к длине последовательности моего белка и произведено множетсвенное выравнивание. Скачать файл в формате .fasta можно по этой ссылке. Скачать множественное выравнивание в формате .jar можно по этой ссылке. Скачать файл множетсвенного выравнивания в формате .jar можно по этой ссылке. Процент консервативных и функционально консервативных колонок приведен в таблице 2
Таблица 2. Процент консервативных и функционально консервативных колонок.
| Всего колонок | Консервативных | Функционально консервативных |
| 1100 | 1% | 1% |
Поиск гомологов фминокислотной последовательности белка β-порфираназы А из бактерии Zobellia galactinovorans в базе данных Refseq.
После этого мною был произведен поиск гомологов моего белка в базе данных Refseq. Результаты оказались намного лучше, к примеру, если "cover" у прежних находок не превышало 70%, то здесь нашлось достаточно много находок с значением покрытия около 90%(Окно выдача бласта представлено на рис.5). Кроме того, находок оказалось намного больше.
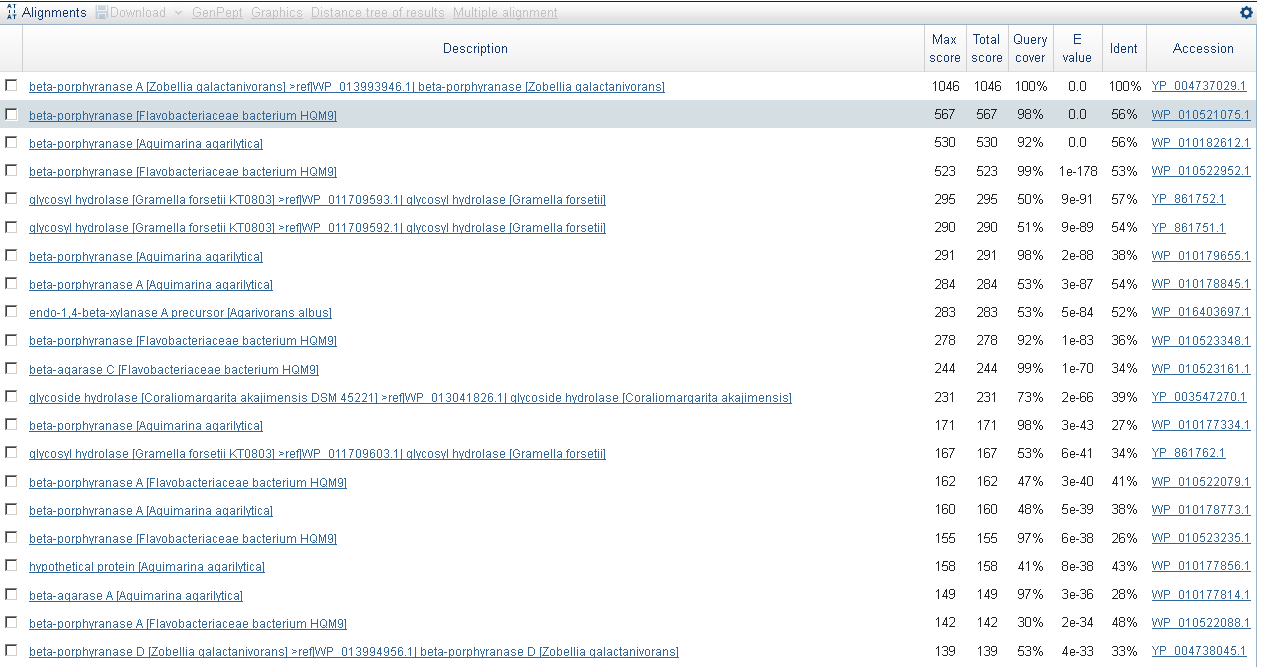
Рис. 5. Окно выдачи BLAST при поиске в базе данных Refseq.
Я выбрал 15 последовательностей(среди них и сам белок) с cover > 70%( файл с их последовательностью можно скачать по этой ссылке) и построил их множественное выравнивание(ссылка на множественное выравнивание в формате .jar)
Поиск гомологов нуклеотидной последовательности, кодирующей белок β-порфираназы А из бактерии Zobellia galactinovorans в нуклеотидной базе данных Refseq.
После этого мною был произведен поиск гомологов моего белка в нуклеотидной базе данных Refseq. Результаты можно увидеть на рис. 6.. Нашлась только исходная последовательность.
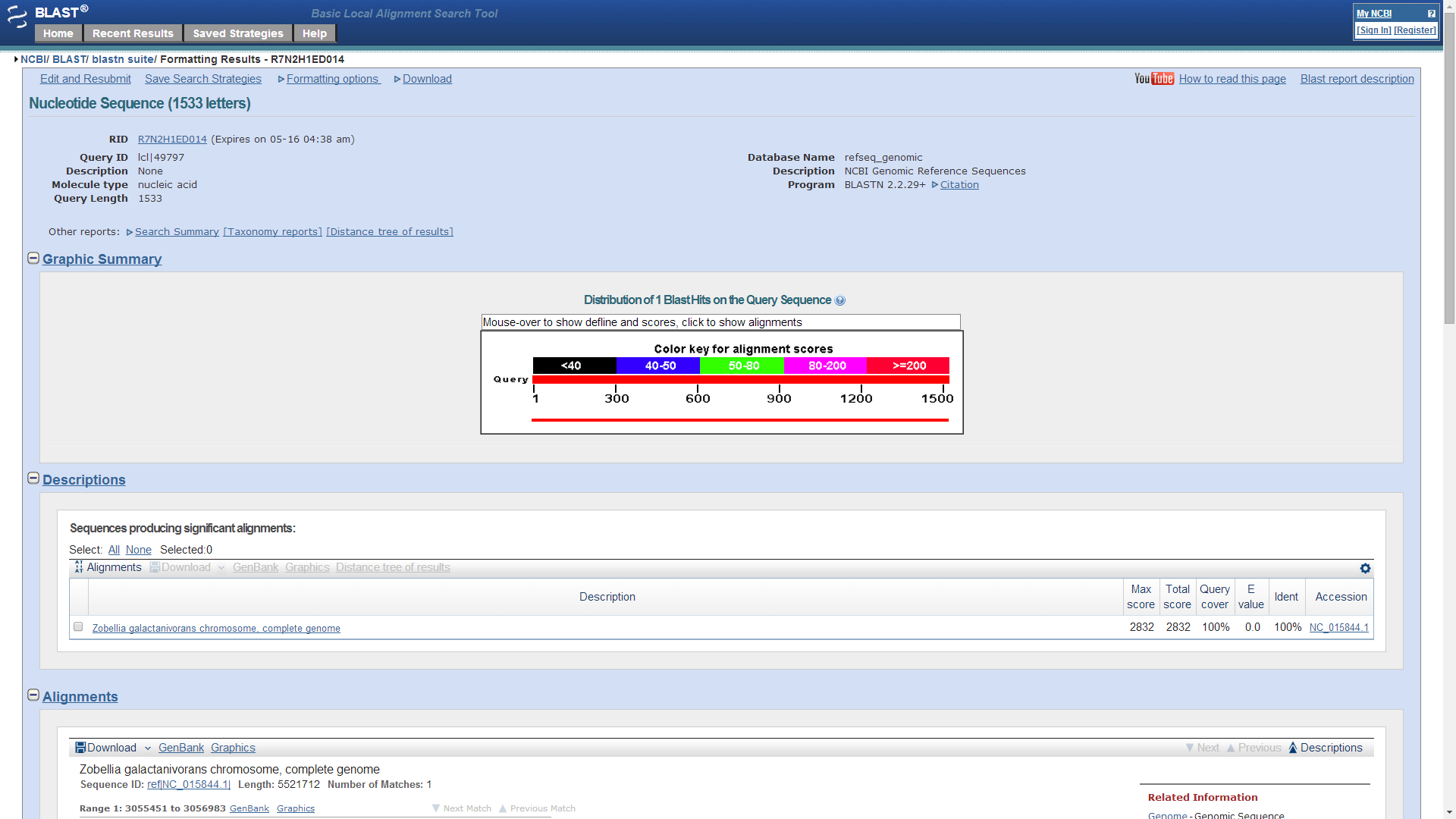
Рис. 6. Окно выдачи BLAST при поиске в нуклеотидной базе данных Refseq. Ничего найдено не было(кроме исходной последовательности)
