Мотивы, MEME & ProSite
Поиск мотивов с помощью MEME.
Мотив - консервативный участок группы последовательностей.
Сервис Prosite предназначен для нескольких целей:
- Поиск мотивов в последовательностях, данных на вход.
- Поиск последовательностей по введенному вами паттерну
- Поиск в последовательностях, данных на вход, последовательностей, удолетворяющих введенному паттерну.
Ввод может осуществляться двумя способами - либо ввести в окно последовательности( не более 10 последовательностей), либо дать идентификатор базы, хранящей последовательности( размер не более 16MB). Создать подобную базу на Prosite можно легко, пройдя по ссылке, появляющейся пи выборе соответсвующей опции. Второй вариант очень выгоден, когда необходимо искать что-либо только в строго определенных последовательностях. Также выгодна третья опция, с помощью неё в дальнейшем будет проверяться нахождение исходных последовательностей по нашему мотиву(для избежания возможных опечаток).
В работе использовалась часть последовательностей из одного из предыдущих заданий. Файл, содержащий последовательности, в формате .fasta можно скачать по этой ссылке
С помощью опции программы MEME "Submit PROTEIN sequences to scan them against the PROSITE collection of motifs"(соответсвенно первой в списке, приведенном выше) были найдены мотивы, встречающиеся в последовательностях.
Скачать файл в формате .pdf с результатами выдачи можно по этой ссылке
. Для дальнейших заданий были выбран мотив - PS00260 GLUCAGON(Glucagon / GIP / secretin / VIP family signature).Построение сильного и слабого паттернов для одного из найденных мотивов.
Паттерн - аналог регулярного выражения - выражение, задающее определенный участок в последовательностях. Различают сильные и слабые паттерны - первые с высокой вероятностью находят только гомологичные последовательности, но при этом возможна потеря каких-то немного отличающихся белков, также являющихся гомологичными, вторые - выдают много ложных находок, но зато среди них есть и все гомологичные.
Процесс составления сильного и слабого паттернов отражен в таблице 1
Таблица 1. Составление сильного и слабого паттерна.
| Номер паттерна | Паттерн | Находки в Swissprot | Находки в TrEMBL | Нахождение изначальных последовательностей | Комментарии |
| 1 | [LIA]-x-[FIY]-[YF]-[FILY]-[DN]-[GN]-[KER]-x(6)-[PF] | 38 | 1869 | да | Слабый паттерн, находятся гомологичные последовательности, но не только они |
| 2 | [FY]-[HY]-[VIT]-Y-[GA]-x-[WY]-W-A(0,1)-[SD]-x(2)-[KET]-[LIA]-x-[FIY]-[YF]-[FILY]-[DN]-[GN]-[KER]-x(6)-[PF] | 5 | 10 | да | Был добавлен участок выравнивания, находящийся рядом с мотивом и схожий у всех последовательностей. Паттерн можно отнести к сильным, т.к как он находит только последовательности, у которых можно предположить гомологию. Файл с результатами одновременного поиска по Swissprot и TrEMBL можно скачать по этой ссылке |
| 3 | [KER]-W-[EIVK]-[KL]-[VIN]-[EP]-[QGNSD]-[LVMF]-S-D-E-F-[NED]-[GD]-x(2)-[LI]-D-[TLE]-x-K-W | 5 | 6 | да | Сильный паттерн, но многие требования не обоснованы. Находит только исходные последовательности. Построен не по мотиву, найденном Prosite, а по одному из блоков выравнивания.. Просто пример паттерна, который необоснаванно сильный. Таким можно пользоваться только если задача состоит не в поиске гомологичных последовательностей, а в чем-то другом(к примеру, есть сайт, на котором хранятся какие-то определенные данные об этих белках, и, чтобы не писать все последовательности белков достаточно просто указать паттерн( удобно для скриптов и т.д.)) |
Результаты обязательного задания
Результаты обязательного задания можно скачать в виде проекта JalView по этой ссылке. Использованный в задании мотив выделен синим цветом.
Поиск лейциновых молний с помощью сильного паттерна
С помощью паттерна L-x(6)-L-x(6)-L-x(6)-L-x(6)-L был проихведен поиск в банке PDB
. Было выбрано три белка и рассмотрены их структуры на предмет наличия лейциновых молний. В двух из трех они были найдены.(см. рис. 1, рис. 2 и рис.3)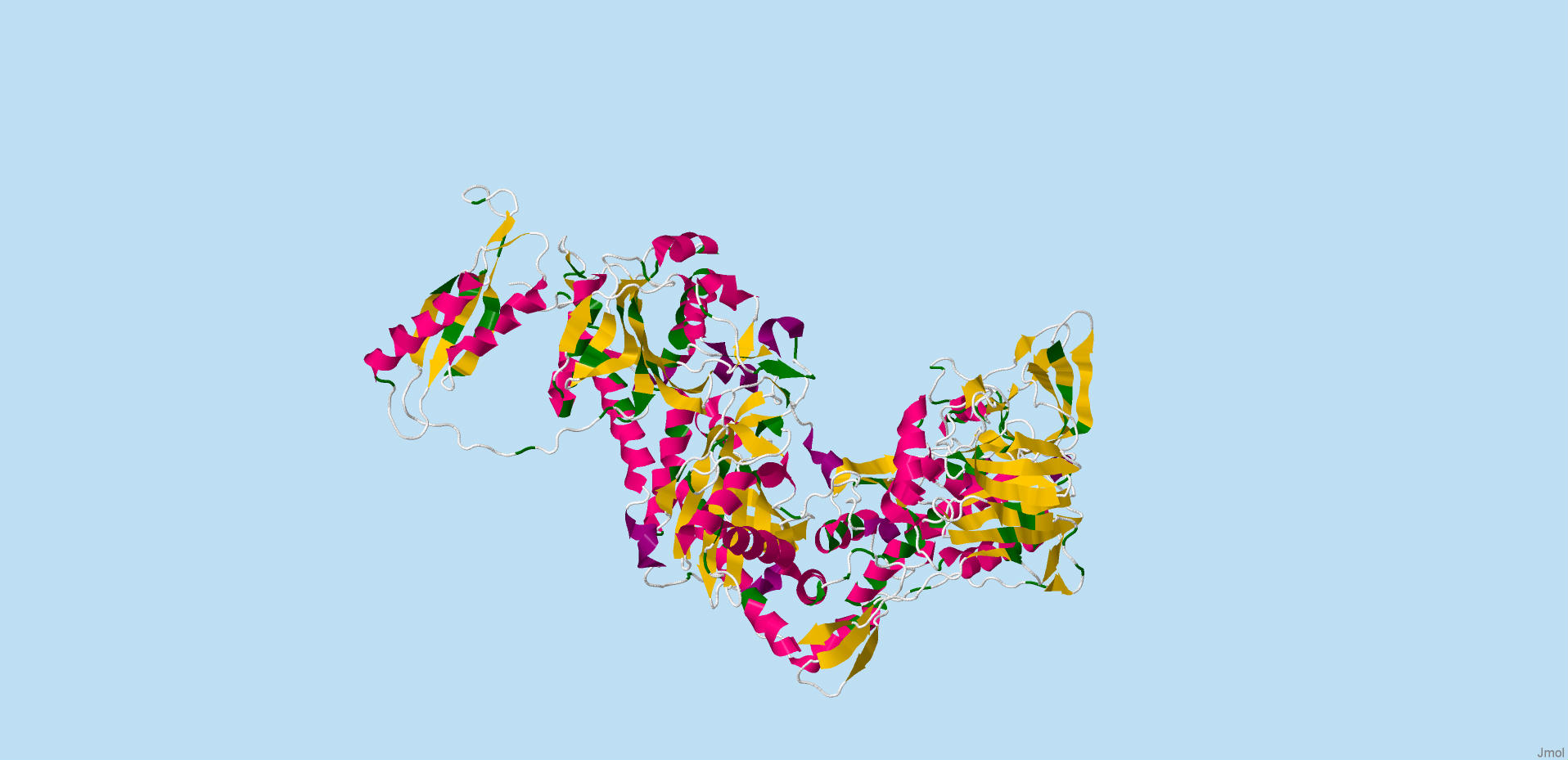
Рис. 1. Белок с идентификатором 1B70 в PDB. Лейциновые молнии не найдены.

Рис. 2. Белок с идентификатором 1C1G в PDB. Лейциновые молнии найдены. Лейцины покрашены в зеленый цвет
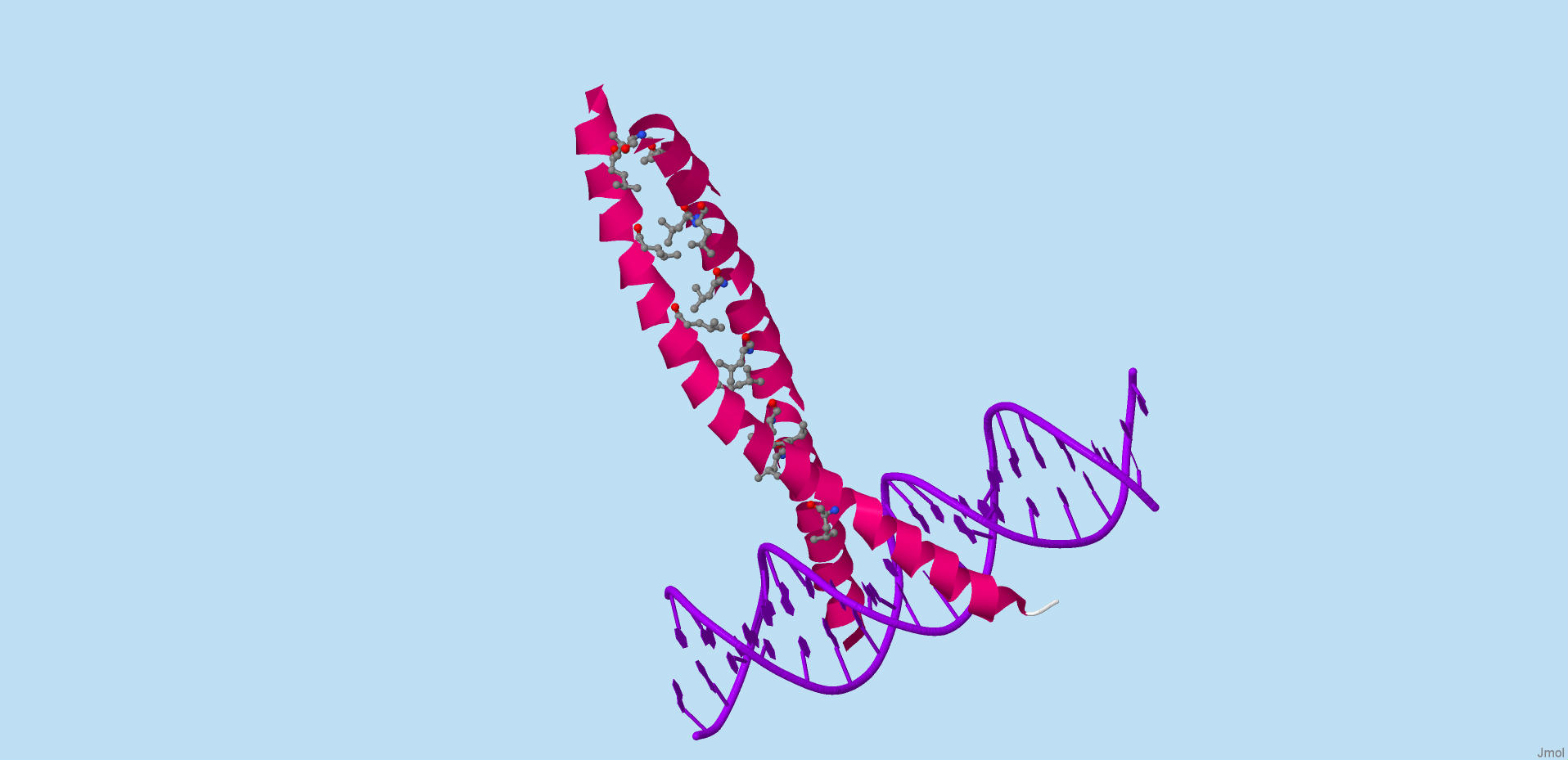
Рис. 3. Белок с идентификатором 1FOS в PDB. Лейциновые молнии найдены. Лейцины выделены с помощью cpk.
Поиск английского слова из 6 букв в Swissprot.
Производился поиск по имени "Alicia"(паттерн, соответственно - A-L-I-C-I-A). Результат можно увидеть на рис. 4. Учитывая, что в банке Swissprot суммарно около 200млн аминокислот в последовательностях и используя статистику Swissprot можно посчитать, сколько встретится таких подпоследовательностей:
8.26*9.66*5.95*1.37*5.95*8.26*2*10^8*10^(-12)=6.3 последовательности. В нашем случае встретилось 3.
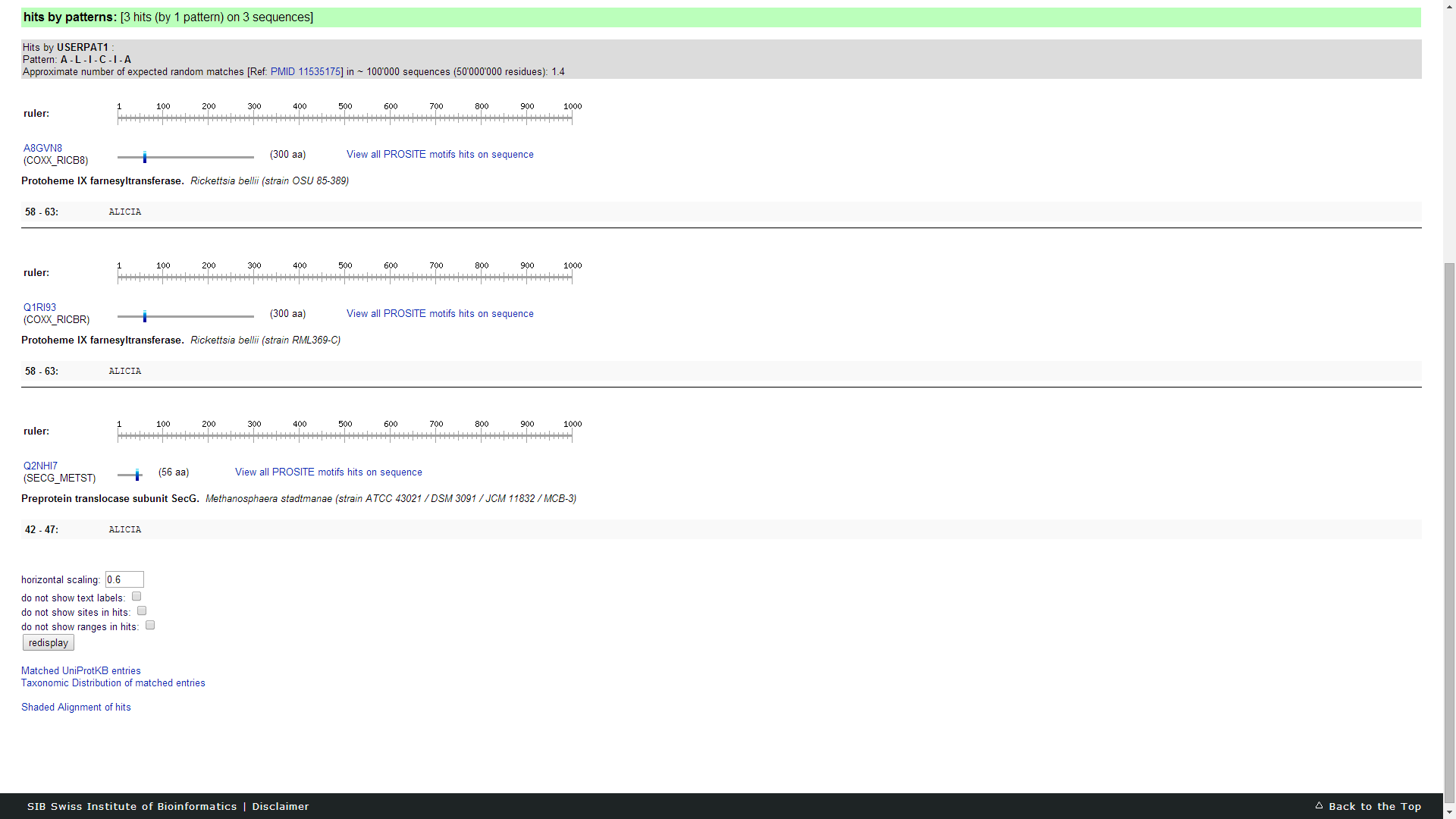
Рис. 4. Результат поиска по паттерну A-L-I-C-I-A
Поиск мотивов в последовательностях при помощи программы MEME
Был произведен поиск мотивов в последовательностях с помощью программы MEME. Результат можно увидеть здесь. Все найденные мотивы встречаются во всех последовательностях по одному разу каждый. В проекте, который можно скачать по этой ссылке во множестенном выравнивании синим цветом отмечены места расположения мотивов в последовательностях. Можно увидеть, что они практически совпадают с блоками в выравнивании.
