BLAST и PSI-BLAST.
На выбор было предложено несколько белков. Случайным образом мною был выбран белок Q3U7X3. В Uniprot можно найти его название: BCL2-like 11 (apoptosis facilitator( регулятор апоптоза)). Белок Bcl-2-like protein 11 в человеке участвует в апоптозе в нейронах и лимфоцитах. Кроме того, в слуае мышей показана его ключевая роль в негативной селекции Т-лимфоцитов.[1]
. После первой итерации PSI-BLAST в числе прочего выдал консервативные домены, которые предположительно есть в данном белке. На рисунке 1 представлена подробная информация об этом.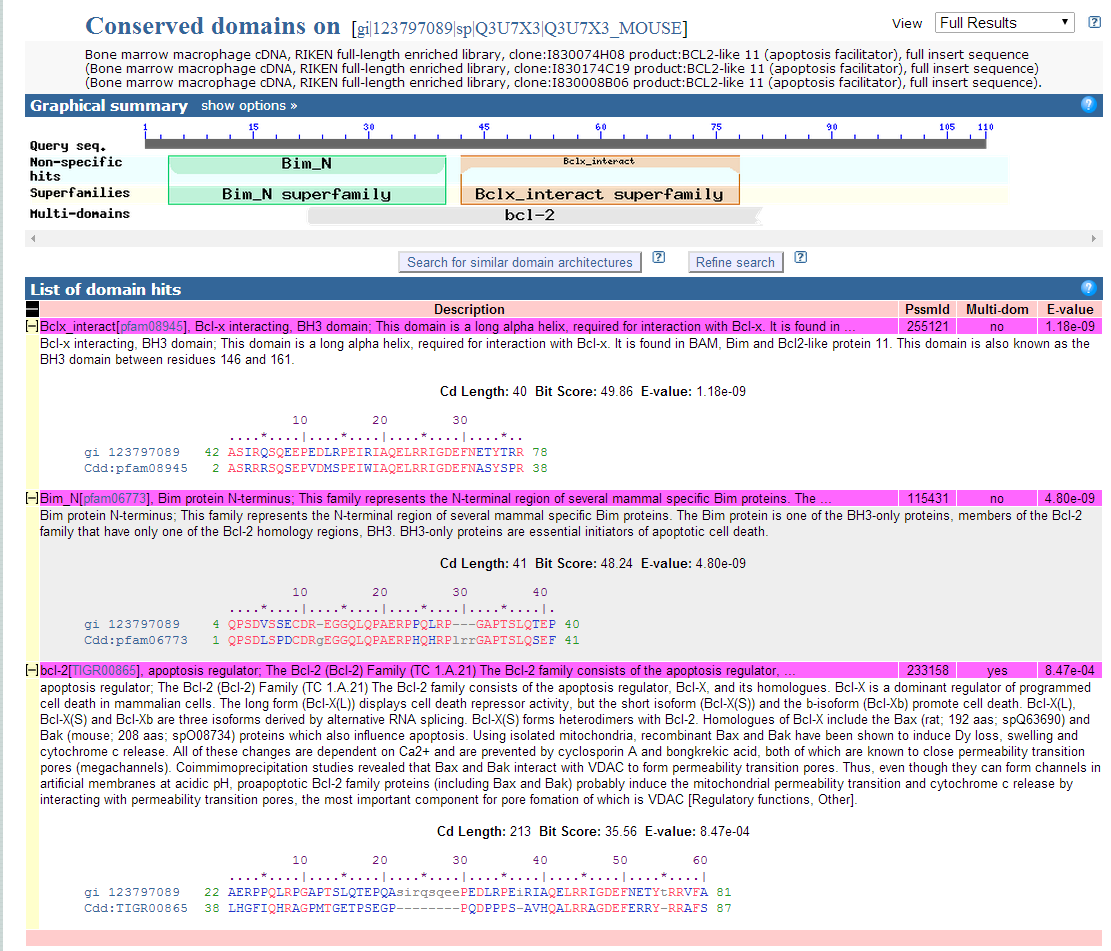
Рис. 1. Консервативные домены в белке Q3U7X3.
Затем, после того, как мною были выбраны белки, с участием которых будет происходить итерация 2 я запустил Psi-Blast еще раз. Таким образом было проведено 13 итераций, пока я не убедился, что новых белков не добавляется. Результаты итераций представлены в таблице 1.
Таблица. 1. Результаты итераций в Psi-Blast.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор лучшей находки ниже порога | E-value этой находки | Идентификатор худшей находки выше порога | E-value этой находки |
| 1 | 245 | bcl2-like 11 (apoptosis facilitator) [Xenopus laevis] | 0.009 | Select seq ref|XP_005139758.1| PREDICTED: bcl-2-like protein 11-like [Melopsittacus undulatus] | 0.005 |
| 2 | 247 | twin-arginine translocation pathway signal protein [Paracoccus zeaxanthinifaciens] | 0.11 | BCL2-like 11 [Danio rerio] | 3e-05 |
| 3 | 247 | PREDICTED: uncharacterized protein LOC100377129 [Saccoglossus kowalevskii] | 0.052 | PREDICTED: bcl-2-like protein 11-like [Melopsittacus undulatus] | 1e-07 |
| 4 | 249 | PREDICTED: uncharacterized protein LOC102776737 [Neolamprologus brichardi] | 0.006 | PREDICTED: bcl-2-like protein 11-like [Oreochromis niloticus] | 5e-04 |
| 5 | 253 | hypothetical protein BRAFLDRAFT_65824 [Branchiostoma floridae] | 0.025 | PREDICTED: bcl-2-like protein 11 isoform X2 [Poecilia formosa] | 5e-04 |
| 6 | 253 | hypothetical protein BRAFLDRAFT_65824 [Branchiostoma floridae] | 0.013 | PREDICTED: bcl-2-like protein 11 isoform X5 [Echinops telfairi] | 3e-08 |
| 7 | 254 | PREDICTED: uncharacterized protein LOC100377129 [Saccoglossus kowalevskii] | 0.048 | hypothetical protein BRAFLDRAFT_65824 [Branchiostoma floridae] | 0.003 |
| 8 | 254 | PREDICTED: uncharacterized protein LOC100377129 [Saccoglossus kowalevskii] | 0.048 | hypothetical protein BRAFLDRAFT_65824 [Branchiostoma floridae] | 0.003 |
| 9 | 254 | PREDICTED: uncharacterized protein LOC100377129 [Saccoglossus kowalevskii] | 0.048 | hypothetical protein BRAFLDRAFT_65824 [Branchiostoma floridae] | 0.001 |
| 10 | 254 | PREDICTED: uncharacterized protein LOC100377129 [Saccoglossus kowalevskii] | 0.047 | PREDICTED: bcl-2-like protein 11 isoform X5 [Echinops telfairi] | 1e-06 |
| 11 | 254 | PREDICTED: uncharacterized protein LOC100377129 [Saccoglossus kowalevskii] | 0.019 | PREDICTED: bcl-2-like protein 11 isoform X5 [Echinops telfairi] | 6e-07 |
| 12 | 254 | PREDICTED: uncharacterized protein LOC100377129 [Saccoglossus kowalevskii] | 0.023 | PREDICTED: bcl-2-like protein 11 isoform X5 [Echinops telfairi] | 7e-07 |
| 13 | 254 | PREDICTED: uncharacterized protein LOC100377129 [Saccoglossus kowalevskii] | 0.023 | PREDICTED: bcl-2-like protein 11 isoform X5 [Echinops telfairi] | 1e-06 |
В ходе этих итераций мною выбиралась часть белков, не прошедших порог E-value<0.005(до 7 итерации). Выбор осуществлялся на основании схожести названий белков первой и второй группы, покрытие белком последовательности Q3U7X3( при этом последний фактор играл роль только при добавлении новых последовательностец, старые не удалялись, так как судя по информации с Википедии и Uniprot размер белков "семейства" может существенно варьировать.) Чтобы облегчить читателю просмотр результатов, все последовательности, добавлявшиеся на разных этапах собраны в файле в формате .jar. В каждом окне проекта JalView находятся последовательности, добавленные на определенном этапе(окна проименованы от ckl1 до ckl6). Скачать файл можно по этой ссылке.
После 13 итерации я окончательно убедился, что новых белков добавляться не будет, а старые не исчезнут из списка(до этого подозрение вызывал сначала худший из белков, прошедших порог, затем лучший из белков, не прошедших).
Затем было построено множественное выравнивание находок, прошедших порог. Однако часть выравнивание части белков(в том числе и "hypothetical protein BRAFLDRAFT_65824 [Branchiostoma floridae]") было некачественным. Поэтому в ходе нескольких последовательных шагов они удалялись из списка и с оставшимися белками строилось новое множественное выравнивание с помощью сервиса MUSCLE. На финальном шаге был выбран участок, встречающийся почти у всех белков и, в том числе, белке Q3U7X3. Белки, у которых этого участка не было, были удалены(т.к оказывали достаточно сильное негативное влияние на выравнивание), оставшиеся были выравнены снова. Все выравнивания до конечного были добавлены в отдельный проект Jalview, где они пронумерованы соответсвующим образом. Скачать этот файл можно по этой ссылке. Также его можно увидеть на рисунке 2. Последнее множественное выравнивание было сохранено в отдельном проекте, скачать который можно по этой ссылке.

Рис. 2. Окончательное множественное выравнивание.
Затем в выравнивание для большего удобства были добавлены консервативные домены, найденные программой, два из них были связаны с PDB-структурами, построенными с помощью SWISS-MODEL(структуру третьего построить не удалось). Участки, общие для большинства последовательностей, заслуживающие доверия и не нуждающиеся в улучшении, были помечены "+". Скачать файл проекта в формате .jar можно по этой ссылке.
Ссылки
