Задание №1. Построение A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA
Построение A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA производилось при помощи программы fiber из пакета 3DNA. Для построения использовался участок gatc повтроренный 5 раз (в случае Z-формы использовалась последовательность gc с 10-и кратным повтором).
Результат представлен в формате pdb.
 A-форма пятикратного повтора gatc в формате pdb
A-форма пятикратного повтора gatc в формате pdb
 B-форма пятикратного повтора gatc в формате pdb
B-форма пятикратного повтора gatc в формате pdb
 Z-форма пятикратного повтора gatc в формате pdb
Z-форма пятикратного повтора gatc в формате pdb
Задание №2.Средства JMol для работы со структурами нуклеиновых кислот.
Выделение разных атомов и химических группировок,при использовании предопределеннх множеств JMol.
Для работы была выбрана A-форма из задания №1. Написан скрипт  Скрипт для получения Рис. 1, Рис. 2, Рис. 3.
Скрипт для получения Рис. 1, Рис. 2, Рис. 3.
jmol_script.txt

Рис. 1 A-форма цепи днк состоящей из 2 цепей, прямая цепь представляет 5 кратый повтор последовательности gatc.
Вид сверху на ДНК: сахарофосфатный остов ДНК показан красным цветом, все азотистые основания показаны синим цветом, но все аденины зеленым. Атом N7 во всех гуанинах показан фиолетовой относительно крупной сферой, а в первом по последовательности гуанине - относительно крупной оранжевой сферой.
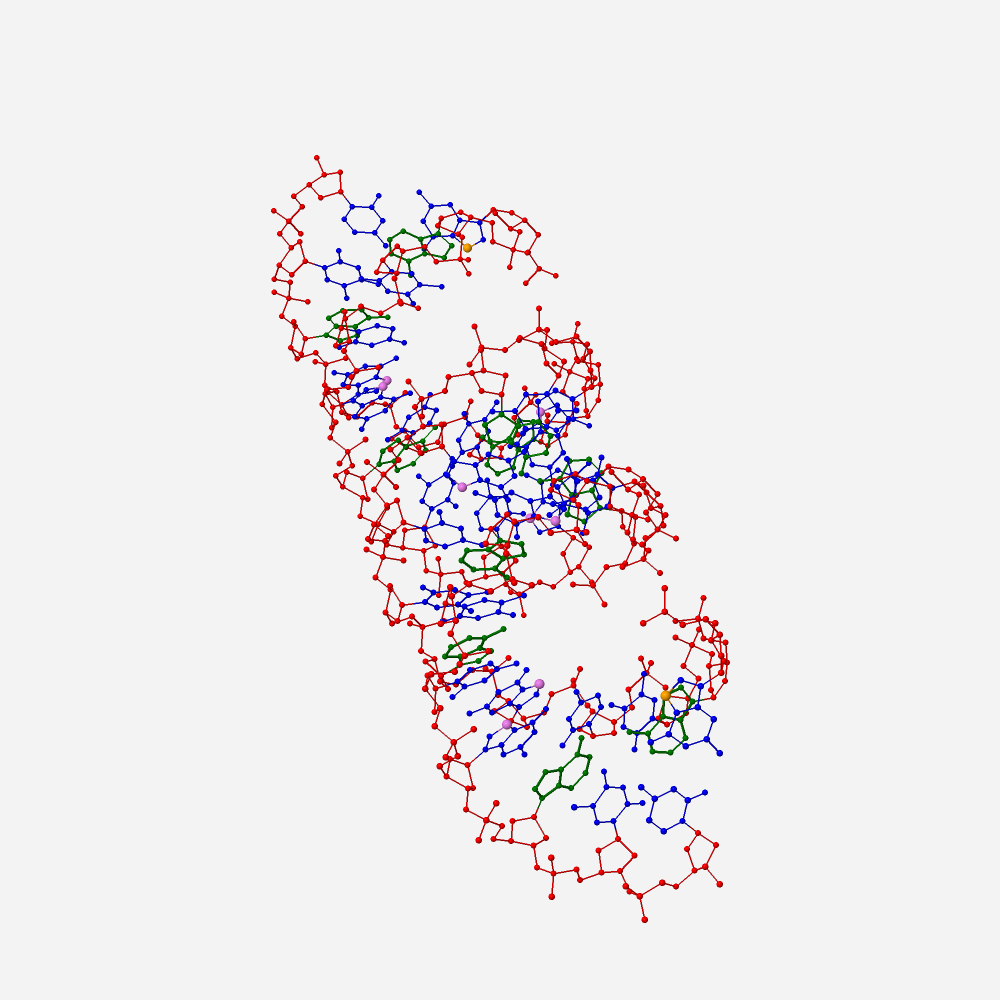
Рис. 2 A-форма цепи днк состоящей из 2 цепей, прямая цепь представляет 5 кратый повтор последовательности gatc.
Вид под углом на ДНК: сахарофосфатный остов ДНК показан красным цветом, все азотистые основания показаны синим цветом, но все аденины зеленым. Атом N7 во всех гуанинах показан фиолетовой относительно крупной сферой, а в первом по последовательности гуанине - относительно крупной оранжевой сферой.
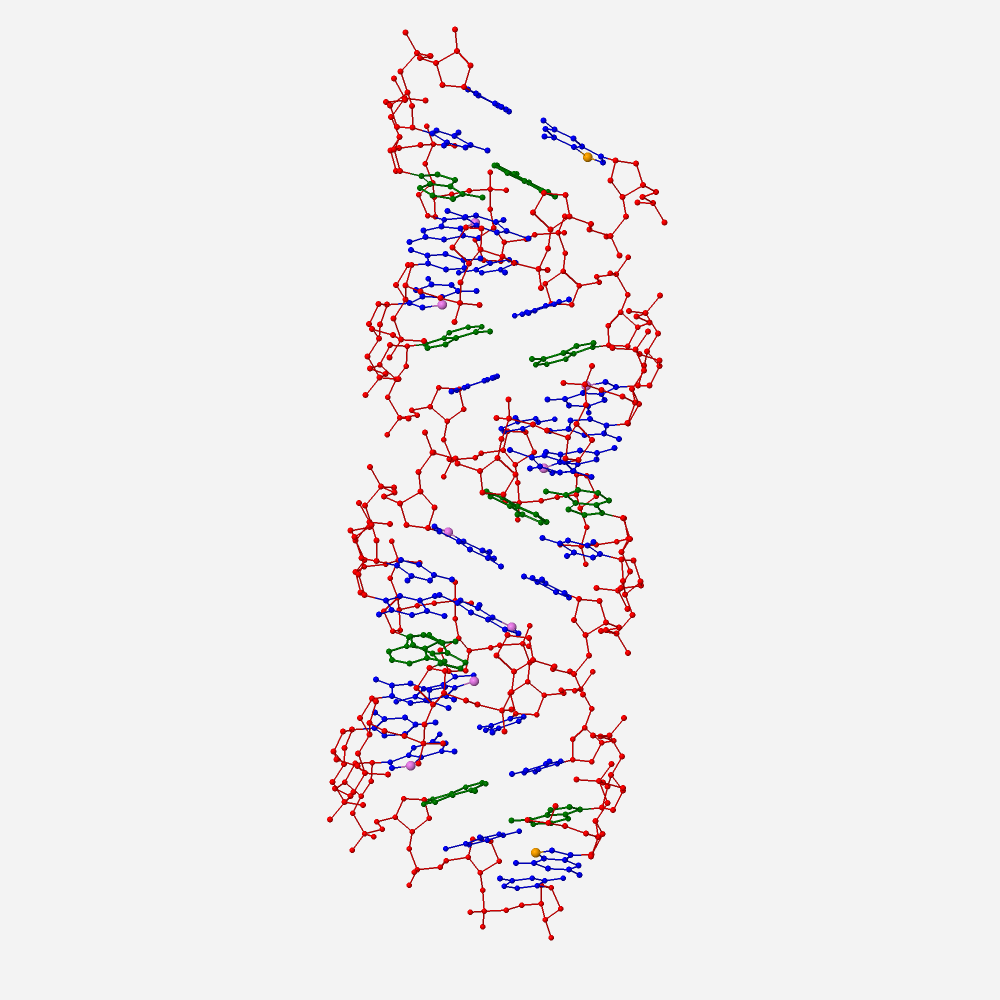
Рис. 3 A-форма цепи днк состоящей из 2 цепей, прямая цепь представляет 5 кратый повтор последовательности gatc.
Вид сбоку на ДНК: сахарофосфатный остов ДНК показан красным цветом, все азотистые основания показаны синим цветом, но все аденины зеленым. Атом N7 во всех гуанинах показан фиолетовой относительно крупной сферой, а в первом по последовательности гуанине - относительно крупной оранжевой сферой.
Получение pdb файлов структур комплексов тРНК и ДНК.
При помощи сервиса получены файлы с pdb идентификаторами 1D5Y и 1EHZ.
Файл  1D5Y содержит Rob фактор транскрипции Escherichia coli в комплексе с ДНК. Этот белок принадлежит к семейству AraC/XylS белков, представители которого участвуют в регуляции транскрипции генов устойчивости к антибиотикам, органическим растворителям и тяжелым металам.
1D5Y содержит Rob фактор транскрипции Escherichia coli в комплексе с ДНК. Этот белок принадлежит к семейству AraC/XylS белков, представители которого участвуют в регуляции транскрипции генов устойчивости к антибиотикам, органическим растворителям и тяжелым металам.
Файл  1EHZ содержит кристаллическую структуру дрожжевой тРНК фенилаланина с разрешением 1.93 , Å
1EHZ содержит кристаллическую структуру дрожжевой тРНК фенилаланина с разрешением 1.93 , Å
Проверка заданных структур ДНК и РНК на наличие разрывов.
Получены изображения средствами Jmol только ДНК или РНК, для анализа на наличие разрывов.
Для получения изображения только ДНК (Рис. 4) и  pdb файла с координатами атомов только ДНК из pdb файла 1D5Y, был написан
pdb файла с координатами атомов только ДНК из pdb файла 1D5Y, был написан  скрипт.
скрипт.
DNA_script.txt
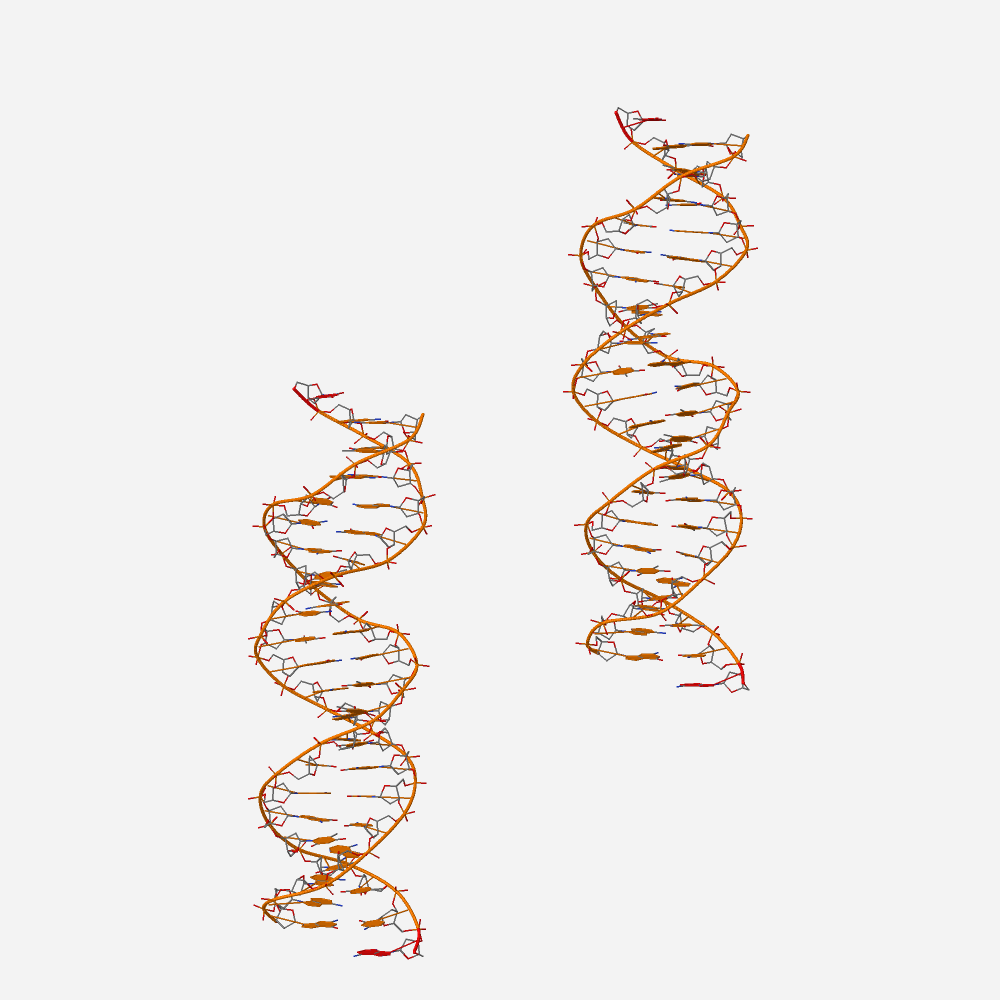
Рис. 4 ДНК цепи из pdb 1D5Y, комбинированное представление cartoon и wireframe. Как видно по рисунку, данные ДНК цепи не содержат разрывов в своей структуре.
Для получения изображения только РНК (Рис. 5) и  pdb файла с координатами атомов только РНК из pdb файла 1EHZ, а так же был написан
pdb файла с координатами атомов только РНК из pdb файла 1EHZ, а так же был написан  скрипт.
скрипт.
RNA_script.txt
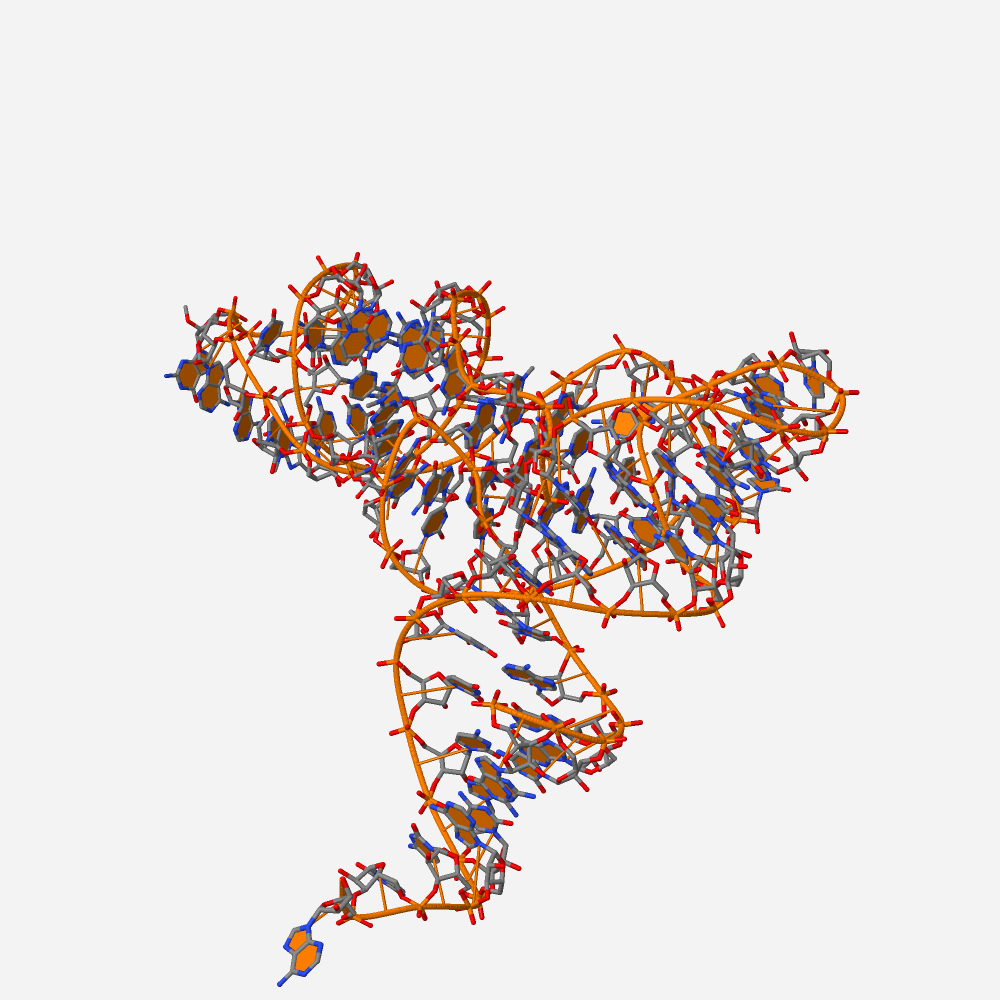
Рис. 5 РНК цепь из pdb 1EHZ, комбинированное представление cartoon и wireframe, покраска выполнена по цветовой схеме cpk. Как видно по рисунку, даная РНК цепь не содержит разрывов в своей структуре.
Задание №3. Сравнение структур 3-х форм ДНК с помощью средств JMol.
Нахождение большой и малой бороздки в ДНК.
В программе Jmol был осуществлен визулальный поиск большой и малой бороздки в B-форме ДНК из Задания №1. Для этого был написан  скрипт, результат работы которого представлен на Рис. 6.
скрипт, результат работы которого представлен на Рис. 6.
DNA_script_groove.txt
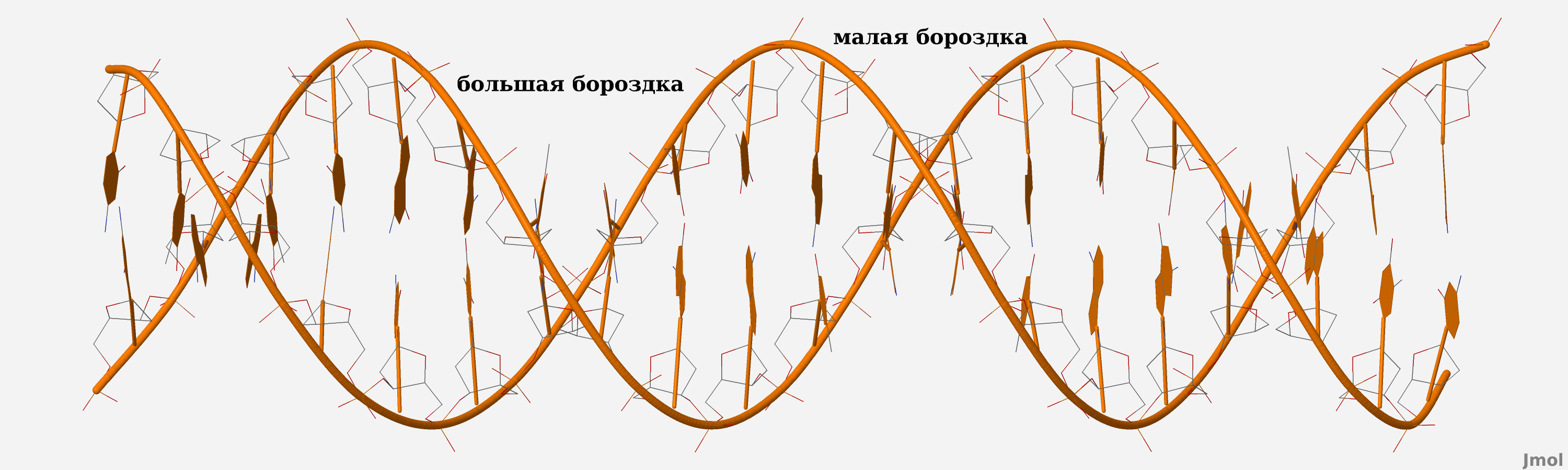
Рис. 6 B-форма ДНК из Задания №1, комбинированное представление cartoon и wireframe, покраска выполнена по цветовой схеме cpk. Подписями отмечено положение большой бороздки и малой бороздки.
Для подробной иллюстрации положения атовов относительно бороздок, рассмотрено азотистое основание аденин, находящееся в 10-м по счету нуклеотиде цепи А B-формы ДНК из задания №1. В программе ChemSketch составлена  структурная формула аденина с пояснениями по поводу бороздок. Результат представлен на Рис. 7.
структурная формула аденина с пояснениями по поводу бороздок. Результат представлен на Рис. 7.

Рис. 7 Структурная формула азотистого основания аденина, с пояснениями по поводу расположения атомов относительно большой и малой бороздки. На рисунке рассмотрен вариант для B-формы ДНК, для A-формы ДНК расположение атомов в стороны большой и малой бороздки инвертируется, а в Z-форме ДНК аденин отсутствует.
Сравнение основных спиральных параметров разных форм ДНК.
Для сравнения основных спиральных параметров разных форм ДНК эти формы были рассмотрены в Jmol'е и для визуализации был написан  скрипт, результаты работы корогого представлены на Рис. 8-10. Итог сравления представлен в Табице 1.
скрипт, результаты работы корогого представлены на Рис. 8-10. Итог сравления представлен в Табице 1.
DNA_script_helix.txt
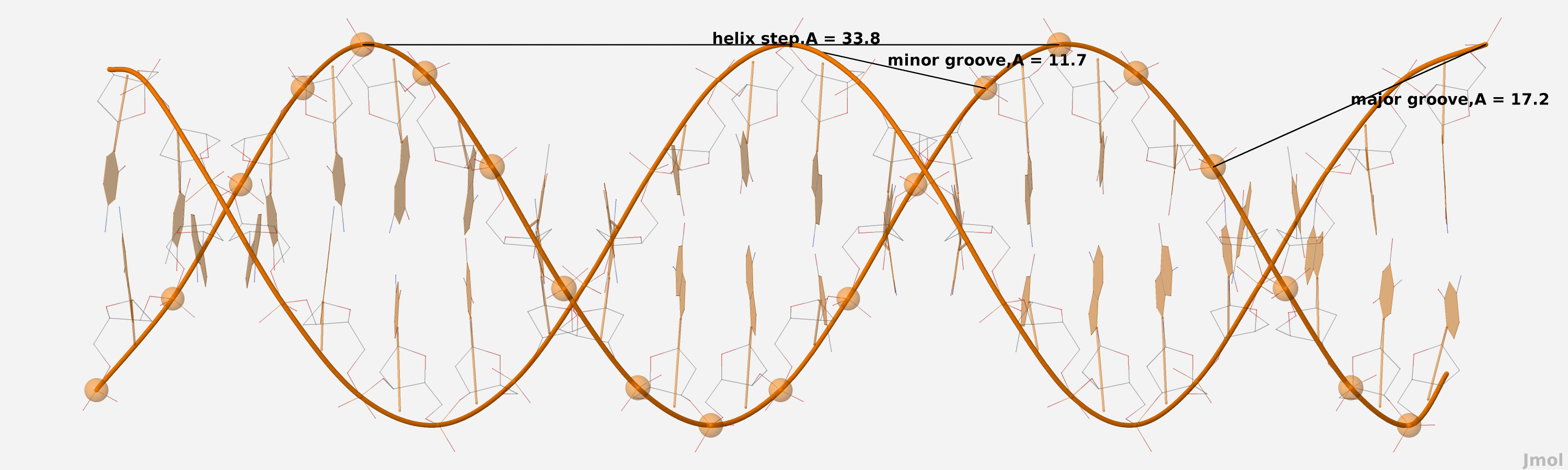
Рис. 8 B-форма ДНК с пояснениями по поводу расположения большой и малой бороздки, их длины, а так же длины витка.
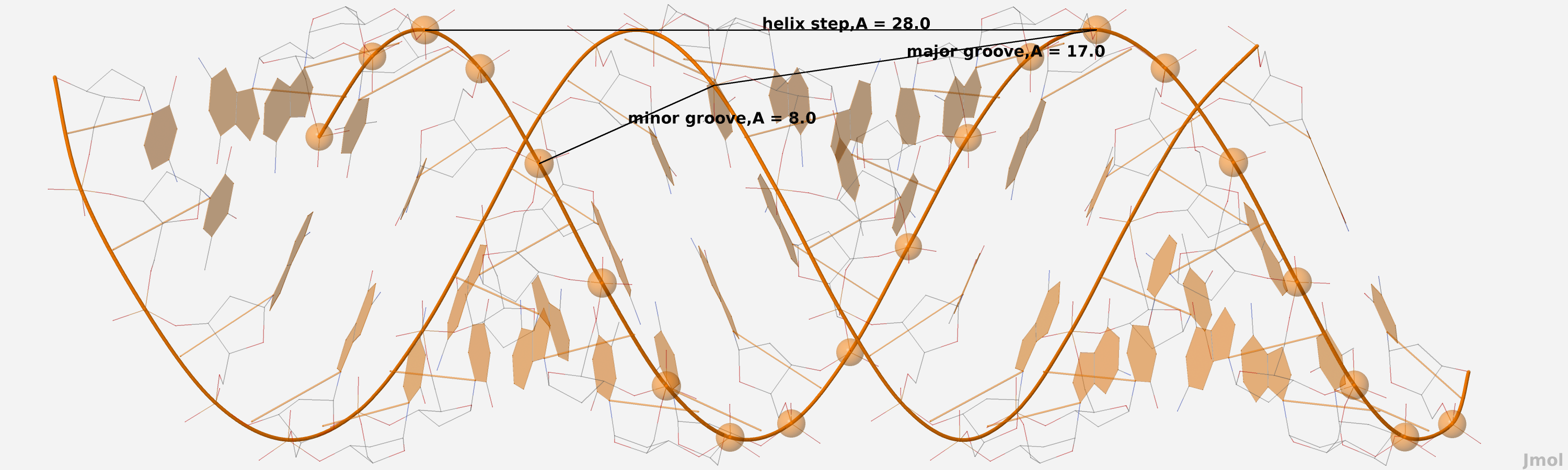
Рис. 9 A-форма ДНК с пояснениями по поводу расположения большой и малой бороздки, их длины, а так же длины витка.
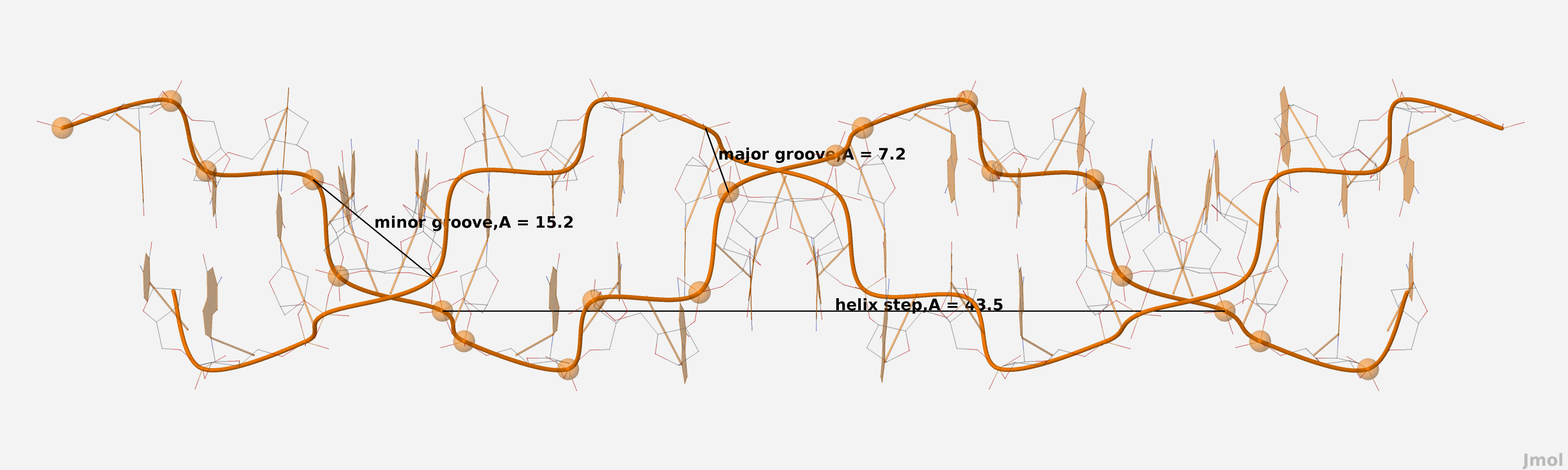
Рис. 10 Z-форма ДНК с пояснениями по поводу расположения большой и малой бороздки, их длины, а так же длины витка.
| Основные спиральные характеристики форм ДНК | A-форма | B-форма | Z-форма |
| Тип спирали (правая или левая) | Правая | Правая | Левая |
| Шаг спирали, Å | 28,0 | 33,8 | 43,5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки, Å | 17,0 ([A]34:B.P) - ([A]10:A.P) | 17,2 ([G]37:B.P) - ([G]1:A.P) | 7,2 ([G]31:B.P) - ([G]13:A.P) |
| Ширина малой бороздки, Å | 8,0 ([G]25:B.P) - ([A]10:A.P) | 11,7 ([A]34:B.P) - ([T]11:A.P) | 15,2 ([C]24:B.P) - ([G]17:A.P) |
Сравнение торсионных углов в структурах А- и В-форм.
Вначале был подробно рассмотрен слайд учебной презентации, посвещенный торсионным углам. (Рис. 11)
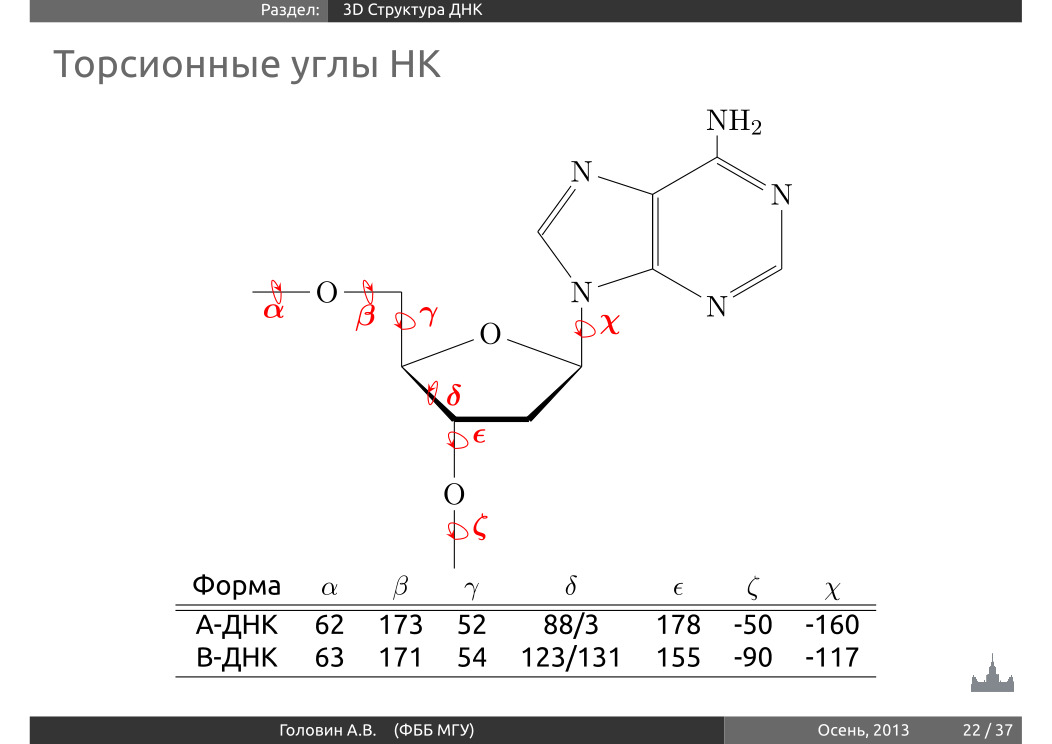
Рис. 11 Расположения и значения торсионных углов на примере аденина в составе A- и B- формы ДНК.
Для сравнения торсионных углов в аденине в разных формах ДНК (A- и B- форма) был написан  скрипт, результаты работы корогого представлены на Рис. 12-13. Итог сравления представлен в Табице 2.
скрипт, результаты работы корогого представлены на Рис. 12-13. Итог сравления представлен в Табице 2.
angles.txt
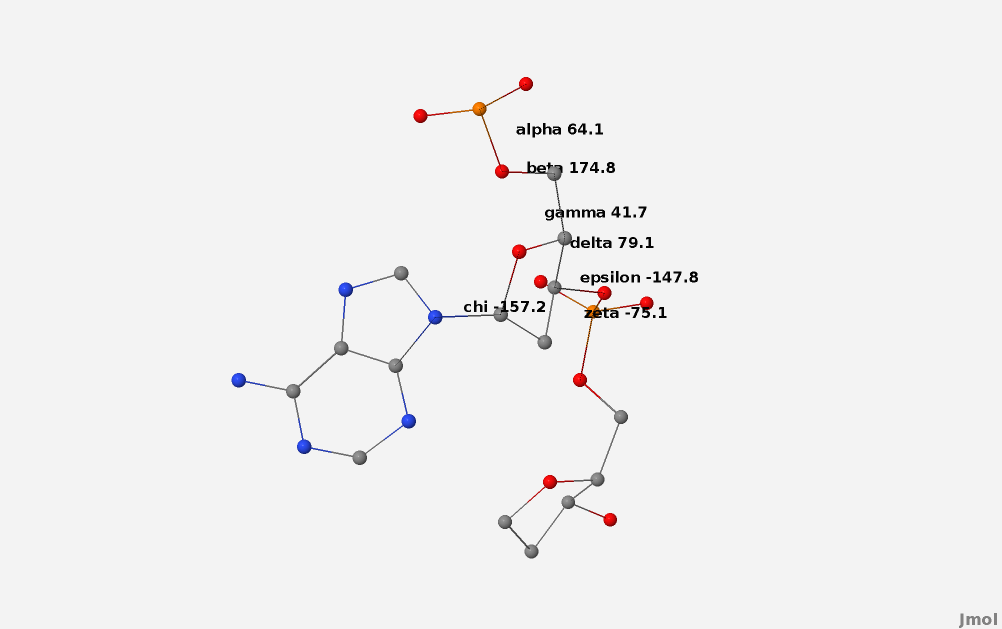
Рис. 12 Структурная формула азотистого основания аденина в A-форме ДНК, с пояснениями по поводу расположения и значения торсионных углов.
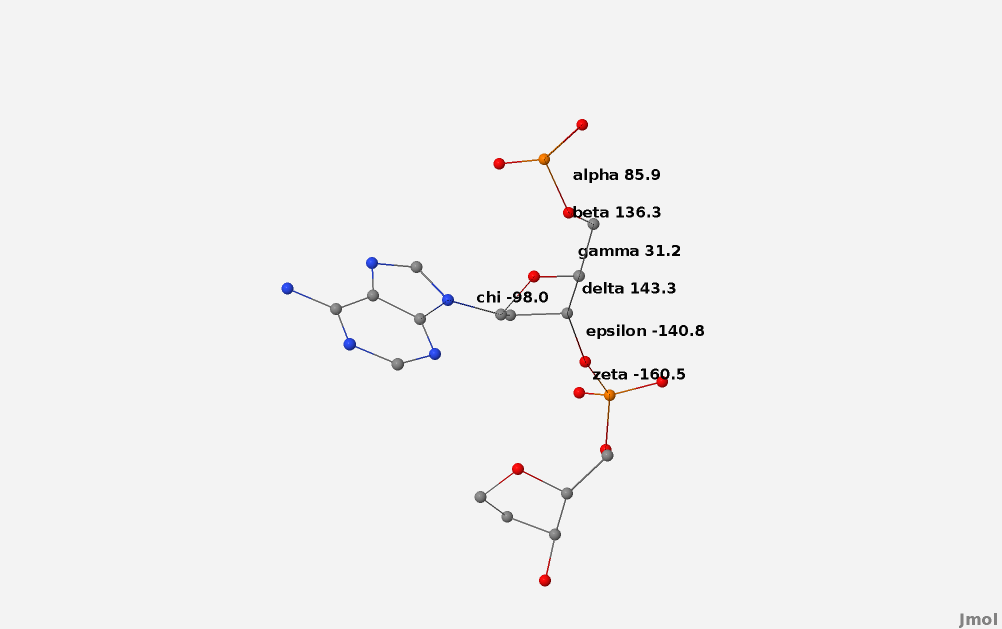
Рис. 13 Структурная формула азотистого основания аденина в B-форме ДНК, с пояснениями по поводу расположения и значения торсионных углов.
| угол | α | β | γ | δ | ε | ξ | χ |
| A-форма (презентация) | 62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| A-форма (Jmol) | 64.1 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157,2 |
| B-форма (презентация) | 63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
| B-форма (Jmol) | 85.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
Задание №4. Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA.
Определение торсионных углов нуклеотидов.
Вначале определим торсионные углы для ДНК из A-,B- и Z- формы. Для этоко был написан следующий скрипт, использующий программы пакета 3DNA и стандартные программы Linux, для получения информации о торсионных углах в pdb структурах A-, B- и Z- форм из Задания №1.
Получение информации от орсионных углах.
for file in gatc-?.pdb;do sed /^REMARK/d $file > clean_${file};remediator --old clean_${file} >clean_old_${file}; find_pair -t -s clean_old_${file} stdout |analyze;rm clean_*${file};done
В результате работы этого скрипта получены файлы, содержащие информацию о торсионных углах в pdb структурах A-,B- и Z- форм ДНК:  clean_old_gatc-a.outs,
clean_old_gatc-a.outs,  clean_old_gatc-b.outs,
clean_old_gatc-b.outs,  clean_old_gatc-z.outs
clean_old_gatc-z.outs
Далее они были обработаны в программе LibreOffice Calc в результате чего был получен  файлс информацией о средних значениях всех торсионных углов для каждого нуклеотида в каждой форме ДНК.
файлс информацией о средних значениях всех торсионных углов для каждого нуклеотида в каждой форме ДНК.
Итог анализа данных для торсионных углов A-,B- и Z-форм ДНК представлен в Таблице 3.
| form | base | α | β | γ | δ | ε | ξ | χ |
| A | G | -51.70 | 174.80 | 41.70 | 79.09 | -147.79 | -75.10 | -157.20 |
| B | G | -29.90 | 136.34 | 31.14 | 143.34 | -140.80 | -160.50 | -98.00 |
| Z | G | 51.93 | 179.00 | -173.80 | 94.90 | -103.60 | -64.80 | 58.70 |
| delta AB | G | 21.80 | 38.46 | 10.56 | 64.25 | 6.99 | 85.40 | 59.20 |
| delta AZ | G | 103.63 | 4.20 | 215.50 | 15.81 | 44.19 | 10.30 | 215.90 |
| delta BZ | G | 81.83 | 42.66 | 204.94 | 48.44 | 37.20 | 95.70 | 156.70 |
| A | A | -51.70 | 174.80 | 41.70 | 79.08 | -147.80 | -75.10 | -157.20 |
| B | A | -29.90 | 136.34 | 31.14 | 143.34 | -140.80 | -160.50 | -98.00 |
| Z | A | none | none | none | none | none | none | none |
| delta AB | A | 21.80 | 38.46 | 10.56 | 64.26 | 7.00 | 85.40 | 59.20 |
| delta AZ | A | none | none | none | none | none | none | none |
| delta BZ | A | none | none | none | none | none | none | none |
| A | T | -51.70 | 174.80 | 41.70 | 79.10 | -147.80 | -75.10 | -157.20 |
| B | T | -29.90 | 136.34 | 31.14 | 143.34 | -140.80 | -160.50 | -97.98 |
| Z | T | none | none | none | none | none | none | none |
| delta AB | T | 21.80 | 38.46 | 10.56 | 64.24 | 7.00 | 85.40 | 59.22 |
| delta AZ | T | none | none | none | none | none | none | none |
| delta BZ | T | none | none | none | none | none | none | none |
| A | C | -51.70 | 174.80 | 41.70 | 79.09 | -147.79 | -75.09 | -157.20 |
| B | C | -29.90 | 136.34 | 31.14 | 143.34 | -140.80 | -160.50 | -97.98 |
| Z | C | -139.50 | -136.77 | 50.87 | 137.60 | -96.50 | 81.97 | -154.30 |
| delta AB | C | 21.80 | 38.46 | 10.56 | 64.25 | 6.99 | 85.41 | 59.23 |
| delta AZ | C | 87.80 | 311.57 | 9.17 | 58.51 | 51.29 | 157.05 | 2.90 |
| delta BZ | C | 109.60 | 273.10 | -19.73 | 5.74 | -44.30 | -242.47 | 56.33 |
Таблица 3. Сравнение торсионных углов в различных нуклеотидах в A-,B- и Z-формах. Запись delta обозначает приращение градусной меры торсионного угла в одной форме относительно другой.
Как видно из Таблицы 3 между формами A и B имеется довольно сильное различие только в торсионных углах δ, ξ и χ. Однако Z-форма ДНК отличается от форм A и B почти по всем торсионным углам для нуклеотидов с гуанином и цитозином.
Определение значения торсионных углов в заданной структуре тРНК.
Для получения информации о торсионных углах тРНК использовался файл pdb из Задания 2, обработанный следующим скриптом,  выходной файл которого был обработан в LibreOffice Calc. Конечный
выходной файл которого был обработан в LibreOffice Calc. Конечный  результат обработки представлен в Таблице 4.
результат обработки представлен в Таблице 4.
Получение информации о торсионных углах.
file=1EHZ_only_RNA.pdb; sed /^REMARK/d $file > clean_${file};remediator --old clean_${file} >clean_old_${file}; find_pair -t -s clean_old_${file} stdout |analyze;rm clean_*${file}
| base | α | β | γ | δ | ε | ξ | χ |
| G | -26.37 | 74.65 | 59.60 | 91.90 | -138.08 | -83.55 | -122.90 |
| C | -33.14 | 94.52 | 64.07 | 90.11 | -144.73 | -50.83 | -156.96 |
| A | -54.06 | 73.18 | 28.98 | 90.04 | -140.73 | -78.13 | -152.26 |
| U | -37.25 | 3.15 | 62.27 | 90.72 | -114.25 | -66.45 | -149.89 |
Таблица 4. Сравнение торсионных углов в различных нуклеотидах в тРНК из pdb 1EHZ, полученное взятием среднего арифметического для каждого нуклеотида.
Как видно из Таблицы 4, данная тРНК по торсионным углам в своих нуклеотидах похожа на А-форму ДНК.
Определение значения торсионных углов в заданной структуре ДНК.
Для получения информации о торсионных углах ДНК использовался файл pdb из Задания 2, обработанный следующим скриптом,  выходной файл которого был обработан в LibreOffice Calc. Конечный
выходной файл которого был обработан в LibreOffice Calc. Конечный  результат обработки представлен в Таблице 5.
результат обработки представлен в Таблице 5.
Получение информации о торсионных углах.
file=1D5Y_only_DNA.pdb; sed /^REMARK/d $file > clean_${file};remediator --old clean_${file} >clean_old_${file}; find_pair -t -s clean_old_${file} stdout |analyze;rm clean_*${file}
| № | base | α | β | γ | δ | ε | ξ | χ | сумма модулей разности торсионных углов и их среднего |
| 1 | T, не учит. | --- | --- | 51.80 | 142.60 | 162.20 | -86.60 | -108.60 | #VALUE! |
| 2 | G | -62.50 | -155.90 | 43.00 | 143.20 | -172.80 | -128.60 | -106.10 | 210.18 |
| 3 | A | -51.00 | 170.90 | 47.10 | 145.10 | 179.50 | -91.70 | -107.20 | 302.33 |
| 4 | C | -56.70 | 177.30 | 47.10 | 136.10 | -147.80 | -150.90 | -110.30 | 244.69 |
| 5 | A | -58.90 | 156.50 | 50.20 | 137.70 | -166.00 | -100.10 | -124.30 | 316.69 |
| 6 | G | -56.20 | 171.60 | 39.20 | 135.70 | -167.60 | -152.10 | -109.70 | 341.41 |
| 7 | C | -27.80 | 143.90 | 46.90 | 141.70 | -161.10 | -116.90 | -115.50 | 240.98 |
| 8 | A | -70.30 | 176.40 | 49.90 | 143.20 | -163.60 | -117.70 | -105.20 | 352.93 |
| 9 | C | -65.90 | 177.30 | 45.10 | 133.70 | 158.00 | -91.90 | -98.80 | 401.97 |
| 10 | T | -50.50 | -159.00 | 41.70 | 139.10 | -178.90 | -94.00 | -103.60 | 318.46 |
| 11 | G | -58.00 | -178.90 | 41.00 | 140.00 | -147.90 | -155.60 | -103.80 | 228.02 |
| 12 | A | -38.80 | 142.40 | 38.40 | 132.30 | 176.50 | -96.00 | -115.40 | 287.26 |
| 13 | A | -49.30 | -163.80 | 33.00 | 139.30 | 171.40 | -96.20 | -100.30 | 405.70 |
| 14 | T | -49.60 | -176.40 | 45.00 | 135.80 | 176.10 | -109.70 | -106.30 | 315.12 |
| 15 | G | -53.40 | -178.30 | 44.00 | 141.40 | 170.90 | -100.40 | -111.40 | 429.08 |
| 16 | T | -62.20 | -157.50 | 41.00 | 140.70 | -160.90 | -133.40 | -101.00 | 313.02 |
| 17 | C | -42.80 | 149.50 | 44.80 | 134.60 | 178.50 | -96.70 | -110.80 | 380.11 |
| 18 | A | -62.90 | -172.00 | 46.80 | 143.30 | -143.30 | -176.20 | -96.00 | 490.04 |
| 19 | A | -48.30 | 131.50 | 53.10 | 143.70 | 175.50 | -94.10 | -117.50 | 262.90 |
| 20 | A | -55.10 | -173.00 | 42.60 | 141.20 | 178.20 | -104.10 | -115.10 | 395.25 |
| 21 | G, не учит. | -72.70 | -155.20 | 42.20 | 147.10 | --- | --- | -93.30 | #VALUE! |
| 22 | A, не учит. | --- | --- | 68.80 | 142.80 | 165.40 | -76.10 | -135.10 | #VALUE! |
| 23 | C | -113.60 | -128.70 | 53.40 | 142.70 | -172.10 | -162.20 | -91.30 | 424.97 |
| 24 | T | -23.10 | 130.40 | 52.00 | 140.90 | 165.60 | -101.30 | -115.50 | 402.55 |
| 25 | T | -68.90 | -154.60 | 52.10 | 143.00 | 171.10 | -87.20 | -107.90 | 322.79 |
| 26 | T | -69.30 | -159.10 | 44.90 | 141.90 | -161.30 | -137.50 | -93.30 | 331.22 |
| 27 | G | -44.80 | 147.70 | 45.40 | 139.80 | -155.50 | -151.50 | -109.60 | 312.02 |
| 28 | A | -53.70 | 157.10 | 47.90 | 142.60 | 177.30 | -83.30 | -121.80 | 296.47 |
| 29 | C | -63.50 | -168.20 | 39.00 | 142.50 | -177.00 | -142.40 | -105.40 | 378.59 |
| 30 | A | -46.10 | 166.30 | 51.50 | 143.80 | 175.60 | -90.00 | -114.20 | 299.30 |
| 31 | T | -65.10 | -163.40 | 39.90 | 134.30 | 161.50 | -91.90 | -98.60 | 317.69 |
| 32 | T | -60.20 | -152.40 | 42.30 | 138.10 | -164.50 | -113.60 | -102.20 | 285.55 |
| 33 | C | -45.30 | 154.30 | 45.60 | 137.50 | -155.70 | -157.70 | -102.80 | 232.18 |
| 34 | A | -46.20 | 144.10 | 53.60 | 144.10 | -172.00 | -90.20 | -123.00 | 321.64 |
| 35 | G | -74.70 | -165.90 | 39.60 | 146.00 | -150.70 | -139.70 | -88.70 | 239.62 |
| 36 | T | -52.80 | 147.10 | 48.40 | 135.90 | -165.90 | -134.80 | -90.30 | 412.19 |
| 37 | G | -39.40 | 157.40 | 39.50 | 135.00 | -161.20 | -153.70 | -117.70 | 347.61 |
| 38 | C | -9.10 | 132.20 | 30.10 | 136.10 | 175.80 | -124.40 | -106.60 | 371.33 |
| 39 | T | -34.90 | 169.10 | 44.90 | 140.60 | 173.20 | -83.10 | -118.90 | 454.08 |
| 40 | G | -78.10 | -157.10 | 41.40 | 142.40 | -172.30 | -143.00 | -99.60 | 242.82 |
| 41 | T | -51.60 | 158.60 | 59.20 | 137.00 | 176.10 | -84.80 | -125.50 | 445.68 |
| 42 | C, не учит. | -82.50 | -161.20 | 48.90 | 145.30 | --- | --- | -102.50 | #VALUE! |
| 43 | T, не учит. | --- | --- | 56.60 | 139.70 | 172.80 | -88.90 | -112.80 | #VALUE! |
| 44 | G | -50.80 | -174.10 | 38.80 | 141.20 | -158.70 | -147.90 | -105.80 | 237.41 |
| 45 | A | -42.80 | 155.00 | 40.60 | 137.20 | 177.80 | -84.30 | -115.30 | 301.66 |
| 46 | C | -53.00 | -177.40 | 42.80 | 131.90 | -168.20 | -129.70 | -115.60 | 362.43 |
| 47 | A | -47.00 | 173.00 | 42.70 | 138.00 | -167.80 | -108.40 | -114.10 | 326.09 |
| 48 | G | -46.60 | 164.90 | 39.00 | 136.30 | -170.30 | -139.80 | -107.60 | 332.21 |
| 49 | C | -40.10 | 161.90 | 44.00 | 137.40 | -176.00 | -107.30 | -116.90 | 266.04 |
| 50 | A | -64.60 | -167.30 | 45.70 | 147.00 | -169.90 | -125.60 | -98.80 | 463.93 |
| 51 | C | -49.80 | 162.40 | 48.90 | 135.70 | 175.30 | -104.70 | -112.00 | 379.01 |
| 52 | T | -60.70 | -167.30 | 48.90 | 140.50 | 178.50 | -95.10 | -94.50 | 328.93 |
| 53 | G | -55.70 | -179.80 | 43.00 | 139.90 | -150.10 | -149.20 | -102.20 | 228.13 |
| 54 | A | -45.40 | 149.50 | 40.80 | 134.60 | 160.60 | -86.40 | -110.40 | 276.60 |
| 55 | A | -69.50 | -149.10 | 44.40 | 141.60 | 175.20 | -108.30 | -102.50 | 393.59 |
| 56 | T | -48.90 | -179.20 | 45.10 | 139.90 | -171.90 | -116.00 | -106.00 | 321.99 |
| 57 | G | -54.80 | 170.00 | 48.10 | 138.20 | 164.30 | -93.40 | -111.90 | 516.98 |
| 58 | T | -72.20 | -146.10 | 41.30 | 142.30 | -161.60 | -172.80 | -93.50 | 360.52 |
| 59 | C | 9.70 | 102.60 | 30.60 | 131.20 | 174.60 | -95.90 | -96.70 | 400.56 |
| 60 | A | -56.70 | -173.90 | 43.30 | 140.90 | -163.70 | -174.20 | -101.10 | 499.20 |
| 61 | A | -36.90 | 132.10 | 54.90 | 143.60 | -179.80 | -83.40 | -132.00 | 343.34 |
| 62 | A | -72.80 | -164.80 | 43.50 | 135.70 | 166.40 | -99.30 | -111.20 | 398.28 |
| 63 | G, не учит. | -63.30 | -157.80 | 47.90 | 153.00 | --- | --- | -99.70 | #VALUE! |
| 64 | A, не учит. | --- | --- | 55.20 | 141.20 | 162.30 | -74.50 | -131.20 | #VALUE! |
| 65 | C | -96.70 | -134.30 | 48.20 | 142.90 | -160.10 | -144.70 | -98.10 | 372.37 |
| 66 | T | -47.70 | 143.90 | 55.40 | 136.20 | 167.40 | -94.90 | -118.10 | 405.68 |
| 67 | T | -62.50 | -153.70 | 43.50 | 139.50 | 160.70 | -87.50 | -104.20 | 296.46 |
| 68 | T | -70.10 | -150.20 | 48.90 | 147.40 | -165.20 | -142.50 | -91.10 | 340.90 |
| 69 | G | -46.90 | 146.80 | 49.20 | 139.70 | -158.20 | -146.50 | -112.50 | 313.52 |
| 70 | A | -50.40 | 158.50 | 46.50 | 145.80 | 165.20 | -73.40 | -112.60 | 289.23 |
| 71 | C | -78.00 | -151.10 | 43.30 | 142.80 | -175.20 | -141.50 | -97.70 | 377.77 |
| 72 | A | -48.10 | 162.30 | 51.00 | 140.30 | -175.70 | -90.10 | -117.50 | 331.21 |
| 73 | T | -67.30 | -169.40 | 37.90 | 134.90 | 169.80 | -110.90 | -96.80 | 322.16 |
| 74 | T | -46.90 | -175.00 | 45.80 | 134.10 | -165.30 | -108.30 | -114.30 | 317.64 |
| 75 | C | -45.50 | 162.10 | 40.70 | 139.80 | -158.70 | -157.10 | -103.40 | 242.94 |
| 76 | A | -36.10 | 137.90 | 49.90 | 140.70 | -167.70 | -84.40 | -126.50 | 324.01 |
| 77 | G | -95.60 | -164.20 | 47.00 | 149.20 | -68.80 | 162.40 | -75.50 | 502.59 |
| 78 | T | -92.80 | 147.90 | 27.80 | 133.60 | 162.50 | -99.40 | -85.90 | 446.14 |
| 79 | G | -40.30 | -166.10 | 37.50 | 138.40 | 179.70 | -110.10 | -102.20 | 427.88 |
| 80 | C | -39.20 | 169.90 | 36.10 | 137.10 | -158.80 | -149.50 | -111.10 | 255.58 |
| 81 | T | -31.20 | 132.90 | 50.90 | 134.40 | -170.00 | -95.40 | -128.60 | 424.17 |
| 82 | G | -75.30 | -168.80 | 41.00 | 140.50 | -177.40 | -131.60 | -101.70 | 241.82 |
| 83 | T | -53.50 | 163.60 | 62.00 | 138.30 | -163.00 | -104.20 | -116.60 | 411.97 |
| 84 | C, не учит. | -73.30 | 177.80 | 48.00 | 144.80 | --- | --- | -104.40 | #VALUE! |
| Среднее | G | -58.32 | -45.67 | 42.29 | 140.43 | -93.54 | -117.54 | -104.13 | |
| A | -52.31 | 52.25 | 46.25 | 140.99 | 18.62 | -102.61 | -112.82 | ||
| T | -56.45 | -48.63 | 46.31 | 138.56 | 1.55 | -109.01 | -105.12 | ||
| C | -51.08 | 58.36 | 42.91 | 137.73 | -59.28 | -129.59 | -105.81 |
Таблица 5. Анализ торсионных углов в ДНК из pdb 1D5Y. Получены торсионные углы для каждого нуклеотида, взято средние для каждого нуклеотида и торсионного угла, а также введена и посчитана величина суммы модулей разности между средним значением соответствующего торсионного угла и торсионного угла нуклеотида.
Как видно из Таблицы 5, самую большую сумму модулей разности тосиооных углов и их среднего имеет нуклеотид №57. Это говорит о том,что этот гуанин больше чем все другие нуклеотиды отличается по торсионным углам от среднего значения.
Определение структуры водородных связей.
Для выполнения этого задания был зпущен следующий скрипт и проанализирован выходной файл.
Для получения информации о структуре тРНК использовался файл pdb из Задания 2, обработанный следующим скриптом,  выходной файл которого был проанализирован.
выходной файл которого был проанализирован.
Получение информации структуре тРНК.
file=1EHZ_only_RNA.pdb; sed /^REMARK/d $file > new_clean_${file};remediator --old clean_${file} >new_clean_old_${file}; find_pair -t new_clean_old_${file} stdout |analyze;rm new_clean_*${file}
Полученные данные о стеблях в тРНК представлены на Рисунке 14.

Рис. 14 Полученная информация о стеблях тРНК при помощи програмного пакета 3DNA, красным цветом выделен акцепторный стебель, зеленым Т-стебель, синим D-стебель и желтым антикодоноый стебель.
На основании данных Рисунка 14, полученно изображение тРНК из pdb 1EHZ, раскрашенное с выделением стеблей. (см. Рисунок 15). Для раскраски был использован  скрипт раскрашивающий стебли в различные цвета.
скрипт раскрашивающий стебли в различные цвета.
stems.txt
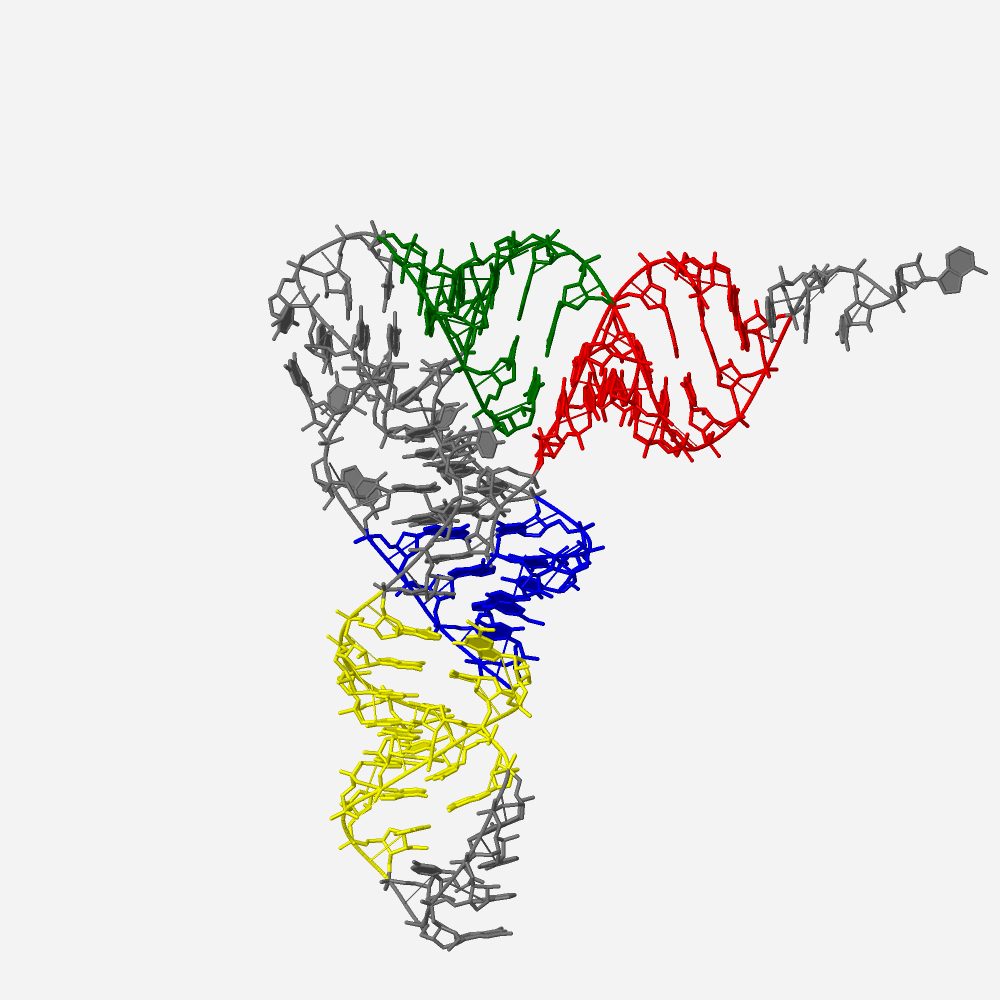
Рис. 15 Cтебли в тРНК pdb 1EHZ, информация о которых получена при помощи програмного пакета 3DNA: красным цветом выделен акцепторный стебель, зеленым Т-стебель, синим D-стебель и желтым антикодоноый стебель. Нуклеотиды не участвующие в образовании стеблей показаны серым.
Неканоничные (или модифицированные) взаимодействия, судя по Рисунку 14, происходят между следующими парами нуклеотидов:
4 (0.018) A:...4_:[..G]G-*---U[..U]:..69_:A (0.015) | 7 (0.008) A:...7_:[..U]Ux----A[..A]:..66_:A (0.016) | 13 (0.013) A:..54_:[5MU]u-**-xa[1MA]:..58_:A (0.012) | 15 (0.015) A:..36_:[..A]Ax*---U[..U]:..33_:A (0.014) | 16 (0.008) A:..38_:[..A]A-*---c[OMC]:..32_:A (0.008) | 17 (0.004) A:..39_:[PSU]P-*---A[..A]:..31_:A (0.007) | 22 (0.015) A:..44_:[..A]Ax*---g[M2G]:..26_:A (0.013) | 27 (0.025) A:..14_:[..A]A-**-xU[..U]:...8_:A (0.016) |
Водородные взаимодействия стабилизирующие структуру, судя по Рисунку 14, происходят между следующими парами нуклеотидов: 54----58 и 55----18, они стабилизируют Т-петлю
Поиск стэкинг-взаимодействия.
Рассматривая стекинг взаимодействия в  файле и информацией о вторичной структуре тРНК находим стекинг взаимодействия с наибольшим и наименьшим перекрытием. (см. Рисунок 16)
файле и информацией о вторичной структуре тРНК находим стекинг взаимодействия с наибольшим и наименьшим перекрытием. (см. Рисунок 16)
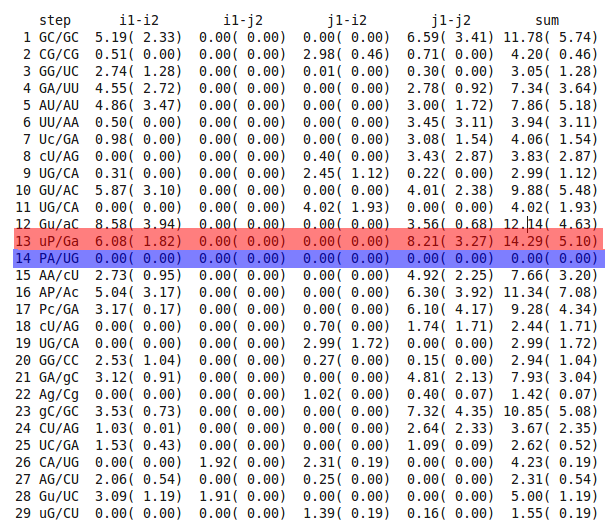
Рис. 16 Данные программы analyze для выбора анализируемых пар нуклеотидов с наибольшим и наименьшим перекрытием.
Для анализа стекинг взаимодействия отобраны структуры №13 и №14 с наибольшим и наименьшим перекрытием соответственно. Для получения графических изображений этих взаимодействий на Рисунке 17 и Рисунке 18 был написан следующий bash-скрипт берущий данные из  выходного файла analyze
выходного файла analyze
bash-скрипт для получения Рисунка 17 и Рисунка 18
ex_str -13 stacking.pdb step13.pdb
ex_str -14 stacking.pdb step14.pdb
stack2img -cdolt step13.pdb step13.ps
stack2img -cdolt step14.pdb step14.ps
convert step13.ps fig_17.png
convert step14.ps fig_18.png
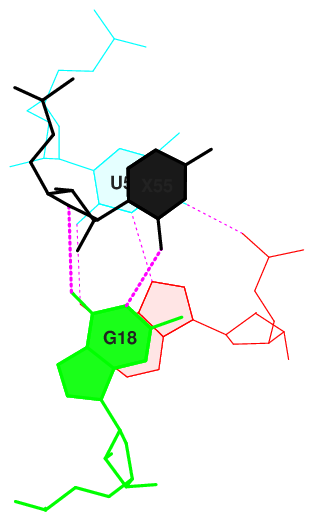
Рис. 17 Графическое изображение нуклеотидов с наибольшим перекрытием: пара U54 и U55 с парой G18 и U58.

Рис. 18 Графическое изображение нуклеотидов с наименьшим перекрытием: пара U33 и A36 с парой G18 и U58.
Так же для более наглядной иллюстрации стекинговых взаимодействий с наибольшим и наименьшим перекрыванием был написан  скрипт для Jmol'a, резульатом работы которого является Рисунок 19 и Рисунок 20, показывающие стекинговые взаимодействия в пространстве.
скрипт для Jmol'a, резульатом работы которого является Рисунок 19 и Рисунок 20, показывающие стекинговые взаимодействия в пространстве.
stacking_Jmol.txt
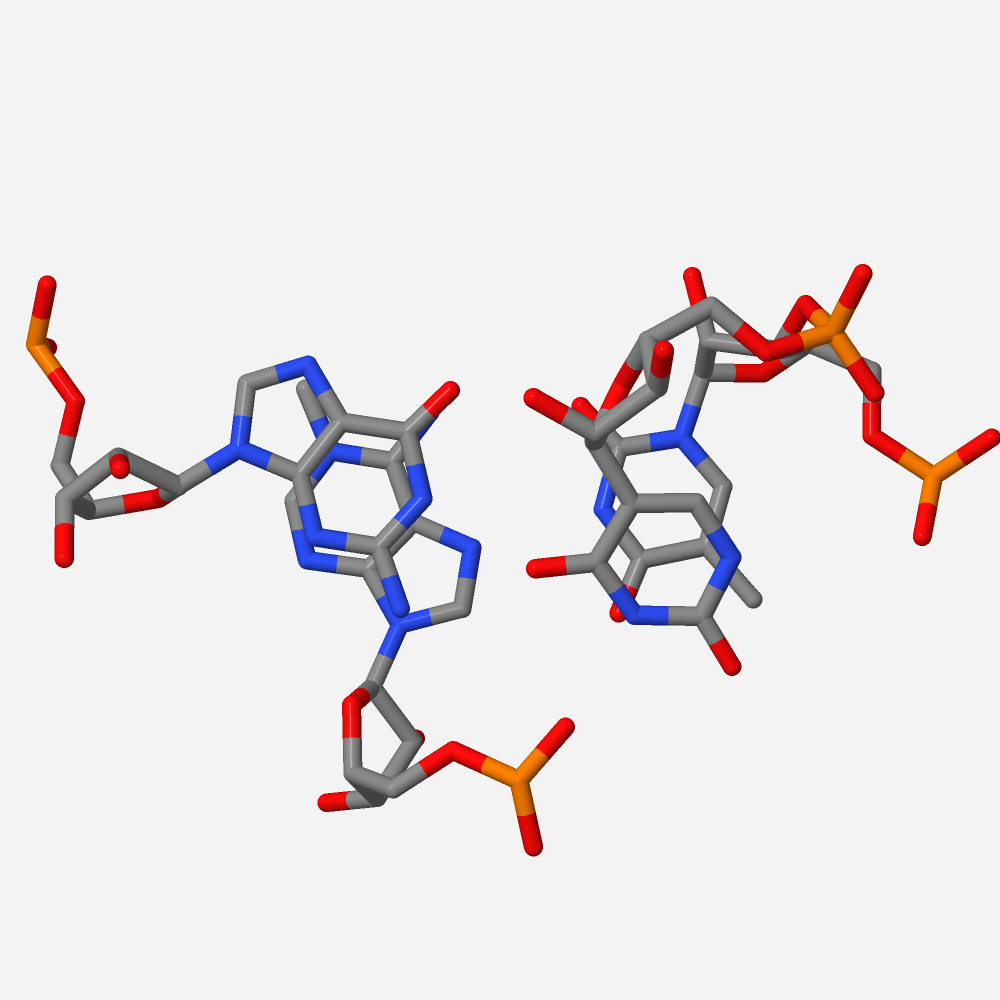
Рис. 19 Графическое изображение в программе Jmol нуклеотидов с наибольшим перекрытием: пара U54 и U55 с парой G18 и U58.
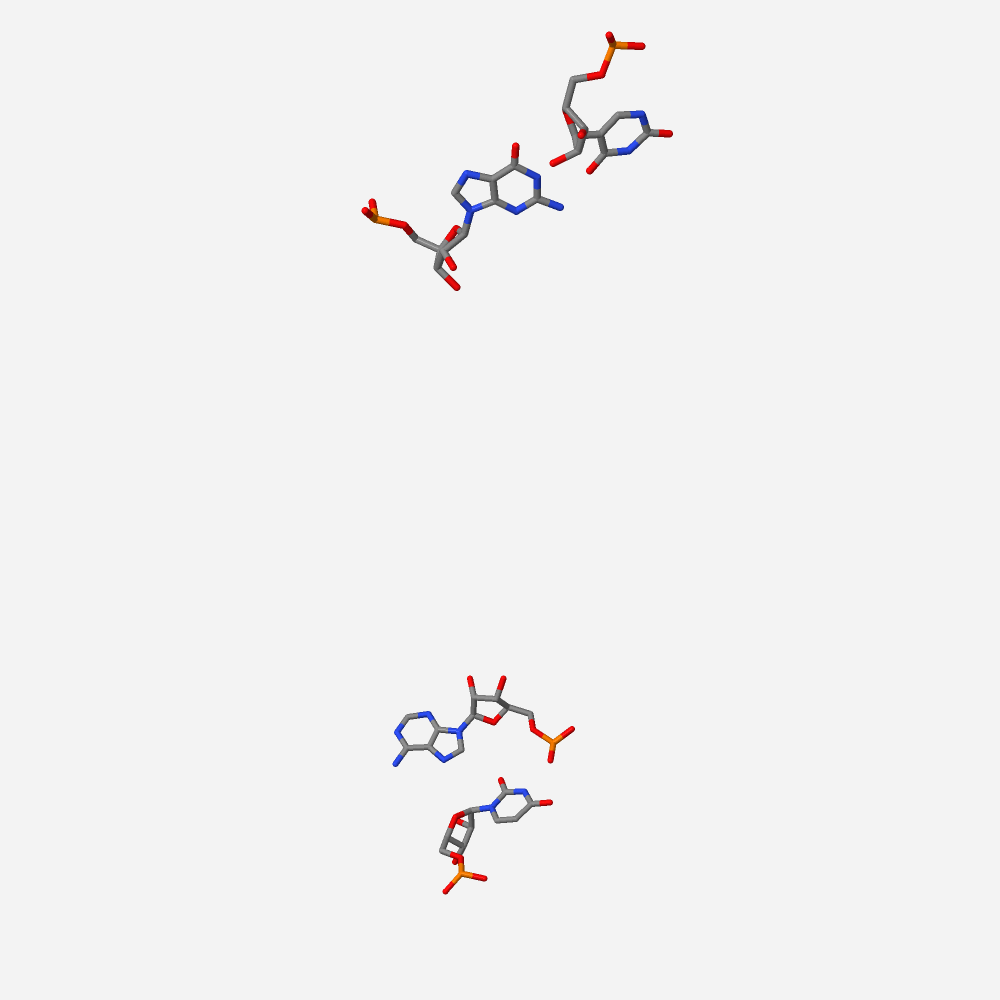
Рис. 20 Графическое изображение в программе Jmol нуклеотидов с наименьшим перекрытием: пара U33 и A36 с парой G18 и U58.
Графическое представление на Рисунке 17,18 совпадает с таким на Рисунке 19,20, на которых подробнее (в пространстве) показаны взаимодействия.
