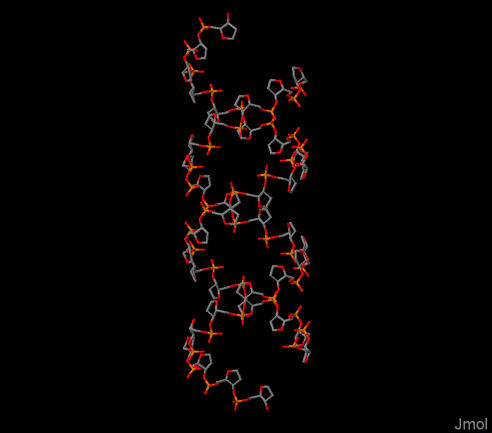
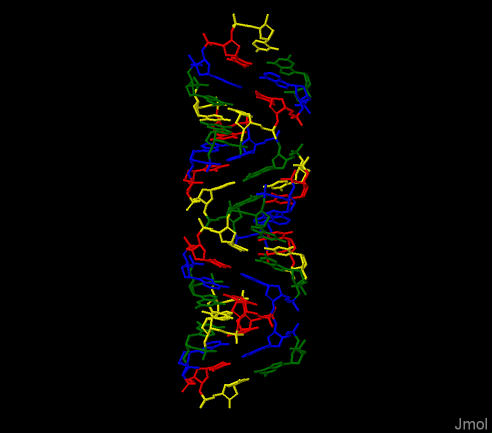
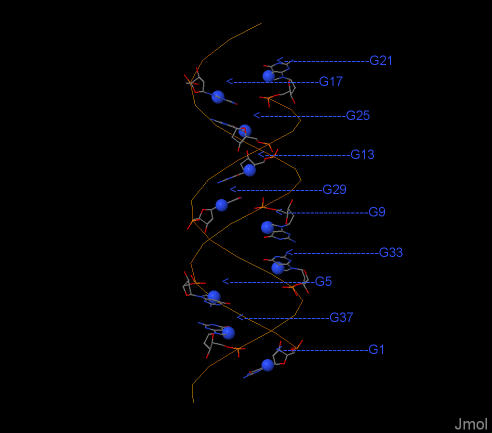
Остов A-формы ДНК.

Нуклеотиды - в модели wireframe, покрашены таким образом: А - синий,
С - жёлтый, G - зелёный, Т - красный.
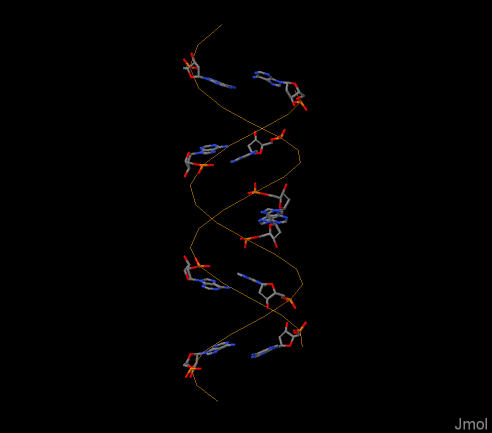
Аденины - в модели wireframe, остов схематически показан моделью backbone
оранжевым цветом.
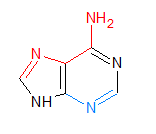
Гуанины - в модели wireframe, остов схематически показан моделью backbone
оранжевым цветом. Атомы N7 в гуанине показаны шариками (spacefill 250) и
подписаны.


| Форма ДНК | А-форма | В-форма | Z-форма |
| Основание | Аденин 38 | Аденин 38 | Гуанин 13 |
| Атомы, обращённые сторону большой бороздки | C5, C6, C8, N6, N7 | C5, C6, C8, N6, N7 | N1, C4, C5, C6, N7, C8, N9 |
| Атомы, обращённые в сторону малой бороздки | C2, N3, C4, N1, N9 | C2, N3, C4, N9 | C2, N3, N2 |
| Другие атомы | - | N1 | - |
| А-форма | В-форма | Z-форма | |
| Тип спирали | правая | правая | левая |
| Шаг спирали (Å) | 28,03 | 33,75 | 43,50 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (Å) | 16,81 | 17,21 | 11,58 |
| Ширина малой бороздки (Å) | 7,98 | 11,69 | 7,2 |
| Форма ДНК | α (P - O5') | β (O5' - C5') | γ (C5' - C4') | δ (C4' - C3') | ε (C3' - O3') | ξ (O3' - P) | χ (C1' - N) |
| А-форма | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| В-форма | -29.9 | 136.3 | 31.1 | 143.3 | -140.8 | -160.5 | -98.0 |
| Форма ДНК | α (P - O5') | β (O5' - C5') | γ (C5' - C4') | δ (C4' - C3') | ε (C3' - O3') | ξ (O3' - P) | χ (C1' - N) |
| А-форма | 62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| В-форма | 63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
| Форма ДНК | α (P - O5') | β (O5' - C5') | γ (C5' - C4') | δ (C4' - C3') | ε (C3' - O3') | ξ (O3' - P) | χ (C1' - N) |
| А-форма | -51,7 | 174,8 | 41,7 | 79,09 | -147,79 | -75,1 | -167,2 |
| В-форма | -29,9 | 136,34 | 31,14 | 143,34 | -140,8 | -160,5 | -97,99 |
| Z-форма | -139,5 или 51,93 |
179 или 136,76 |
-173,8 или 50,87 |
94,9 или 137,6 |
-103,6 или -96,5 |
-64,8 или 81,97 |
58,7 или -154,3 |
| Структура | α (P - O5') | β (O5' - C5') | γ (C5' - C4') | δ (C4' - C3') | ε (C3' - O3') | ξ (O3' - P) | χ (C1' - N) |
| тРНК | -52,062 | 77,118 | 56,626 | 86,721 | -147,611 | -75,144 | -151,943 |
| А-форма ДНК | -51,7 | 174,8 | 41,7 | 79,09 | -147,79 | -75,1 | -167,2 |
| В-форма ДНК | -29,9 | 136,34 | 31,14 | 143,34 | -140,8 | -160,5 | -97,99 |
| Структура | α (P - O5') | β (O5' - C5') | γ (C5' - C4') | δ (C4' - C3') | ε (C3' - O3') | ξ (O3' - P) | χ (C1' - N) |
| ДНК | -53,756 | 2,606 | 44,595 | 139,017 | -33,481 | -106,631 | -107,183 |
| А-форма ДНК | -51,7 | 174,8 | 41,7 | 79,09 | -147,79 | -75,1 | -167,2 |
| В-форма ДНК | -29,9 | 136,34 | 31,14 | 143,34 | -140,8 | -160,5 | -97,99 |
Strand I Strand II Helix
1 (0.008) A:...1_:[..G]G-----C[..C]:..72_:A (0.006) |
2 (0.006) A:...2_:[..C]C-----G[..G]:..71_:A (0.013) |
3 (0.021) A:...3_:[..G]G-----C[..C]:..70_:A (0.018) |
4 (0.018) A:...4_:[..G]G-*---U[..U]:..69_:A (0.015) |
5 (0.018) A:...5_:[..A]A-----U[..U]:..68_:A (0.011) |
6 (0.011) A:...6_:[..U]U-----A[..A]:..67_:A (0.020) |
7 (0.008) A:...7_:[..U]Ux----A[..A]:..66_:A (0.016) |
8 (0.020) A:..49_:[5MC]c-----G[..G]:..65_:A (0.017) |
9 (0.024) A:..50_:[..U]U-----A[..A]:..64_:A (0.014) |
10 (0.030) A:..51_:[..G]G-----C[..C]:..63_:A (0.016) |
11 (0.020) A:..52_:[..U]U-----A[..A]:..62_:A (0.016) |
12 (0.027) A:..53_:[..G]G----xC[..C]:..61_:A (0.012) |
13 (0.013) A:..54_:[5MU]u-**-xa[1MA]:..58_:A (0.012) |
14 (0.024) A:..55_:[PSU]Px**+xG[..G]:..18_:A (0.028) x
15 (0.015) A:..36_:[..A]Ax*---U[..U]:..33_:A (0.014) |
16 (0.008) A:..38_:[..A]A-*---c[OMC]:..32_:A (0.008) |
17 (0.004) A:..39_:[PSU]P-*---A[..A]:..31_:A (0.007) |
18 (0.005) A:..40_:[5MC]c-----G[..G]:..30_:A (0.013) |
19 (0.013) A:..41_:[..U]U-----A[..A]:..29_:A (0.019) |
20 (0.010) A:..42_:[..G]G-----C[..C]:..28_:A (0.007) |
21 (0.025) A:..43_:[..G]G-----C[..C]:..27_:A (0.011) |
22 (0.015) A:..44_:[..A]Ax*---g[M2G]:..26_:A (0.013) |
23 (0.018) A:..10_:[2MG]g-----C[..C]:..25_:A (0.011) |
24 (0.005) A:..11_:[..C]C-----G[..G]:..24_:A (0.013) |
25 (0.012) A:..12_:[..U]U-----A[..A]:..23_:A (0.030) |
26 (0.013) A:..13_:[..C]C----xG[..G]:..22_:A (0.017) |
27 (0.025) A:..14_:[..A]A-**-xU[..U]:...8_:A (0.016) |
28 (0.021) A:..15_:[..G]G-**+xC[..C]:..48_:A (0.015) |
29 (0.188) A:..16_:[H2U]ux**+xU[..U]:..59_:A (0.029) x
30 (0.026) A:..19_:[..G]G-----C[..C]:..56_:A (0.014) +
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 GC/GC 5.19( 2.33) 0.00( 0.00) 0.00( 0.00) 6.59( 3.41) 11.78( 5.74)
2 CG/CG 0.51( 0.00) 0.00( 0.00) 2.98( 0.46) 0.71( 0.00) 4.20( 0.46)
3 GG/UC 2.74( 1.28) 0.00( 0.00) 0.01( 0.00) 0.30( 0.00) 3.05( 1.28)
4 GA/UU 4.55( 2.72) 0.00( 0.00) 0.00( 0.00) 2.78( 0.92) 7.34( 3.64)
5 AU/AU 4.86( 3.47) 0.00( 0.00) 0.00( 0.00) 3.00( 1.72) 7.86( 5.18)
6 UU/AA 0.50( 0.00) 0.00( 0.00) 0.00( 0.00) 3.45( 3.11) 3.94( 3.11)
7 Uc/GA 0.98( 0.00) 0.00( 0.00) 0.00( 0.00) 3.08( 1.54) 4.06( 1.54)
8 cU/AG 0.00( 0.00) 0.00( 0.00) 0.40( 0.00) 3.43( 2.87) 3.83( 2.87)
9 UG/CA 0.31( 0.00) 0.00( 0.00) 2.45( 1.12) 0.22( 0.00) 2.99( 1.12)
10 GU/AC 5.87( 3.10) 0.00( 0.00) 0.00( 0.00) 4.01( 2.38) 9.88( 5.48)
11 UG/CA 0.00( 0.00) 0.00( 0.00) 4.02( 1.93) 0.00( 0.00) 4.02( 1.93)
12 Gu/aC 8.58( 3.94) 0.00( 0.00) 0.00( 0.00) 3.56( 0.68) 12.14( 4.63)
13 uP/Ga 6.08( 1.82) 0.00( 0.00) 0.00( 0.00) 8.21( 3.27) 14.29( 5.10)
14 PA/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
15 AA/cU 2.73( 0.95) 0.00( 0.00) 0.00( 0.00) 4.92( 2.25) 7.66( 3.20)
16 AP/Ac 5.04( 3.17) 0.00( 0.00) 0.00( 0.00) 6.30( 3.92) 11.34( 7.08)
17 Pc/GA 3.17( 0.17) 0.00( 0.00) 0.00( 0.00) 6.10( 4.17) 9.28( 4.34)
18 cU/AG 0.00( 0.00) 0.00( 0.00) 0.70( 0.00) 1.74( 1.71) 2.44( 1.71)
19 UG/CA 0.00( 0.00) 0.00( 0.00) 2.99( 1.72) 0.00( 0.00) 2.99( 1.72)
20 GG/CC 2.53( 1.04) 0.00( 0.00) 0.27( 0.00) 0.15( 0.00) 2.94( 1.04)
21 GA/gC 3.12( 0.91) 0.00( 0.00) 0.00( 0.00) 4.81( 2.13) 7.93( 3.04)
22 Ag/Cg 0.00( 0.00) 0.00( 0.00) 1.02( 0.00) 0.40( 0.07) 1.42( 0.07)
23 gC/GC 3.53( 0.73) 0.00( 0.00) 0.00( 0.00) 7.32( 4.35) 10.85( 5.08)
24 CU/AG 1.03( 0.01) 0.00( 0.00) 0.00( 0.00) 2.64( 2.33) 3.67( 2.35)
25 UC/GA 1.53( 0.43) 0.00( 0.00) 0.00( 0.00) 1.09( 0.09) 2.62( 0.52)
26 CA/UG 0.00( 0.00) 1.92( 0.00) 2.31( 0.19) 0.00( 0.00) 4.23( 0.19)
27 AG/CU 2.06( 0.54) 0.00( 0.00) 0.25( 0.00) 0.00( 0.00) 2.31( 0.54)
28 Gu/UC 3.09( 1.19) 1.91( 0.00) 0.00( 0.00) 0.00( 0.00) 5.00( 1.19)
29 uG/CU 0.00( 0.00) 0.00( 0.00) 1.39( 0.19) 0.16( 0.00) 1.55( 0.19)